2. 广西壮族自治区畜牧研究所, 南宁 530001
2. Guangxi Institute of Animal Science, Nanning 530001, China
水牛作为发展中国家重要的乳、肉、役兼用畜种,被国际粮农组织(FAO)认为是最具开发潜力和开发价值的家畜[1]。水牛奶因其脂肪、乳糖、蛋白质和灰分的含量比普通牛奶高而备受到人们的青睐[2],并享有“奶中之王”的美称[3]。水牛奶的钙、磷、铁和维生素含量均高于荷斯坦奶牛,其他微量元素也比较均衡。水牛奶的脂肪球和酪蛋白胶粒都比较大,加工干酪优势明显[4]。但因产奶量和繁殖率低,制约了其奶业发展的步伐[5]。
microRNAs(miRNAs)是在动物和植物中发现的一类通过转录后抑制基因表达的内源性非编码RNA[6-8]。越来越多的证据表明,miRNA具有控制基因表达[9],细胞分化、增殖和凋亡[10]等功能, 可能对基因功能研究和疾病防治探索有重要意义。近些年,人们在各种动物乳汁中发现了miRNA,由此扩展了人们对于miRNA调控功能范围的理解, 表明miRNA有可能通过哺乳从母体传递给新生儿,进而调控新生儿发育过程中的某些关键生理生化途径, 有助于新生个体的正常发育[11-12]。众所周知,乳腺在发育和泌乳过程中受到激素、生长因子和一些蛋白质严格且复杂的调控。随着生物信息学的发展,miRNA以其独特的调控模式和表达特性迅速成为生物学领域的研究热点[13]。但纵观这几年miRNA的研究,其主要集中于乳腺癌[14-15],而关于正常生理状态的乳腺发育和泌乳研究较少[16]。miRNA由于其自身序列短、同源性高,利用传统的克隆法、基因芯片技术等检测miRNA非常困难。随着测序技术的发展,高通量测序技术相继诞生并逐渐成熟[17]。该技术具有高通量、成本低、灵敏度高等特点,可以获得低丰度的表达基因,不局限于已知的基因组序列信息,适用于未知基因组序列的物种,不需要克隆,操作简单,应用领域广[18]。目前,高通量测序技术已成功运用于人[19]、奶牛、奶山羊等生物miRNA的研究,发现奶牛中有885条premiRNAs编码921条成熟体miRNAs,这些成熟体中有884条是唯一序列,并且成熟体中有544条在2个时期都表达。这些唯一序列中有283条已知miRNAs,96条是与其他物种同源的miRNAs,还有505条新发现的miRNAs[20]。利用Solex测序技术检测出山羊泌乳中期乳腺上皮细胞中miRNA的读数约占小RNAs的47.7%,共鉴定出1 143个miRNAs,山羊和牛特有的miRNAs为116个,并发现17个差异表达miRNAs的靶基因主要参与乳腺发育、细胞增殖和乳脂合成等重要代谢过程[21]。但是水牛乳腺组织miRNA测序与表达谱的相关研究较少。
本试验通过对本地水牛泌乳期和非泌乳期乳腺组织miRNA进行Solexa测序,探究乳腺特异性表达miRNA,并对其进行生物信息学分析,为研究水牛乳腺发育和泌乳提供理论参考。
1 材料与方法 1.1 试验材料从广西大学牛场随机选取2头健康经产水牛,其中1头8岁,泌乳3个月作为泌乳期样品来源(Lactation),另1头10岁,非泌乳未妊娠时期作为非泌乳期样本来源(Non-lactation)。分别从2头水牛乳腺随机采集乳腺组织样品, 迅速放入无Rase的冷存管中,放入液氮带回实验室转入-80 ℃超低温冰箱中保存,用于Solexa高通量测序。
1.2 主要试剂、仪器和软件 1.2.1 主要试剂按照J.Sambrook等[22]的方法配制常规试剂,以重蒸水为溶剂,15bf/in2 (1.034×105 Pa)为高压灭菌条件,蒸汽灭菌20 min。
内切酶和T4连接酶(TaKaRa);TRIzol、脂质体LipofectamineTM2000(Invitrogen公司);AMV逆转录酶、pMD-18T和荧光定量用SYBR®Premix Ex TaqTM(TaKaRa公司);RIPA裂解液;无特别说明的其他生化试剂均为国产分析纯。
1.2.2 使用仪器Millipore超纯水器、生化培养箱、离心机、定量PCR仪(ABI 7500)、Eppendorf移液器、EC250-90水平电泳仪、Gel Doc 2000凝胶成像系统、PCR仪(AB 2720)、Boxun 5W-CJ-2F超净工作台、BIO-RAD凝胶成像系统。
1.2.3 主要使用软件使用到的相关分子生物学软件有DNAMAN、Vector NTI、OLIGO6.0、BioEdit等。
1.3 试验方法 1.3.1 组织RNA的提取将所采集的组织在液氮中磨碎,每50~100 mg组织加入1 mL TRIzol,用匀浆仪进行匀浆处理。样品体积不超过TRIzol体积的10%,将匀浆样品在室温(15~30 ℃)放置5 min,使核酸蛋白复合物完全分离,从上清液中提取总RNA,RNA的数量和浓度用NanoDrop分光光度计进行确认,只有RNA完整指数≥7时方可被用来进一步测序和miRNA的分离鉴定,只有完整指数≥8时RNA方可被用做mRNA测序和分析。
1.3.2 Solexa测序Solexa高通量测序属于新一代测序方法(二代),主要原理是合成和测序同时进行(Sequencing by synthesis)[23]。
Solexa测序流程:经过屏蔽适配器过滤被rRNA、tRNA、mRNA、snRNA、snoRNA污染的序列,评估序列质量和计算小RNA序列的长度分布,干净的序列用计算机分析处理,最后,干净序列在miRBase的version 17.0中进行比对。
Solexa序列用SOAP放在牛基因库中(Btau 4.0)[23],完美匹配的序列被保留作为下一步分析对象。为了进一步分析包含完美匹配Solexa序列的RNA二级结构,提取这些序列每边的基因组侧翼100个核苷酸,用RNAfold对二级结构进行预测,用默认设置的MIREAP计算机工具进行分析,MIREAP是特别设计用于鉴别小RNA文库中的miRNA的,能够完整分析生物种源、测序深度和miRNAs的结构特征,以此提高miRNA鉴定的敏感性和特异性。只有完全满足以下条件的茎环发夹结构才被认为是miRNA:(1) 成熟miRNA必须是在发夹前的一个臂上,缺乏大量内环或凸起;(2) 发夹的二级结构必须稳定,混合淡化技术自由能低于75.35 kJ·mol-1;(3) 发夹位于内含子的基因间区。最后,在miRBase database 17.0中用BLAST查询候选miRNA序列,容许2个错配。
1.3.3 miRNAs靶基因的预测在Ensembl genome browser中使用查询功能将每个确认的miRNA定位于基因组上(http://www.ensembl.org/Bos_taurus)。靶基因的预测用MIREAP完成。MIREAP的基本预测规则:(1) 在sRNA和靶基因间(G-U计算为0.5错配)不大于4个错配; (2) 在miRNA/target duplex不大于2个邻近错配; (3) 在miRNA/target duplex的2~12位置上没有邻近错配; (4) 在miRNA/target duplex的10~11位置上没有错配; (5) 在miRNA/target duplex的1~12位置上≤2.5个错配;(6) miRNA/target duplex的最小自由能应该≥完全互补miRNA的最小自由能的75%。执行Blastx查询,用靶基因序列和EMBL数据库预测可能靶基因的功能。
1.3.4 miRNAs的差异表达分析为了比较泌乳期和非泌乳期2个文库miRNA的表达差异, 首先,miRNAs在2类样品中的表达被标准化到每百万转录本中的表达量。如果一个miRNA的标准化表达(NE)值标准化为0,那么它的校正表达值为0.01;如果一个miRNA的NE值在2个文库中都小于1,用公式计算标准化表达的倍数和P-value。
标准化表达(Normalized expression)=(实际miRNA测序序列读数/总干净序列读数)×1 000 000;
差异倍数(Fold change)=log2(泌乳期标准化表达值/非泌乳期标准化表达值);
| $p\left( {x|y} \right) = \left( {\frac{L}{N}} \right)\frac{{\left( {x + y} \right)!}}{{x!y!1 + {{\left( {\frac{L}{N}} \right)}^{(x + y + 1)}}}}$ |
| $C(y \le {y_{{\rm{min}}}}|x) = \sum\limits_{y = 0}^{y \le {y_{{\rm{min}}}}} {p\left( {y|x} \right)} $ |
| $D(y \ge {y_{{\rm{max}}}}|x) = \sum\limits_{y \ge {y_{{\rm{max}}}}}^\infty {p\left( {y|x} \right)} $ |
N和x分别代表非泌乳期sRNA文库的一个给定miRNA的干净序列总读数和标准化表达水平,L和y分别代表泌乳期sRNA文库的一个给定miRNA的干净序列总读数和标准化表达水平。
1.3.5 miRNAs表达簇的分级聚类PermutMatrix软件的Pearson distance[24]用来执行miRNA表达簇的分析,可将每个miRNA在这个库中的相对克隆频率计算出来,作为序列读数的总量分流一个特定库的每个miRNA的序列数量。
2 结果 2.1 水牛泌乳期和非泌乳期miRNA表达谱分析为了鉴定水牛泌乳期和非泌乳期差异表达的miRNAs,成功构建了2个用于Solexa高通量测序的sRNA文库,泌乳期和非泌乳期乳腺组织文库分别测序获得了13 667 160和13 150 323条reads。将低质量的序列移除,少量标记和测序,最终获得非泌乳期和泌乳期18~31 nt的12 569 467和12 768 110条高质量reads(表 1)。
|
|
表 1 两个文库序列的表达谱 Table 1 Expression profile of sequenced reads in 2 libraries |
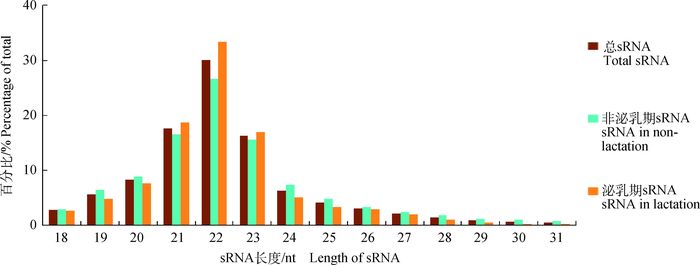
所有水牛基因组的测序读数长序列用SOAP程序与牛基因组(Btau_4.0) 进行比对[25],结果表明,非泌乳期文库12 569 467条高质量序列中有8 456 705条序列与牛基因组匹配,匹配率达67.28%。泌乳期文库12 768 110条序列中有7 665 749条与牛基因组匹配(表 2),匹配率为60.04%。统计显示,水牛泌乳期和非泌乳期的sRNA分布的宽度模式介于18~31 nt之间,在22 nt达到一个高峰值。在泌乳期乳腺组织中,22 nt序列占总sRNA数量的33.4% (图 1)。
|
|
表 2 已知miRNA检测分析 Table 2 Known micoRNA detection |
|
图 1 总sRNAs和2个文库序列长度的分布和频度百分数 Figure 1 Length distribution and frequency percent of sequences in total sRNAs and the 2 libraries |
为了鉴定这些sRNA是否是真正的miRNA,用MIREAP软件查阅牛基因组包括候选miRNA的发夹结构(水牛的基因组还未完全测序获得),利用Solexa深度测序从sRNA文库来区分已知和未知miRNAs。在miRbase17.0库中全部成熟microRNAs和前体microRNAs分别是676和662个,本次水牛乳腺组织测序确认的成熟microRNAs和前体microRNAs分别有359和363个;数据库中共有500个microRNA基因家族,本次水牛乳腺组织测序获得的miRNAs归属259个miRNAs家族(表 2)。
经过移除与蛋白质外显子编码区重叠且自由能小于-75.35 kJ·mol-1的miRNAs, 剩余的有新发夹结构的262位点被认为是miRNA的候选基因,其中230个miRNAs是水牛乳腺组织中新发现的miRNAs。在新发现的候选miRNAs中,35个新候选miRNAs中可以鉴定出反义miRNAs,为它们作为候选miRNA提供了新的证据(miRNA*)。5个miRNAs被认为是水牛特有的,因为在miRNAbase17.0中没有其同源序列。
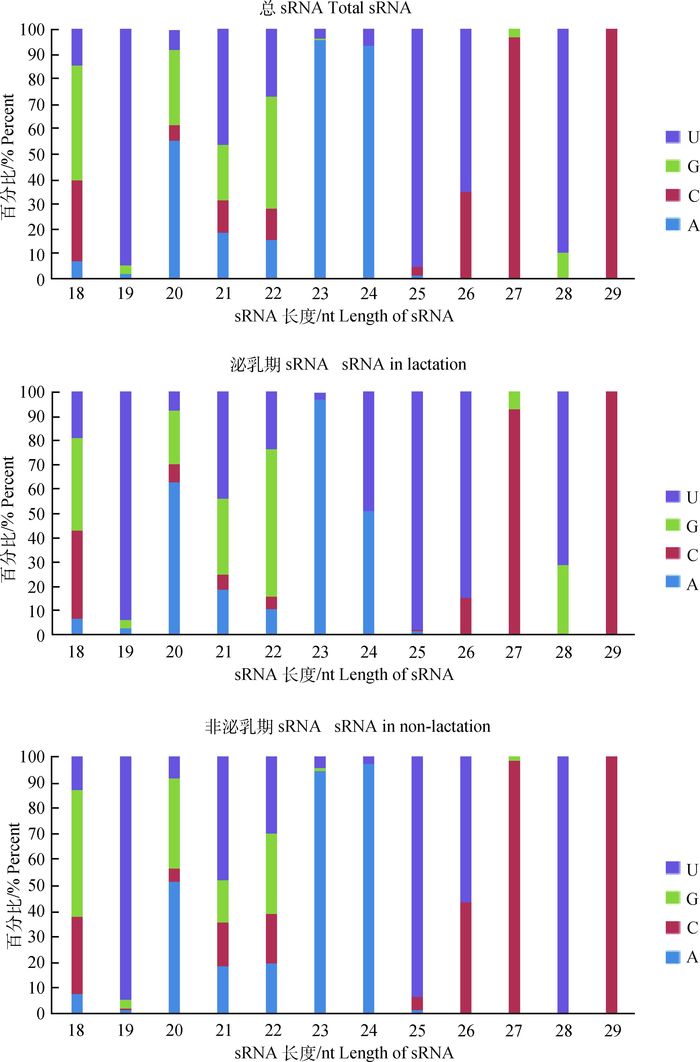
2.3 候选miRNAs的第一个核苷酸偏好性分析对所有鉴定miRNAs的第一个核苷酸偏好性分析显示(图 2),U是19和25 nt miRNAs 5′端最普遍的第一核苷酸(94.15%和97.90%)。而A是泌乳期乳腺组织20、23和24 nt新miRNAs 5′端最普遍的第一核苷酸,分别为96.71%、50.72%和62.51%。在非泌乳期乳腺组织中,U是19 (94.80%)、25(93.35%)和28 nt (100%)新miRNAs 5′端的最普遍第一核苷酸,而A是23 (94.59%)和24 nt (97.62%)新miRNAs 5′端的最普遍第一核苷酸。
|
图 2 候选18~29 nt miRNAs的第一核苷酸偏好性 Figure 2 The first nucleotide bias of the 18-29 nt candidate miRNAs |
本研究分析了已知miRNA的染色体位点(Btau 4.0为参考基因组序列),通过BLAST分析寻找水牛已知和新的前体miRNAs,结果见表 3。发现,68.77%的已知miRNA和84.69%的新miRNA位于常染色体上,成功定位于乳腺组织染色体上的基因间隔区。已知miRNA主要分布在21号和X染色体,分别为74和37个,占总数量(635) 的11.65%和5.83%,X染色体共有37个已知miRNAs(表 3)。
|
|
表 3 已知和新miRNA在染色体上的分布 Table 3 Distribution of known and new miRNAs in chromosomes |
|
|
表 4 染色体上miRNA的表达和密度分析 Table 4 Expression number and density of miRNAs in chromosome |
表 4显示,miRNA主要分布在常染色体和X染色体上,总miRNA密度为0.09~1.05个·Mb-1不等。21号染色体是miRNAs主要表达的染色体,分布最多,总miRNAs、泌乳期表达miRNAs和非泌乳期miRNAs分别为1.05、1.34和1.35个·Mb-1。泌乳期和非泌乳期miRNAs在相同染色体上的分布密度基本相同。
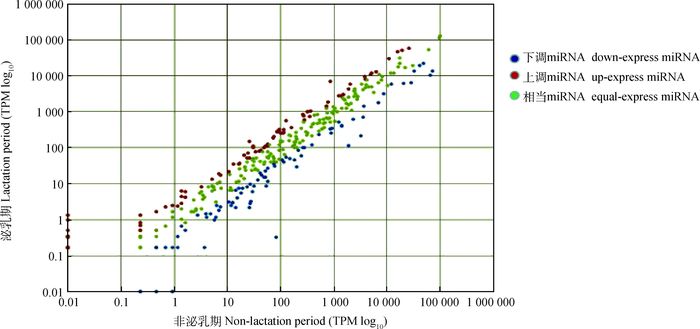
2.5 水牛泌乳期与非泌乳期miRNAs的差异表达Solexa测序方法通过测定序列频度提供了一个检测miRNAs表达谱的重要方法。2个文库中miRNAs的序列频度见图 3,基于miRNAs相对克隆频度的分层表明,水牛泌乳时miRNA表达是不同的。
|
X和Y轴分别表示泌乳期和非泌乳期乳腺组织中miRNA的表达量。红点代表ratio>2的miRNA,绿点代表 1/2<ratio<2的miRNA,蓝点代表ratio<1/2的miRNA。比值=处理的标准化表达/对照的标准化表达 The expression level of miRNAs in non-lactation (X-axis) and lactation (Y-axis) periods in mammary gland tissues. Red symbols represent miRNAs with ratio > 2; Green symbols represent miRNAs with 1/2 < ratio < 2; Blue symbols represent miRNAs with ratio < 1/2. Ratio=normalized expression of the treatment/normalized expression of the control 图 3 乳腺组织泌乳期和非泌乳期miRNA表达 Figure 3 The expression levels of miRNAs in mammary gland in non-lactation and lactation periods |
在非泌乳期,bbu-miR-148a、bbu-let-7b、bbu-let-7a、bbu-miR-21、bbu-miR-143、bbu-miR-200c、bbu-miR-26a、bbu-miR-200a和bbu-let-7f共9个miRNAs是主要表达的miRNAs, 大于20 000个序列,组成了总已知miRNA序列的53.8%,表明它们在非泌乳期组织中高丰度表达。57个miRNAs序列频度(比如bbu-miR-92b、bbu-miR-421和bbu-miR-129等)较低(1≤序列数≤10)。
在泌乳期,7个miRNAs (bbu-let-7b、bbu-let-7a、bbu-miR-26a、bbu-miR-125b、bbu-miR-21、bbu-miR-29a和bbu-let-7c)每个有20 000多条序列表达,丰度较高。可见bbu-let-7b、bbu-let-7a,miR-26a和miR-21这4个miRNAs在2个时期均呈现高表达。有124个miRNAs(比如bbu-miR-34c、bbu-miR-2284c和bbu-miR-383) 显示出较低的序列频度(<10 reads)。比较2个文库,bbu-miR-148a、bbu-miR-143、bbu-miR-200a、bbu-miR-141和bbu-miR-30a-5p等miRNAs在泌乳期的表达量降至非泌乳期的一半以下。另外一些miRNAs(比如bbu-miR-26a, bbu-miR-29a, bbu-miR-125b, bbu-let-7c和bbu-miR-99a)在泌乳期的表达量≥2倍的非泌乳期表达量。在2个时期的乳腺组织中,316个已知miRNAs在整个乳腺组织持续表达,在已知这些miRNAs中,分别发现19和24个非泌乳期和泌乳期特异表达的miRNAs。
2.6 靶基因和信号通路预测使用MIREAP软件对已知和新发现miRNA的潜在靶基因进行预测。发现了已知miRNA有246 786个假定的靶基因位点,乳腺组织中新发现的miRNA有209 655个假定靶基因位点。利用KEGG对20个差异高表达miRNA的预测靶基因进行功能分类(表 5)。已知miRNA的109个预测靶基因都被标记在MAPK信号通路,其他重要途径有Jak-STAT信号通路、促性腺激素释放激素信号通路(GnRH)和胰岛素信号通路等。
|
|
表 5 水牛乳腺中20个差异高表达miRNAs的靶基因在KEGG信号通路中的富集 Table 5 KEGG pathways enriched for target genes of the 20 differential highly expressed miRNAs in buffalo mammary gland |
在人类、小鼠、山羊和牛等物种中开展了很多关于miRNA的研究,但是水牛乳腺组织泌乳期和非泌乳期miRNA的系统和全面研究工作还未见报道。本研究应用Solexa测序技术对水牛泌乳期和非泌乳期乳腺组织进行了高通量测序,得到13 667 160和13 150 323个序列,而荷斯坦奶牛泌乳期和非泌乳期乳腺组织高通量测序共得到13 711 046和170 680 43个序列。长度分析结果显示,荷斯坦奶牛小RNA中52.6%为22 nt RNA[20],而水牛22 nt小RNA为33.4%,相差1.5倍左右,可能因为水牛的基因组尚未公布,有些序列难以比对。大部分新鉴定的水牛miRNA能够同源于物种上的已知miRNA,甚至有很近的关系,证明miRNA的是具有进化上的保守性[26]。研究共发现230个水牛乳腺组织新miRNA,有5个在miRNAbase 17.0中没有同源序列,为水牛特有。
不同阶段差异miRNA表达模式的分析能够为miRNA在乳腺的生长、发育和泌乳方面的作用提供有价值的信息。基于Solexa测序结果,不同miRNAs家族展示出显著的差异表达水平。例如,bta-let-7家族在水牛泌乳期乳腺中的丰度在深度测序中从12 reads (bta-let-7a*)到687 63 reads (bta-let-7b)不等,鉴定的bbu-miR-29家族有bbu-miR-29-a、bbu-miR-29-b、bbu-miR-29-c、bbu-miR-29-d和bbu-miR-29-e等5个成员,但miRNA家族中是由其主要成员发挥调控功能的。一些在泌乳期表达量降至非泌乳期表达量一半以下的miRNA,是在水牛开始泌乳后表达改变的,可能对泌乳发挥重要调控作用。bbu-miR-148a、bbu-miR-143、bbu-miR-200c、bbu-miR-200a和bbu-let-7f等为非泌乳期特异性高表达miRNAs,bbu-miR-125b、bbu-miR-29a和bbu-let-7c等为泌乳期特异性高表达miRNAs,对不同阶段也许起着重大调控作用。通过比较,非泌乳期bbu-miR-148a、bbu-miR-143、bbu-miR-200a等miRNAs差异高表达,泌乳期bbu-miR-26a、bbu-miR-29a、bbu-miR-125b、bbu-let-7c等miRNAs差异高表达,这些特异性miRNAs的实际表达量及其对水牛泌乳机制的调控作用值得进一步深入研究。
miRNA通过调控靶基因翻译来行使功能。目前,研究miRNA在正常生理条件下牛乳腺发育和泌乳相关的报道较少。比如miR-107被证明能够抑制乳腺组织乳腺细胞分裂和阻滞细胞周期,与细胞脂肪代谢有关[27]。D.P.Bartel[26]研究发现,当导管系统开始活跃生长时,miR-23在泌乳期和非泌乳期表现出相反的表达模式。预测miR-29、miR-15b和miR-107的靶基因XDH(Xanthine dehydrogenase)活性的增加是在乳腺的早期发育中[28]。miR-142、miR-23、miR-374b、miR-30a和miR-27b等通过调节泌乳关键基因PRLR,从而维持乳腺腺泡的存活、紧密连接和控制奶蛋白和乳糖的合成[29]。研究表明,miR-141的靶基因是STAT5,STAT5能作为乳腺中泌乳和催乳激素信号转导通路上的节点而对泌乳产生作用[30]。miR-141、miR-484和miR-500通过靶向STAT5和HK2基因对调控泌乳至关重要。本研究发现,泌乳期bbu-miR-141表达量下降至非泌乳期时的一半以下, 对水牛泌乳至关重要,是否也是通过STAT5和HK2基因调控泌乳来发挥作用的有待进一步研究。另外,通过对山羊泌乳期和非泌乳期乳腺组织进行高通量测序,得知miR-103泌乳期表达量为非泌乳期的4.3倍,并通过调控靶基因PANK3来调控奶山羊的乳汁分泌[31]。T.Tanaka等[32]采用微阵列分析乳腺不同发育阶段细胞中miRNA的表达差异,得出miR-101通过靶向cox-2影响乳腺发育。研究表明,敲除人正常乳腺上皮细胞的miR-29a可提高乳腺细胞的生长速度[33],miR-29a、miR-29b、miR-29c均能提高奶牛乳腺上皮细胞的活力,促进细胞增殖,并且对奶牛乳腺上皮细胞β-酪蛋白、甘油三酯及乳糖的分泌均有促进作用[34],与本研究测序发现水牛bbu-miR-29a在泌乳期特异性高表达结果一致。本研究中,109个miRNAs都被标记在MAPK信号通路,这个通路几乎在乳腺发育的每一步都发挥重要作用,其他重要途径有Jak-STAT信号通路,其与乳腺泌乳密切相关,尤其是在催乳素(PRL)信号转导中[35]。还有一条显著的高丰度miRNA靶向的通路是胰岛素信号通路, 已知其涉及乳糖代谢和奶生产性能[36]。
4 结论本试验通过对水牛泌乳期和非泌乳期乳腺组织进行高通量测序,研究其miRNA的表达谱。测序获得359个物种间保守的miRNAs、5个水牛特有的miRNAs,4个2个时期均高表达的miRNAs、9个非泌乳时期高丰度表达的miRNAs、7个泌乳时期高丰度表达的miRNAs和10个差异表达的miRNAs。miRNA靶基因预测和功能分析得知,miRNA参与乳腺发育与泌乳相关的细胞信号通路,为进一步研究miRNA在乳腺发育与泌乳中的调控作用奠定了理论基础。
| [1] |
林浪, 龚云, 王梦, 等. 水牛缝隙连接蛋白43基因克隆及表达[J]. 中国畜牧兽医, 2013, 40(9): 1–7.
LIN L, GONG Y, WANG M, et al. Cloning and expression of buffalo Cx43 gene[J]. China Animal Husbandry & Veterinary Medicine, 2013, 40(9): 1–7. (in Chinese) |
| [2] | AHMAD S, GAUCHER I, ROUSSEAU F, et al. Effects of acidification on physico-chemical characteristics of buffalo milk:A comparison with cow's milk[J]. Food Chem, 2008, 106(1): 11–17. DOI: 10.1016/j.foodchem.2007.04.021 |
| [3] |
唐善生. 广西奶水牛业发展现状与思考[J]. 广西农学报, 2014, 29(1): 50–52, 55.
TANG S S. Dairy buffalo industry developmental current situation and considerations in Guangxi[J]. Journal of Guangxi Agriculture, 2014, 29(1): 50–52, 55. (in Chinese) |
| [4] |
简保权, 秦学敏, 龚芳. 世界水牛奶业发展现状和典型模式分析[J]. 世界农业, 2015(3): 115–118.
JIAN B Q, QIN X M, GONG F. Buffalo dairy industry developmental current situation and the analysis of typical model in the world[J]. World Agriculture, 2015(3): 115–118. (in Chinese) |
| [5] |
王锦. 水牛乳腺基因表达谱与生长激素转基因水牛的初步研究[D]. 南宁: 广西大学, 2013.
WANG J. Preliminary studies on buffalo mammary gene expression profile and growth hormone transgenic buffalo[D]. Nanning:Guangxi University, 2013. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10593-1013359122.htm |
| [6] | BARTEL D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–297. DOI: 10.1016/S0092-8674(04)00045-5 |
| [7] | AMBROS V. The functions of animal MicroRNAs[J]. Nature, 2004, 431(7006): 350–355. DOI: 10.1038/nature02871 |
| [8] | KIM V N. MicroRNA biogenesis:coordinated cropping and dicing[J]. Nat Rev Mol Cell Biol, 2005, 6(5): 376–385. |
| [9] | WANG K, LONG B, JIAO J Q, et al. miR-484 regulates mitochondrial network through targeting Fis1[J]. Nat Commun, 2012, 3: 781. DOI: 10.1038/ncomms1770 |
| [10] | WIGHTMAN B, HA I, RUVKUN G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans[J]. Cell, 1993, 75(5): 855–862. DOI: 10.1016/0092-8674(93)90530-4 |
| [11] | WEBER J A, BAXTER D H, ZHANG S L, et al. The microRNA spectrum in 12 body fluids[J]. Clin Chem, 2010, 56(11): 1733–1741. DOI: 10.1373/clinchem.2010.147405 |
| [12] | KOSAKA N, IZUMI H, SEKINE K, et al. microRNA as a new immune-regulatory agent in breast milk[J]. Silence, 2010, 1(1): 7. DOI: 10.1186/1758-907X-1-7 |
| [13] |
张抒, 张强, 张萌, 等. miRNA对乳腺发育及泌乳调节作用的研究进展[J]. 中国畜牧兽医, 2015, 42(3): 663–667.
ZHANG S, ZHANG Q, ZHANG M, et al. Research progress on regulation effect of microRNA on mammary gland development and lactation[J]. China Animal Husbandry & Veterinary Medicine, 2015, 42(3): 663–667. (in Chinese) |
| [14] | DENIS H, VAN GREMBERGEN O, DELATTE B, et al. MicroRNAs regulate KDM5 histone demethylases in breast cancer cells[J]. Mol BioSyst, 2015, 12(2): 404–413. |
| [15] | PHAN B, MAJID S, URSU S, et al. Tumor suppressor role of microRNA-1296 in triple-negative breast cancer[J]. Oncotarget, 2016, 7(15): 19519–19530. DOI: 10.18632/oncotarget.v7i15 |
| [16] | COCHRANE D R, SPOELSTRA N S, RICHER J K. The role of miRNAs in progesterone action[J]. Mol Cell Endocrinol, 2012, 357(1-2): 50–59. DOI: 10.1016/j.mce.2011.09.022 |
| [17] |
熊显荣, 兰道亮, 李键, 等. 牦牛卵巢小RNA高通量测序及生物信息学分析[J]. 畜牧兽医学报, 2016, 47(1): 55–63.
XIONG X R, LAN D L, LI J, et al. Solexa sequencing of small RNAs in yak (Bos grunniens) ovaries and bioinformatics analysis[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(1): 55–63. (in Chinese) |
| [18] |
周华, 张新, 刘腾云, 等. 高通量转录组测序的数据分析与基因发掘[J]. 江西科学, 2012, 30(5): 607–611.
ZHOU H, ZHANG X, LIU T Y, et al. Data processing and gene discovery of high-throughput transcriptome sequencing[J]. Jiangxi Science, 2012, 30(5): 607–611. (in Chinese) |
| [19] | MORIN R D, O'CONNOR M D, GRIFFITH M, et al. Application of massively parallel sequencing to microRNA profiling and discovery in human embryonic stem cells[J]. Genome Res, 2008, 18(4): 610–621. DOI: 10.1101/gr.7179508 |
| [20] | LI Z, LIU H Y, JIN X L, et al. Expression profiles of microRNAs from lactating and non-lactating bovine mammary glands and identification of miRNA related to lactation[J]. BMC Genomics, 2012, 13(1): 731. DOI: 10.1186/1471-2164-13-731 |
| [21] |
林先滋. 奶牛羊乳腺组织乳脂代谢相关miRNAs的筛选及功能验证[D]. 杨凌: 西北农林科技大学, 2013.
LIN X Z. Screening and functional investigation of miRNAs regulation milk fat metabolism in dairy goat mammary gland[D]. Yangling:Northwest A & F University, 2013. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10183-1016091232.htm |
| [22] |
SAMBROOK J, RUSSELL D W. 分子克隆实验指南[M]. 3版. 黄培堂, 译. 北京: 科学出版社, 2008.
SAMBROOK J, RUSSELL D W. Molecular cloning:a laboratory manual[M]. 3rd ed. HUANG P T, trans. Beijing:Science Press, 2008. (in Chinese) |
| [23] | The Bovine Genome Sequencing and Analysis Consortium, ELSIK C G, TELLAM R L, et al. The genome sequence of taurine cattle:a window to ruminant biology and evolution[J]. Science, 2009, 324(5926): 522–528. DOI: 10.1126/science.1169588 |
| [24] | CARAUX G, PINLOCHE S. PermutMatrix:a graphical environment to arrange gene expression profiles in optimal linear order[J]. Bioinformatics, 2005, 21(7): 1280–1281. DOI: 10.1093/bioinformatics/bti141 |
| [25] | LI R Q, LI Y R, KRISTIANSEN K, et al. SOAP:short oligonucleotide alignment program[J]. Bioinformatics, 2008, 24(5): 713–714. DOI: 10.1093/bioinformatics/btn025 |
| [26] | BARTEL D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–297. DOI: 10.1016/S0092-8674(04)00045-5 |
| [27] | FINNERTY J R, WANG W X, HÉBERT S S. The miR-15/107 group of microRNA genes:evolutionary biology, cellular functions, and roles in human diseases[J]. J Cytol Mol Biol, 2010, 402(3): 491–509. DOI: 10.1016/j.jmb.2010.07.051 |
| [28] | HAYDEN T J, BRENNAN D, QUIRKE K, et al. Xanthine oxidase/dehydrogenase in mammary gland of mouse:relationship to mammogenesis and lactogenesis in vivo and in vitro[J]. J Dairy Res, 1991, 58(4): 401–409. DOI: 10.1017/S0022029900030004 |
| [29] | MCMANAMAN J L, NEVILLE M C. Mammary physiology and milk secretion[J]. Adv Drug Deliv Rev, 2003, 55(5): 629–641. DOI: 10.1016/S0169-409X(03)00033-4 |
| [30] | LIU X, ROBINSON G W, WAGNER K U, et al. Stat5a is mandatory for adult mammary gland development and lactogenesis[J]. Genes Dev, 1997, 11(2): 179–186. DOI: 10.1101/gad.11.2.179 |
| [31] | LIN X Z, LUO J, ZHANG L P, et al. MiR-103 Controls milk fat accumulation in goat (Capra hircus) mammary gland during lactation[J]. PLoS One, 2013, 8(11): e79258. DOI: 10.1371/journal.pone.0079258 |
| [32] | TANAKA T, HANEDA S, IMAKAWA K, et al. A microRNA, miR-101a, controls mammary gland development by regulating cyclooxygenase-2 expression[J]. Differentiation, 2009, 77(2): 181–187. DOI: 10.1016/j.diff.2008.10.001 |
| [33] | WU Z H, HUANG X N, HUANG X, et al. The inhibitory role of Mir-29 in growth of breast cancer cells[J]. J Exp Clin Cancer Res, 2013, 32(1): 98. DOI: 10.1186/1756-9966-32-98 |
| [34] |
边艳杰. miR-29家族对奶牛乳腺上皮细胞泌乳调控机制的研究[D]. 哈尔滨: 东北农业大学, 2015.
BIAN Y J. Regulation mechanism of miR-29 family to lactation of dairy cow mammary epithelial Cells[D]. Harbin:Northeast Agricultural University, 2015.(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10224-1015396362.htm |
| [35] | WATSON C J, BURDON T G. Prolactin signal transduction mechanisms in the mammary gland:the role of the Jak/Stat pathway[J]. Rev Reprod, 1996, 1(1): 1–5. DOI: 10.1530/ror.0.0010001 |
| [36] | FREEMAN M E, KANYICSKA B, LERANT A, et al. Prolactin:structure, function, and regulation of secretion[J]. Physiol Rev, 2000, 80(4): 1523–1631. |



