肉鸡体内脂肪的过度沉积是导致饲料转换率低、繁殖效率和肉品质下降等问题的主要原因,而如何降低脂肪过度沉积已成为目前肉鸡产业亟待解决的难题之一[1]。开展脂肪组织生长发育的分子生物学研究,不但有助于解决肉鸡体内脂肪含量沉积过多问题,还有助于解释人类肥胖和相关代谢疾病的分子发生机制。脂肪性状是受到多种遗传和环境因素控制的复杂性状,其遗传改良迫切需要建立分子水平的育种技术,这是当前国内外肉鸡育种领域的研究热点[2]。国内外研究者利用芯片(SNP芯片、表达谱芯片等)技术、候选基因功能研究、基因组测序等技术方法,深入探讨了肉鸡脂肪性状的遗传基础和规律[3-5]。本课题组长期以来致力于肉鸡腹脂率双向选择系的选育,结合各种分子细胞生物学、遗传学和基因组学技术方法,重点研究了与脂肪代谢、沉积相关的关键转录因子和重要基因的功能及作用机制[6-13]。
RNA编辑(RNA editing)是自然界中存在的一种分子生物学现象,可以通过转录后修饰改变初始转录本,产生与DNA模板之间存在差异的RNA序列。RNA编辑现象1986年首次在锥形虫线粒体中被发现,此后也被发现存在于细胞核、叶绿体和质粒中[14]。随着研究的深入开展,发现RNA编辑主要分为核苷酸的转换、颠换或插入缺失3种类型[15]。RNA编辑涉及多种类型的RNA,包括mRNA[16]、tRNA[17-19]、miRNA[20]和rRNA[21]等。随着高通量测序技术的逐渐成熟和成本下降,RNA编辑现象被大规模的识别[22-23]。哺乳动物的ApoB基因是一个经典范例,RNA编辑会引入终止密码子,影响其在哺乳动物小肠(APOB48) 和肝(APOB100) 内蛋白的长度和功能。最近的研究表明,在人脑组织中发现了大量A-to-Ⅰ RNA编辑的存在,这些位点位于Alu序列,在siRNA的剪切通路以及免疫调节机制中起到重要作用[24]。2011年,B.R.Rosenberg等[25]通过比较野生型小鼠和Apobec1基因敲除小鼠小肠组织的RNA-Seq数据,识别出33个C-to-U RNA编辑位点。2016年,K.Larsen等[26]在猪脑组织的IGFBP7基因中,发现2个A-to-Ⅰ RNA编辑位点(R98G,K95G), 结果表明随着脑组织的老化,RNA编辑水平不断提高。L.Frésard等[14, 23]通过RNA-seq技术识别鸡胚胎、脂肪和肝组织中的RNA编辑,结果发现RNA编辑水平会随着个体年龄的增加而增加。由此可见,RNA编辑在生物体中大量存在,并在个体生长代谢方面也起到重要作用。
东北农业大学肉鸡高、低腹脂双向选择品系从1996年起开始根据腹脂率选育, 至今高、低脂肉鸡品系在腹脂重和腹脂率上存在着显著的差异。本研究以东北农业大学高、低脂双向选择系第十九世代7周龄肉鸡的腹部脂肪组织为试验材料,通过高通量测序和生物信息学分析,筛选出同脂肪组织生长发育相关的RNA编辑位点。肉鸡腹部脂肪RNA编辑的研究对于探究脂肪形成、发育及代谢过程具有借鉴意义。
1 材料与方法 1.1 试验材料东北农业大学肉鸡高、低腹脂双向选择品系经过20个世代的选育,高、低脂系鸡腹脂重和腹脂率存在着显著的差异。本研究以东北农业大学肉鸡高、低腹脂双向选择品系第十九世代鸡群为试验材料,高、低脂系各选取5只公鸡(个体编号为H01-H05以及L01-L05) 采集血液样品。这10只公鸡个体也被用于RNA编辑位点的验证试验。第十九世代时,高脂系肉鸡7周龄腹脂率高达4.7%,而低脂系在相同饲养管理条件下,腹脂率仅为0.7%。7周龄屠宰时收集血液和腹部脂肪组织,分别置于液氮中速冻,-80 ℃分装保存备用。
1.2 基因组DNA和RNA测序 1.2.1 DNA和RNA提取 1.2.1.1 DNA提取从-80 ℃冰箱中取出血液样品,室温融化后取10 μL。严格按照天根血液基因组DNA提取试剂盒(TIANamp Blood DNA Kit)提取血液样品基因组DNA,-20 ℃保存备用。
1-2-1-2 RNA提取TRIzol法提取总RNA。取1 mL TRIzol加入已经研磨(液氮中)好的50 mg脂肪组织样品,充分混匀。加入0.2 mL氯仿,离心后,将水相转移到新离心管中。加入等体积异丙醇用来沉淀水相中的RNA,离心后移去上清。加入经DEPC处理过的75%乙醇1 mL洗涤(可见白色沉淀),RNA沉淀后移去上清,用DEPC水溶解RNA,-80 ℃保存备用。
1.2.2 DNA与RNA测序文库的构建及测序DNA与RNA文库的构建以及测序过程均在北京诺禾致源生物信息科技股份有限公司完成。整个试验严格按照测序合同书执行。
1.2.2.1 DNA测序文库的构建通过Covaris破碎机随机打断DNA样品,回收长度为350 bp的片段。DNA测序文库的构建严格使用说明书推荐的试剂和耗材(NEBNext Ultra DNA Library Prep Kit for Illumina)。DNA片段经末端修复、加ployA尾、加测序接头、纯化、PCR扩增等步骤完成整个文库的制备。构建好的文库通过Illumina HiSeq 4000进行测序。
1.2.2.2 RNA测序文库的构建RNA检测合格后,将RNA打断成150 bp长的片段,用六碱基随机引物(Random hexamers)合成一链cDNA,然后加入缓冲液、dNTPs(dUTP、dATP、dGTP和dCTP)和DNA polymerase Ⅰ合成二链cDNA。随后纯化双链cDNA,再进行末端修复、加ployA尾并连接测序接头,用AMPure XP beads进行片段大小选择,用USER酶降解含有U的cDNA第二链,最后进行PCR富集,得到链特异性cDNA文库。
1.2.2.3 DNA与RNA测序首先,使用Qubit 2.0进行初步定量,稀释文库至1 ng·μL-1。随后使用Agilent 2100检测文库的插入片段大小(Insert size)。插入片段大小符合预期要求后,使用qPCR方法对文库的有效浓度进行准确定量(文库有效浓度>2 nmol·L-1),以确保文库质量。库检合格后,不同文库按照有效浓度及数据需求量,进行Illumina HiSeq 4000测序。
1.3 生物信息学分析测序所得数据通过生物信息学分析的大致流程:首先,对测序结果进行预处理,将原始DNA、RNA测序数据(Raw data)进行整理,通过质量控制(Quality control)后得到高质量的数据(Clean data);然后,将序列进行有参比对(参考鸡基因组为Galgal 4版本)。通过BWA、SAMTOOLs、TopHat2软件分析,以及利用Perl、Python、R语言编写的脚本进行筛选比对后得到RNA编辑候选位点候选集。
1.3.1 基因组测序数据分析DNA序列通过使用BWA软件与鸡的参考基因组(Galgal 4版本)进行比对。测序结果为pair-ends数据,根据BWA软件使用说明书,调用双末端测序数据比对命令。
为了保证信息分析质量,必须对原始测序数据进行过滤,得到高质量reads用于后续分析。具体过滤条件:1) 去除带接头(adaptor)的reads;2) 去除N(N表示无法确定的碱基)的比例>10%的reads;3) 去除低质量碱基(质量值≤20) 含量大于一定比例(低质量碱基数目占整个reads的50%以上)的reads。
1.3.2 RNA测序数据分析首先使用Bowtie 2建立参考基因组索引,TopHat 2能够调用高效短reads,比对软件Bowtie 2将RNA-seq测序结果比对到参考基因组(Galgal 4版本),然后使用SAMTOOLs软件筛选出覆盖数>10的reads,作为后续分析的reads集合。
1.3.3 候选RNA编辑位点的识别 1.3.3.1 SNP与InDel(插入缺失)的识别SAMTOOLs软件的mpileup命令用于对每个个体分别识别SNP(mpileup-m 2-F 0.002-d 1000),并检测长度<50 bp的小片段InDel,然后用ANNOVAR软件对检测出的InDel进行注释,获得VCF格式的文件用于后续分析。
1.3.3.2 RNA序列与DNA序列比对及分类首先将候选RNA编辑位点根据reads覆盖数(覆盖数>5,且RNA的覆盖数>DNA的覆盖数)进行分类,然后根据DNA、RNA的基因型分型,通过TopHat 2软件比较RNA序列与DNA序列,得到RNA编辑候选位点集合文件,包括SNP型编辑位点及插入缺失型编辑位点集合。具体分类:1) DNA为纯合型-RNA为纯合型;2) DNA为纯合型-RNA为杂合型;3) DNA为杂合型-RNA为纯合型;4) 插入缺失的编辑位点。
1.3.4 排除假阳性RNA编辑位点为了使每个编辑位点更加准确,我们通过一些R和Perl脚本进行RNA编辑位点偏差的排除。通过以下筛选条件的限制,可以有效减少假阳性位点的存在[23]。
1) 假阳性偏差的排除:为了降低识别的假阳性,我们保留至少有5条序列覆盖的编辑位点;
2) reads方向的偏差:正向reads/反向reads>0.5的编辑位点被保留;
3) 可能落在剪接接头的偏差:落在外显子第1~8位碱基或者内含子第1~8位碱基则去掉该位点;
4) 重复的偏差:候选编辑位点落在一个单一重复序列(SSR)时,去掉该位点;以候选位点为中心,取上、下游各40 bp连续序列重新与DNA序列比对,如果为单一序列则保留,反之则去掉该位点。
1.4 RNA编辑位点验证试验验证试验内容包括:通过Primer 5.0软件设计特异性引物(博仕生物公司)、反转录PCR、PCR产物Sanger法测序及序列比对。合成的引物按照浓度要求稀释,并进行预试验,确定最适退火温度。反转录所用的试剂均保存在-20 ℃冰箱中。具体流程:1) RNA编辑位点的验证试验选取了用于基因组测序和RNA-seq相同的10只公鸡。分别提取了这10只公鸡腹部脂肪组织的DNA和RNA。2) RNA通过凝胶电泳图确定质量,并使用分光光度计测量RNA浓度。3) 按照ImProm-Ⅱ TM Reverse Transcriptase说明书进行反转录试验,得到cDNA。具体操作流程:a.根据RNA浓度,加入2 μg RNA后,加入oligo(dT)补足至5 μL; b.上述体系放入PCR仪,设置恒温70 ℃ 5 min,取出后待用;c.配制混合体系:6 μL DEPC水,4 μL Buffer,1 μL dNTP,2.5 μL MgCl2,0.5 μL RRI(RNA酶抑制剂),1 μL反转录酶;d. PCR条件42 ℃,时间1 h,反转录产物-20℃冰箱保存。4) 分别混合特异性引物与基因组或cDNA模板,通过PCR扩增特定基因片段,胶回收产物并测序。5) 测序结果通过DNAMAN来比较DNA序列与cDNA序列中存在的差异位点,验证RNA编辑位点与预测结果是否相同。
2 结果 2.1 DNA测序与RNA-seq测序结果分析测序质量分析表明,基因组测序质量相对较高,Q20在94%以上,Q30在88%以上(表 1)。RNA-seq的平均测序深度至少达到了5X,比对率普遍在98%左右(表 2)。测序质量和结果精度较高。
|
|
表 1 高、低脂系肉鸡10个个体的基因组测序结果 Table 1 Genome sequencing results for 10 birds divergently selected for abdominal fat percentage |
|
|
表 2 高、低脂系肉鸡10个个体的RNA-seq结果 Table 2 RNA-seq statistics for 10 birds divergently selected for abdominal fat percentage |
通过TopHat 2软件比对结果,获取基因组和转录组错配位点,作为RNA编辑的候选位点集合。为排除测序错误和比对错误等干扰因素所导致的偏差,本研究进一步利用Python、R、Perl等语言编写脚本程序过滤掉假阳性位点,最终识别高、低脂系肉鸡中的RNA编辑位点共有391个,其中高脂系191个,转换型RNA编辑位点30个,颠换型RNA编辑位点84个,插入缺失型RNA编辑位点77个;低脂系肉鸡中的RNA编辑位点共有200个,包括转换型RNA编辑位点28个,颠换型RNA编辑位点101个,插入缺失型RNA编辑位点71个。使用ANNOVAR软件进一步对这些编辑位点进行功能注释,包括RNA编辑位点所在基因的染色体位置信息、起始位点、终止位点,所在基因功能区域以及基因的相关功能信息等。
2.2.1 转换、颠换型(SNP)RNA编辑类型及分布通过BWA和SAMTOOLs软件识别出基因组中所存在的SNP位点;通过TopHat 2软件分析RNA-seq数据中所存在的RNA-DNA差异位点;利用Bedtools软件识别出SNP位点处发生的RNA编辑现象。根据基因组和转录组错配碱基类型,可以将RNA编辑分为12类,如图 1A中所示。脂肪组织中存在的T-to-G和A-to-T型的RNA编辑数目所占总数比均为13%,而在前人研究中报道较多的A-to-Ⅰ(G)和C-to-U型的RNA编辑在脂肪组织中所占总数比均为7%。通过ANNOVAR软件对RNA编辑位点进行注释,发现这些编辑位点普遍存在于基因间区域和内含子区域,如图 1B所示,可以看出绝大多数RNA编辑位点落在内含子、基因间区域及基因下游区域(内含子区41.91%,基因间区域33.08%,基因下游20.59%,基因上游2.94%和外显子区1.47%)。
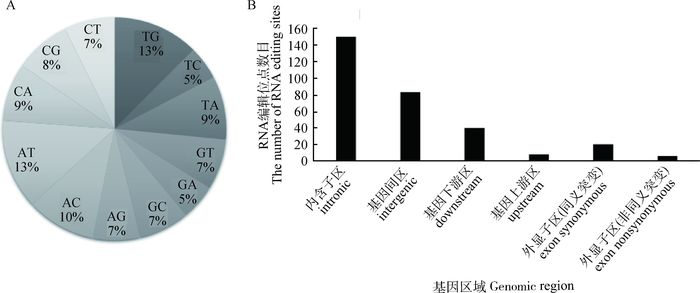
|
A.不同RNA编辑类型的分布比例;B. RNA编辑位点的基因组分布区域 A. Proportion of different RNA editing types; B. Distribution of RNA editing sites in genomic regions 图 1 鸡基因组中RNA编辑位点的分布 Figure 1 Types of RNA editing sites and their distributions in the chicken genome |
根据TopHat2对基因组DNA数据和RNA-seq数据进行比对,建立DNA-RNA差异候选集。通过生物信息学对数据集进行筛选,对于转换或者颠换型RNA编辑位点进行统计。如图 2所示,在5个高脂个体中共识别出114个转换或颠换型RNA编辑位点,有3个位点重复出现了3次(即N=3),分别为G-to-T型RNA编辑、A-to-G型RNA编辑和A-to-T型RNA编辑。在低脂系中共识别出129个RNA编辑位点,有2个RNA编辑位点在3个不同个体中重复出现了3次,其中A-to-C、T-to-G 2种类型的RNA编辑位点数量明显高于其他10种类型,由此可见,颠换型RNA编辑较转换型RNA编辑更易发生。
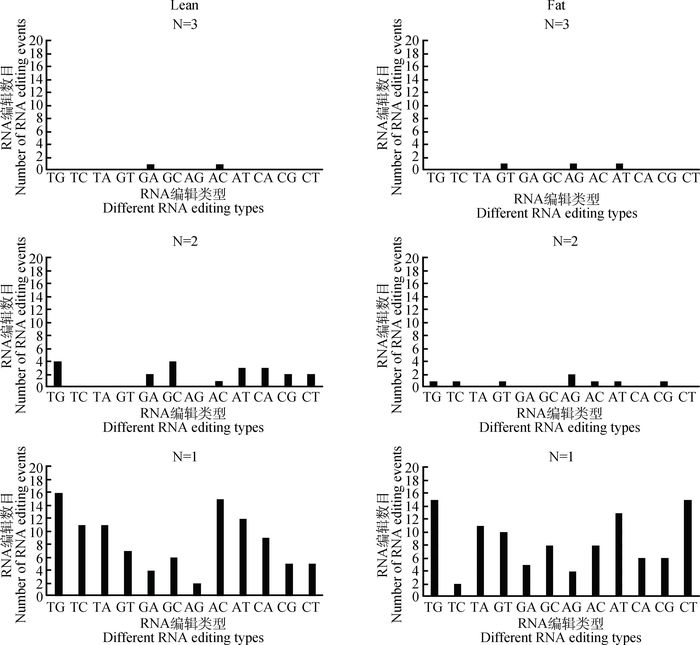
|
N=3表示1个RNA编辑位点在3个个体中均出现;N=2表示1个RNA编辑位点在2个个体中均出现;N=1表示1个RNA编辑位点仅在1个个体中出现 N=3 indicate the RNA editing site is both in 3 chickens; N=2 indicate the RNA editing site is both in 2 chickens; N=1 indicate the RNA editing site is in 1 chicken 图 2 RNA编辑位点个体重复数 Figure 2 Distribution of number of chickens with different RNA editing sites |
RNA编辑包括转换、颠换和插入缺失3种编辑类型。插入缺失型RNA编辑最早在锥形虫线粒体中被发现(插入U型RNA编辑)。根据插入缺失位点的基因型信息,将本研究筛选到的这些位点分为8种编辑类型(图 3)。结果表明,插入缺失型RNA编辑在肉鸡脂肪组织中是普遍存在的,其中插入A型RNA编辑(即InA)在高、低脂系脂肪组织中均存在,数量相对较多,此外插入型RNA编辑数量大于缺失型RNA编辑。插入缺失型RNA编辑在基因中的分布情况与SNP型(单点突变)RNA编辑近似,大多数位于基因间区域(图 4)。高脂系有25个编辑位点在基因间区,低脂系则达到32个。此外,在5个生物学重复中,低脂系肉鸡脂肪组织中的插入缺失型RNA编辑的数量要少于高脂系。在本研究涉及的10个试验个体中,插入缺失型RNA编辑位点在个体中的生物学重复数如图 5所示。结果表明,低脂系肉鸡中有2个RNA编辑位点(InAC、InT)在4个个体中被识别;高脂系中有一个位点(DelC)在4个个体中被识别。由此可见,这3个插入缺失型RNA编辑在肉鸡脂肪组织中具有相对较高的保守性。另外,插入A(InA)型RNA编辑和插入T(U)型RNA编辑具有较高的出现频率。
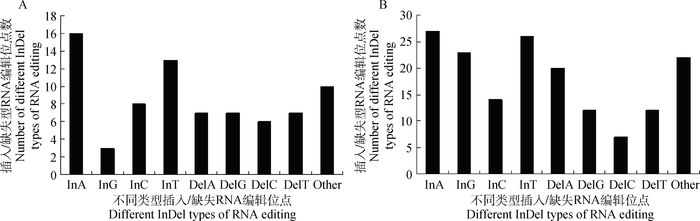
|
A、B.高、低脂系肉鸡:InA.插入A碱基;DelA.缺失A碱基 A, B. Indicate the fat and lean chicken lines, respectively: InA. Insertion A; DelA. Deletion A 图 3 插入缺失型RNA编辑位点的分布比例 Figure 3 Proportion of different insertion and deletion (InDel) types of RNA editing |
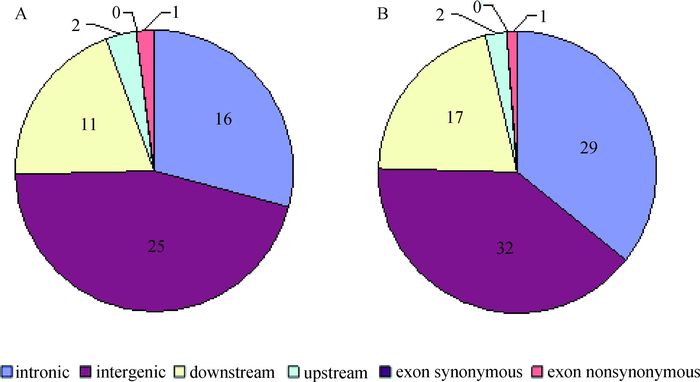
|
A和B分别代表高脂系和低脂系 A and B indicate the fat and lean chicken lines 图 4 插入缺失型RNA编辑在基因区域的分布 Figure 4 InDel RNA editing types distribution in different genomic regions |
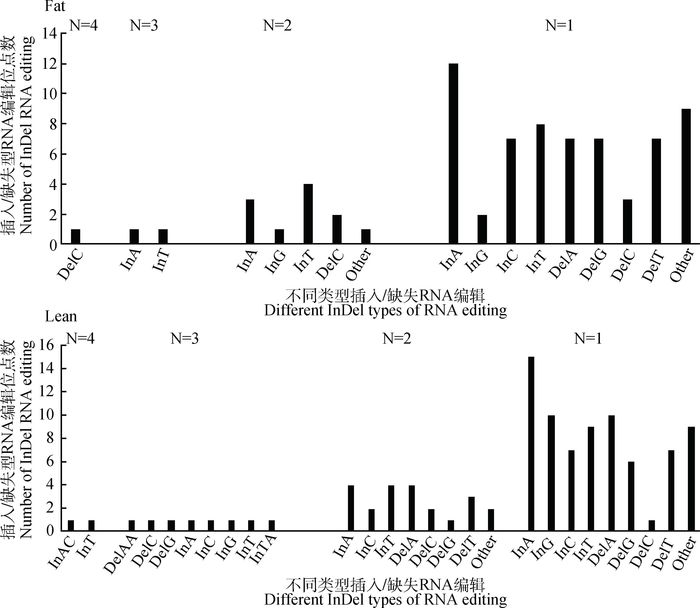
|
图 5 插入缺失型RNA编辑的个体重复数 Figure 5 Number of chickens with InDel RNA editing |
如表 3所示,根据RNA编辑位点在不同个体中重复出现的次数(Replicates),验证了4个编辑位点,这些RNA编辑位点位于可能与脂肪组织生长发育有关的基因内。在识别到的编辑位点中,NDUFS6和FLNB基因中的RNA编辑位点的基本信息与文献报导一致[14]。根据生物信息学预测结果,对4个编辑位点进行Sanger测序验证,测序结果与之前预测结果相同,如图 6所示。其中,FLNB基因在12号染色体第8 909 946 bp位置处发生A-to-Ⅰ RNA编辑,该编辑位点位于外显子区;SLC6A6基因在12号染色体第10 988 203 bp位置发生G-to-T RNA编辑,该位点位于内含子区域;ATP1B3基因在9号染色体第9 399 313 bp位置发生A-to-Ⅰ RNA编辑,该位点位于内含子区域;CMIP基因在11号染色体第15 070 093 bp位置发生InT RNA编辑,该位点位于内含子区域。其中,FLNB、SLC6A6和ATP1B3基因与脂肪形成存在重要关系。显然,大部分RNA编辑位点位于内含子区域,并且SNP型(单点突变)RNA编辑相对数量更多,作用范围更加广泛。
|
|
表 3 鸡脂肪组织中RNA编辑位点的验证 Table 3 Validation of RNA editing sites in chicken adipose tissues |

|
A. FLNB基因;B. SLC6A6基因;C.ATP1B3基因;D. CMIP基因 A.FLNB; B. SLC6A6; C. ATP1B3;D. CMIP 图 6 4个基因中RNA编辑位点的测序验证 Figure 6 Sequencing validation of 4 RNA editing sites |
RNA编辑属于基因序列在转录后发生变异的一种自然现象。RNA编辑不仅能够改变和补充遗传信息,增加基因产物多样性,还与细胞生长、发育和分化具有密切联系[20]。对于畜禽中插入缺失型RNA编辑在以往的研究中鲜有报道。本研究从高、低脂系各取了5只肉公鸡进行RNA编辑位点识别。不同个体中RNA编辑位点的数目和类型稍有差异,这也说明了RNA编辑具有个体差异性的特点。
以往的研究报道表明,存在RNA编辑的基因对脂肪组织的生长发育有不同的影响作用。ATP1B3中的RNA编辑可能会影响脂类物质代谢[27-29],它是ATP酶的β-亚基,位于人类基因组3号染色体的q22-423区域[30-31]。研究表明,ATP1B3可上调淋巴细胞活性,促进IFN-γ、IL-2、IL-4和IL-10的产生[32-33],ATP1B3基因能够调控钠钾离子转运过程,与肠病毒71(EV71) 的复制具有重要联系[29]。敲除和过表达ATP1B3基因证明其对EV71具有抑制作用。脂类物质主要在小肠上段经各种酶及胆汁酸盐的作用,肠病毒71能够干扰这一过程,由此推测ATP1B3基因的RNA编辑可能会影响脂类物质代谢;CMIP是一个新识别出的基因,它编码一个86 ku大小的蛋白,该基因能够合成c-maf诱导蛋白,过量表达会影响细胞内电信号[34-37]。有研究表明,增加c-maf诱导蛋白丰度能够影响足细胞的功能和产生机制[38]。在足细胞发育过程中,转基因鼠选择性表达CMIP基因会产生大量蛋白尿症状。另外,通过RNAi沉默CMIP基因能够防止尿蛋白的产生[37];在SLC6A6基因间区存在一个G-to-T型RNA编辑位点,它是一种牛磺酸转运蛋白基因,能够维持组织中牛磺酸的含量[39]。牛磺酸作为一种渗透调节和抗氧化物质,在视网膜神经细胞保护过程中起重要作用[40-43],并且能够促进脂肪和脂溶性物质的消化吸收, 促进糖类、蛋白质和矿物质元素等代谢, 以及对神经、内分泌、生殖和免疫系统等有调节作用[44-46]。一些研究发现, 在胰岛素依赖型糖尿病患者体内, 血浆和血小板中牛磺酸含量均明显下降, 而膳食补充牛磺酸可明显改善这种糖尿病引起的牛磺酸含量下降情况[47]。NDUFS6基因在我们所选的10个个体中均被发现,该基因是肉鸡腹部脂肪组织中保守发生RNA编辑的基因,对于脂类物质代谢和骨骼的发育具有一定影响[14, 48]。另外,FLNB基因属于纤丝状肌动蛋白基因家族成员之一,该家族基因能够定义细胞形状,调节细胞间作用力及影响信息传递,这些过程是各个器官正常生长发育所必需的[49-53]。该基因与骨骼和脂肪组织生长发育有着重要联系,特异性地敲除FLNB基因会导致心血管或者骨骼生长发育过程中的功能紊乱[54-55]。
关于典型的A-to-Ⅰ RNA及C-to-U RNA编辑的报道已有很多[14, 24-26, 28],然而非典型的RNA编辑发生机制仍鲜有报道,尤其对于插入缺失型RNA编辑的发生机制以及对肉鸡脂肪组织的影响仍需要进一步去思考和研究。另外,通过在鸡不同周龄的胚胎、脂肪和肝组织中识别RNA编辑位点,研究发现,RNA编辑水平会随着个体年龄的增加而增加,这种趋势受到基因型、年龄、饲喂条件、性别的影响[14]。相同的情况在不同年龄人脑组织中也同样被发现[24]。由此可见,RNA编辑对胚胎发育和个体生长发育可能有着重要作用。
本研究在转录组范围内进行了肉鸡脂肪组织中各类RNA编辑位点的系统识别,为开发从头预测编辑位点的算法提供了原始数据,也为进一步研究肉鸡脂肪组织中RNA编辑的发生机制和RNA编辑的分子生物学功能做了有价值的试验铺垫。
4 结论本研究以东北农业大学高、低脂双向选择系肉鸡脂肪组织为试验材料,通过高通量测序以及RNA-seq,以生物信息学预测和分子生物学试验相结合的方法初步发掘了与鸡脂肪性状相关的RNA编辑位点。主要结果:(1) 通过生物信息学手段共识别高、低脂系肉鸡腹部脂肪组织的RNA编辑位点391个。(2) 在高、低脂系肉鸡腹部脂肪组织中分别识别出77和71个插入缺失型RAN编辑位点。(3) 测序验证了4个RNA编辑位点,为进一步研究其影响脂肪组织生长发育的功能奠定了基础。
| [1] |
史铭欣, 荣恩光, 周纬男, 等. 高、低脂鸡脂肪组织ATGL基因的表达及其激素调控的差异分析[J]. 畜牧兽医学报, 2013, 44(12): 1874–1881.
SHI M X, RONG E G, ZHOU W N, et al. Adipose tissue expression and hormonal regulation of ATGL gene in the fat and lean broiler lines[J]. Acta Veterinaria et Zootechnica Sinica, 2013, 44(12): 1874–1881. (in Chinese) |
| [2] |
李辉, 杜志强, 王守志, 等. 白羽快大型肉鸡育种的过去、现在和将来[J]. 中国家禽, 2016, 38(19): 1–8.
LI H, DU Z Q, WANG S Z, et al. The past, present and future of white feather fast and large poultry breeding[J]. China Poultry, 2016, 38(19): 1–8. (in Chinese) |
| [3] | FU R Q, LIU R R, ZHAO G P, et al. Expression profiles of key transcription factors involved in lipid metabolism in Beijing-You chickens[J]. Gene, 2014, 537(1): 120–125. DOI: 10.1016/j.gene.2013.07.109 |
| [4] |
王彦博, 王宁, 王丽, 等. 鸡Perilipin1抗血清制备及组织表达特性分析[J]. 畜牧兽医学报, 2011, 42(3): 349–355.
WANG Y B, WANG N, WANG L, et al. Preparation of antiserums against chicken perilipin1 and tissue expression analyses of Perilipin1[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(3): 349–355. (in Chinese) |
| [5] |
王海霞, 张志威, 贺綦, 等. 鸡KLF3基因的表达规律及其对脂肪细胞分化的影响研究[J]. 畜牧兽医学报, 2015, 46(1): 26–31.
WANG H X, ZHANG Z W, HE Q, et al. Expression pattern of chicken krüppel-like factor 3 gene and its effect on adipocyte differentiation[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(1): 26–31. (in Chinese) |
| [6] | SUN Y N, GAO Y, QIAO S P, et al. Epigenetic DNA methylation in the promoters of Peroxisome Proliferator-Activated Receptor γ in chicken lines divergently selected for fatness[J]. J Anim Sci, 2014, 92(1): 48–53. DOI: 10.2527/jas.2013-6962 |
| [7] |
王启贵, 高广亮, 马广伟, 等. 鸡肝脏胆汁酸结合蛋白基因启动子活性分析[J]. 东北农业大学学报, 2014, 45(4): 88–93.
WANG Q G, GAO G L, MA G W, et al. Activity analysis of chicken liver bile acid binding protein gene promoter[J]. Journal of Northeast Agricultural University, 2014, 45(4): 88–93. (in Chinese) |
| [8] |
于莹莹, 乔书培, 孙婴宁, 等. 鸡IGFBP2基因3'UTR区1196C>A单核苷酸多态性的功能性鉴定及分析[J]. 生物化学与生物物理进展, 2014, 41(11): 1163–1172.
YU Y Y, QIAO S P, SUN Y N, et al. Identification and analysis of a functional SNP 1196C>A in 3'UTR of chicken IGFBP2 gene[J]. Progress in Biochemistry and Biophysics, 2014, 41(11): 1163–1172. (in Chinese) |
| [9] |
牟彦双, 王宇祥, 李辉. 鸡类胰岛素生长因子结合蛋白2基因编码区克隆及表达[J]. 畜牧与兽医, 2014, 46(7): 77–80.
MU Y S, WANG Y X, LI H. Cloning and expression of coding region of insulin-like growth factor binding protein 2 gene in chicken[J]. Animal Husbandry & Veterinary Medicine, 2014, 46(7): 77–80. (in Chinese) |
| [10] |
李辉, 赵士初, 黄学涛, 等. ACOX3基因多态性对鸡体脂沉积的影响[J]. 东北农业大学学报, 2014, 45(10): 42–48, 85.
LI H, ZHAO S C, HUANG X T, et al. Effect of ACOX3 gene polymorphisms on body fat deposition in chickens[J]. Journal of Northeast Agricultural University, 2014, 45(10): 42–48, 85. DOI: 10.3969/j.issn.1005-9369.2014.10.007 (in Chinese) |
| [11] |
薛倩, 王金玉, 张跟喜, 等. 京海黄鸡甲状腺激素应答蛋白Spot14α基因外显子1多态性与屠体和腹脂性状的相关性分析[J]. 中国畜牧兽医, 2014, 41(7): 169–174.
XUE Q, WANG J Y, ZHANG G X, et al. Relationship between polymorphisms of exon 1 of THRSPα gene and the carcass and abdominal fat traits in Jinghai Yellow chicken[J]. China Animal Husbandry & Veterinary Medicine, 2014, 41(7): 169–174. (in Chinese) |
| [12] |
周艳, 雷秋霞, 韩海霞, 等. 脂滴包被蛋白基因(PLIN)多态性与鸡胴体及脂肪性状的相关性[J]. 农业生物技术学报, 2014, 22(8): 1001–1008.
ZHOU Y, LEI Q X, HAN H X, et al. Association on single nucleotide polymorphism of perilipin gene (PLIN) with carcass and fatness traits in Luqin chicken (Gallus gallus)[J]. Journal of Agricultural Biotechnology, 2014, 22(8): 1001–1008. (in Chinese) |
| [13] |
高广亮, 关天竹, 王海威, 等. A-FABP基因与肉鸡生长和体组成性状的相关分析[J]. 中国畜牧杂志, 2014, 50(15): 28–32.
GAO G L, GUAN T Z, WANG H W, et al. Association between chicken A-FABP gene polymorphisms and growth and body composition traits[J]. Chinese Journal of Animal Science, 2014, 50(15): 28–32. DOI: 10.3969/j.issn.0258-7033.2014.15.007 (in Chinese) |
| [14] | ROUX P F, FRÉSARD L, BOUTIN M, et al. The extent of mRNA editing is limited in chicken liver and adipose, but impacted by tissular context, genotype, age, and feeding as exemplified with a conserved edited site in COG3[J]. G3(Bethesda), 2015, 6(2): 321–335. |
| [15] | KNOOP V. When you can't trust the DNA:RNA editing changes transcript sequences[J]. Cell Mol Life Sci, 2011, 68(4): 567–586. DOI: 10.1007/s00018-010-0538-9 |
| [16] | BÖRNER G V, MvRL M, JANKE A, et al. RNA editing changes the identity of a mitochondrial tRNA in marsupials[J]. EMBO J, 1996, 15(21): 5949–5957. |
| [17] | GOTT J M, SOMERLOT B H, GRAY M W. Two forms of RNA editing are required for tRNA maturation in Physarum mitochondria[J]. RNA, 2010, 16(3): 482–488. DOI: 10.1261/rna.1958810 |
| [18] | TORRES A G, PIÑEYRO D, FILONAVA L, et al. A-to-Ⅰ editing on tRNAs:biochemical, biological and evolutionary implications[J]. FEBS Lett, 2014, 588(23): 4279–4286. DOI: 10.1016/j.febslet.2014.09.025 |
| [19] | WARNEFORS M, LIECHTI A, HALBERT J, et al. Conserved microRNA editing in mammalian evolution, development and disease[J]. Genome Biol, 2014, 15(6): R83. DOI: 10.1186/gb-2014-15-6-r83 |
| [20] | EIFLER T, POKHAREL S, BEAL P A. RNA-Seq analysis identifies a novel set of editing substrates for human ADAR2 present in Saccharomyces cerevisiae[J]. Biochemistry, 2013, 52(45): 7857–7869. DOI: 10.1021/bi4006539 |
| [21] | KANG L, LIU X Q, GONG Z L, et al. Genome-wide identification of RNA editing in hepatocellular carcinoma[J]. Genomics, 2015, 105(2): 76–82. DOI: 10.1016/j.ygeno.2014.11.005 |
| [22] |
张德新, 赵澎涛, 赵青川, 等. 小鼠肝脏RNA编辑酶ADAR14种剪切体:克隆、表达及功能分析[J]. 生物化学与生物物理进展, 2006, 33(7): 635–640.
ZHANG D X, ZHAO P T, ZHAO Q C, et al. Functional characterization and comparison of four splicing alternatives of mouse Liver A-to-Ⅰ RNA editing enzyme ADAR1[J]. Progress in Biochemistry and Biophysics, 2006, 33(7): 635–640. (in Chinese) |
| [23] | FRÉSARD L, LEROUX S, ROUX P F, et al. Genome-wide characterization of RNA editing in chicken embryos reveals common features among vertebrates[J]. PLoS One, 2015, 10(5): e0126776. DOI: 10.1371/journal.pone.0126776 |
| [24] | HWANG T, PARK C K, LEUNG A K L, et al. Dynamic regulation of RNA editing in human brain development and disease[J]. Nat Neurosci, 2016, 19(8): 1093–1099. DOI: 10.1038/nn.4337 |
| [25] | ROSENBERG B R, HAMILTON C E, MWANGI M M, et al. Transcriptome-wide sequencing reveals numerous APOBEC1 mRNA-editing targets in transcript 3'UTRs[J]. Nat Struct Mol Biol, 2011, 18(2): 230–236. DOI: 10.1038/nsmb.1975 |
| [26] | LARSEN K, KRISTENSEN K K, MOMENI J, et al. A-to-Ⅰ RNA editing of the IGFBP7 transcript increases during aging in porcine brain tissues[J]. Biochem Biophys Res Commun, 2016, 479(3): 596–601. DOI: 10.1016/j.bbrc.2016.09.125 |
| [27] | GOU L, SUN B, SHANG Z, et al. Comparison of adipose tissue cellularity in chicken lines divergently selected for fatness[J]. Poult Sci, 2011, 90(9): 2024–2034. DOI: 10.3382/ps.2010-00863 |
| [28] | NAKAMUTA M, TSAI A, CHAN L, et al. Sequence elements required for apolipoprotein B mRNA editing enhancement activity from chicken enterocytes[J]. Biochem Biophys Res Commun, 1999, 254(3): 744–750. DOI: 10.1006/bbrc.1998.9963 |
| [29] | LU Y F, HOU H Y, WANG F, et al. ATP1B3:a virus-induced host factor against EV71 replication by up-regulating the production of type-Ⅰ interferons[J]. Virology, 2016, 496: 28–34. DOI: 10.1016/j.virol.2016.05.013 |
| [30] | BESIRLI C G, GONG T W L, LOMAX M I. The Atp1b3 gene for Na, K-ATPase β3 subunit maps to mouse Chromosome 9, and a related gene, Atp1b3-rs, maps to mouse Chromosome 3[J]. Mamm Genome, 1998, 9(2): 171–172. DOI: 10.1007/s003359900713 |
| [31] | MALIK N, CANFIELD V, SANCHEZ-WATTS G, et al. Structural organization and chromosomal localization of the human Na, K-ATPase β3 subunit gene and pseudogene[J]. Mamm Genome, 1998, 9(2): 136–143. DOI: 10.1007/s003359900704 |
| [32] | CHIAMPANICHAYAKUL S, SZEKERES A, KHUNKAEWLA P, et al. Engagement of Na, K-ATPase β3 subunit by a specific mAb suppresses T and B lymphocyte activation[J]. Int Immunol, 2002, 14(12): 1407–1414. DOI: 10.1093/intimm/dxf112 |
| [33] | CHRUEWKAMLOW N, PATA S, MAHASONGKRAM K, et al. β3 subunit of Na, K ATPase regulates T cell activation with no involvement of Na, K ATPase activity[J]. Immunobiology, 2015, 220(5): 634–640. DOI: 10.1016/j.imbio.2014.11.017 |
| [34] | ZHANG S Y, KAMAL M, DAHAN K, et al. c-mip impairs podocyte proximal signaling and induces heavy proteinuria[J]. Sci Signal, 2010, 3(122): ra39. |
| [35] | YU L X, LIN Q X, FENG J H, et al. Inhibition of nephrin activation by c-mip through Csk-Cbp-Fyn axis plays a critical role in Angiotensin Ⅱ-induced podocyte damage[J]. Cell Signal, 2013, 25(3): 581–588. DOI: 10.1016/j.cellsig.2012.11.017 |
| [36] | LIU Y, SU L, LIN Q, et al. Induction of c-mip by IL-17 plays an important role in adriamycin-induced podocyte damage[J]. Cell Physiol Biochem, 2015, 36(4): 1274–1290. DOI: 10.1159/000430296 |
| [37] | NEWBURY D F, WINCHESTER L, ADDIS L, et al. CMIP and ATP2C2 modulate phonological short-term memory in language impairment[J]. Am J Hum Genet, 2009, 85(2): 264–272. DOI: 10.1016/j.ajhg.2009.07.004 |
| [38] | SAHALI D, SENDEYO K, MANGIER M, et al. Immunopathogenesis of idiopathic nephrotic syndrome with relapse[J]. Semin Immunopathol, 2014, 36(4): 421–429. DOI: 10.1007/s00281-013-0415-3 |
| [39] | YAHARA T, TACHIKAWA M, AKANUMA S I, et al. Amino acid residues involved in the substrate specificity of TauT/SLC6A6 for taurine and γ-aminobutyric acid[J]. Biol Pharm Bull, 2014, 37(5): 817–825. DOI: 10.1248/bpb.b13-00991 |
| [40] | TOMI M, TERAYAMA T, ISOBE T, et al. Function and regulation of taurine transport at the inner blood-retinal barrier[J]. Microvasc Res, 2007, 73(2): 100–106. DOI: 10.1016/j.mvr.2006.10.003 |
| [41] | ANDO D, KUBO Y, AKANUMA S I, et al. Function and regulation of taurine transport in Müller cells under osmotic stress[J]. Neurochem Int, 2012, 60(6): 597–604. DOI: 10.1016/j.neuint.2012.02.018 |
| [42] | MIYAMOTO Y, KULANTHAIVEL P, LEIBACH F H, et al. Taurine uptake in apical membrane vesicles from the bovine retinal pigment epithelium[J]. Invest Ophthalmol Vis Sci, 1991, 32(9): 2542–2551. |
| [43] | EL-SHERBENY A, NAGGAR H, MIYAUCHI S, et al. Osmoregulation of taurine transporter function and expression in retinal pigment epithelial, ganglion, and müller cells[J]. Invest Ophthalmol Vis Sci, 2004, 45(2): 694–701. DOI: 10.1167/iovs.03-0503 |
| [44] | MOCHIZUKI T, SATSU H, NAKANO T, et al. Regulation of the human taurine transporter by TNF-α and an anti-inflammatory function of taurine in human intestinal Caco-2- cells[J]. Biofactors, 2004, 21(1-4): 141–144. DOI: 10.1002/biof.v21:1/4 |
| [45] |
陈宠霞, 金一. 添加牛磺酸对兔精液液态保存质量参数的影响[J]. 畜牧与兽医, 2006, 38(8): 25–27.
CHEN C X, JIN Y. Effect of taurine supplementation on quality of liquid-stored semen of rabbits[J]. Animal Husbandry & Veterinary Medicine, 2006, 38(8): 25–27. (in Chinese) |
| [46] | STURMAN J A. Taurine in development[J]. J Nutr, 1988, 118(10): 1169–1176. |
| [47] | RASMUSSEN R N, LAGUNAS C, PLUM J, et al. Interaction of GABA-mimetics with the taurine transporter (TauT, Slc6a6) in hyperosmotic treated Caco-2, LLC-PK1 and rat renal SKPT cells[J]. Eur J Pharm Sci, 2016, 82: 138–146. DOI: 10.1016/j.ejps.2015.11.020 |
| [48] | KMITA K, WIRTH C, WARNAU J, et al. Accessory NUMM (NDUFS6) subunit harbors a Zn-binding site and is essential for biogenesis of mitochondrial complex I[J]. Proc Natl Acad Sci USA, 2015, 112(18): 5685–5690. DOI: 10.1073/pnas.1424353112 |
| [49] | ARBER S, BARBAYANNIS F A, HANSER H, et al. Regulation of actin dynamics through phosphorylation of cofilin by LIM-kinase[J]. Nature, 1998, 393(6687): 805–809. DOI: 10.1038/31729 |
| [50] | GARDEL M L, SCHNEIDER I C, ARATYN-SCHAUS Y, et al. Mechanical integration of actin and adhesion dynamics in cell migration[J]. Annu Rev Cell Dev Biol, 2010, 26: 315–333. DOI: 10.1146/annurev.cellbio.011209.122036 |
| [51] | OLSON E N, NORDHEIM A. Linking actin dynamics and gene transcription to drive cellular motile functions[J]. Nat Rev Mol Cell Biol, 2010, 11(5): 353–365. DOI: 10.1038/nrm2890 |
| [52] | PARSONS J T, HORWITZ A R, SCHWARTZ M A. Cell adhesion:integrating cytoskeletal dynamics and cellular tension[J]. Nat Rev Mol Cell Biol, 2010, 11(9): 633–643. DOI: 10.1038/nrm2957 |
| [53] | WILSON S G, JONES M R, MULLIN B H, et al. Common sequence variation in FLNB regulates bone structure in women in the general population and FLNB mRNA expression in osteoblasts in vitro[J]. J Bone Miner Res, 2009, 24(12): 1989–1997. DOI: 10.1359/jbmr.090530 |
| [54] | FENG Y Y, CHEN M H, MOSKOWITZ I P, et al. Filamin A (FLNA) is required for cell-cell contact in vascular development and cardiac morphogenesis[J]. Proc Natl Acad Sci USA, 2006, 103(52): 19836–19841. DOI: 10.1073/pnas.0609628104 |
| [55] | ZHAO Y T, SHAPIRO S S, ETO M. F-actin clustering and cell dysmotility induced by the pathological W148R missense mutation of filamin B at the actin-binding domain[J]. Am J Physiol Cell Physiol, 2016, 310(1): C89–C98. |



