解整合素-金属蛋白酶家族(A disintegrin and metalloproteinase,ADAMs)是一种瞄定在细胞膜上的蛋白酶,该酶具有在细胞膜上切割跨膜蛋白的酶活性,因而被称为“脱落酶”,同时该家族蛋白在细胞黏附、细胞迁移、白细胞募集、炎症反应等方面也发挥重要的作用[1]。ADAM10基因作为ADAMs家族中的一员,最早是从牛的脑髓鞘膜中分离纯化所得,广泛表达于多种生物体内,并均具有相似的生物学功能。从胚胎到成体,ADAM10在大部分的组织中都有表达[2-4]。人ADAM10基因结构研究的比较清楚,其编码的蛋白包含748个氨基酸的跨膜蛋白,其由多个功能域组成,包括信号肽序列、前引导序列、金属蛋白酶域、解聚素结构域、富含半胱氨酸结构域、表皮生长因子样序列、膜结构域及胞浆尾部[5]。
ADAM10与ADAM成员有类似的功能,ADAM10蛋白活化后锚定在细胞膜表面,水解各种底物,参与信号通路调控过程。ADAM10蛋白的底物主要包括NOTCH信号通路、细胞黏附因子CD44、肿瘤坏死因子TNF-α和多种细胞因子等。最近研究发现,ADAM10基因敲除小鼠脑中神经元祖细胞显著减少,进一步研究发现,Notch-1和下游信号通路的表达显著受到抑制,说明ADAM10靶向Notch-1信号通路对成年小鼠脑中的神经元维持具有显著的作用[6]。在繁殖方面,ADAM10通过NOTCH信号通路招募卵泡祖细胞调控卵泡生成[7]。而在多种肿瘤细胞中,ADAM10能够切割细胞表面黏附蛋白促进肿瘤细胞的迁移与侵袭[8-10]。针对冠状动脉支架内再狭窄的研究发现,在糖尿病和非糖尿病小型猪模型中支架内再狭窄内膜ADAM10基因表达量要比非支架内再狭窄内膜高,进一步研究发现,ADAM10基因过表达可通过Notch-1和Notch-3信号通路促进血管平滑肌细胞的生长与迁移[11]。同样,ADAM10通过其脱落酶活性促进TNF-α的分泌激活内源性细胞信号因子通路,促进细胞增殖[12-13]。
ADAM10基因结构和功能在人上已研究的较多,而对于猪ADAM10基因不同剪接体的结构和功能鲜有报道。鉴于此,本研究首先采用分子生物学方法从猪PK15肾细胞克隆获得ADAM10基因不同剪接体编码区序列,并在脾和肾中验证了2种剪接体的存在,然后利用生物信息学在线工具与软件分别对其序列进行分析,最后利用半定量PCR方法检测猪ADAM10基因不同剪接体在组织中的表达差异,为进一步研究猪ADAM10基因不同剪接体的功能提供理论基础与依据。
1 材料与方法 1.1 样品采集成年纯种长白母猪来自杭州大观山种猪育种有限公司,分别采集12个组织:心、肝、脾、肺、肾、乳腺、腿肌、输卵管、卵巢、子宫、小肠、胃,所有组织迅速置于液氮中冷冻,并带回实验室-80 ℃保存。
1.2 总RNA提取与cDNA合成利用Trizol试剂(Invitrogen,美国)提取猪不同组织和肾上皮PK15细胞的总RNA。分别用1.2%琼脂糖凝胶电泳和NanoDrop 2000分光光度计(Thermo Scientific, 美国)检测总RNA的完整性和浓度。利用HiFiScript cDNA Synthesis kit(北京康为世纪生物科技有限公司)进行cDNA第一链的合成。cDNA合成体系:总RNA 1 μg,primer mix 2 μL,5×RT Buffer 4 μL,dNTP mix 4 μL,DTT 2 μL,HiFiScript 1 μL,RNase-Free water补足到20 μL。反应条件:42 ℃ 15 min,85 ℃ 5 min;-20 ℃保存备用。
1.3 引物设计根据NCBI数据库中猪ADAM10基因(登录号:NM_001130531.1) 序列,利用Primer Premier 5.0引物设计软件设计3对引物(ADAM10-CDS用来扩增猪ADAM10基因的CDS序列,ADAM10-1和ADAM10-2作为半定量引物),以猪RPL32基因作为内参基因(登录号:NM_001001636),引物序列见表 1。引物均由上海英骏生物技术有限公司合成。
|
|
表 1 猪ADAM10和RPL32基因的引物信息 Table 1 Primer information of the porcine ADAM10 and RPL32 genes |
利用PK15细胞的cDNA为模板进行PCR反应,反应体系为10 μL:2×Es Taq MasterMix(北京康为世纪生物科技有限公司)5 μL,cDNA 1 μL,上、下游引物(10 mmol·mL-1)各0.3 μL,ddH2O 3.4 μL。PCR扩增程序:95 ℃预变性3 min;94 ℃变性30 s,退火30 s (退火温度见表 1),72 ℃延伸2.5 min,35个循环;72 ℃终延伸5 min。最后用1.2%琼脂糖凝胶电泳检测PCR产物将回收的PCR产物纯化,再与pMD18-T载体(宝生物工程大连有限公司)连接,转化DH5α大肠杆菌感受态细胞后,菌液PCR鉴定阳性克隆,然后送铂尚生物技术有限公司测序。
为进一步在猪的组织中验证猪ADAM10基因的选择性剪接,分别以猪脾和肺组织cDNA为模板,用ADAM10基因CDS引物进行PCR扩增,1.2%琼脂糖凝胶电泳检测,进一步核实剪接型ADAM10基因的存在。
1.5 生物信息学分析DNAMAN软件进行DNA序列比对;ExPASy-ProtParam tool在线系统分析蛋白氨基酸组成、理论分子质量和等电点等;SignalP-4.1预测信号肽;Protscale预测蛋白质疏水性;TMHMM预测跨膜区结构;Conserved Domains在线软件分析蛋白质的保守结构域;SOPMA在线软件对蛋白质的二级结构进行预测分析;利用Phyre2程序根据基因编码的氨基酸序列推测其三维结构。预测分析方法及网址见表 2。
|
|
表 2 蛋白结构功能预测方法及相关网址 Table 2 The methods and websites of protein structure and function prediction |
为了研究ADAM10基因在进化上的特点,选取NCBI中人、小鼠、大鼠、山羊、鸡、非洲爪蟾以及斑马鱼等几个物种的相关序列进行多序列比对,并用MEGA 5.0软件和选择邻接法(Neighbor joining)建立系统进化树。
1.6 猪ADAM10基因在不同组织中的表达分析选择猪RPL32基因作为内参,分别设计用于扩增猪ADAM10基因完整型和剪接体特异性引物(表 1),利用半定量PCR检测猪ADAM10基因及其剪接体在各个组织中的表达量。反应体系:2×Es Taq MasterMix 5 μL,cDNA 1 μL,上、下游引物(10 mmol·mL-1)各0.3 μL,ddH2O 3.4 μL。PCR扩增程序:95 ℃预变性3 min;94 ℃变性30 s,退火30 s (退火温度见表 1),72 ℃延伸15 s,34个循环;72 ℃终延伸5 min。PCR扩增结束后,各取5 μL产物经1.2%琼脂糖凝胶电泳进行检测。
2 结果 2.1 猪ADAM10基因cDNA的克隆与鉴定利用PK15细胞的cDNA为模板,用ADAM10-CDS引物扩增ADAM10基因的整个编码区。PCR扩增产物经1.2%琼脂糖凝胶电泳检测,从图 1可以看出,扩增出的主带大小为2 200 bp左右,与预计片段的大小相符;同时在1 400 bp左右的位置出现一条微弱的条带,推测是ADAM10基因的选择性剪接产物。
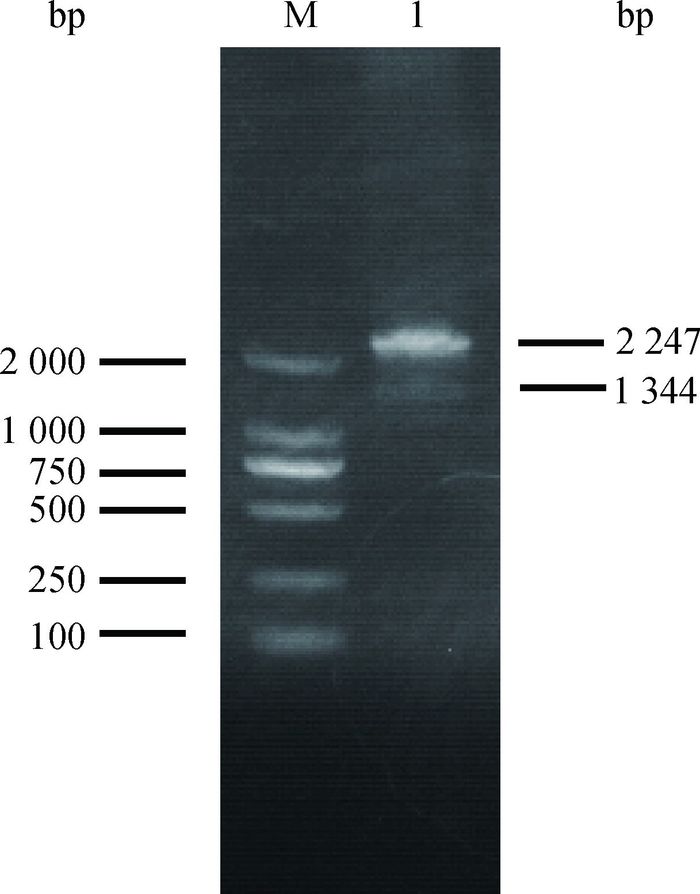
|
M. DNA相对分子质量标准;1. ADAM10 PCR产物 M. DL2000 marker; 1. PCR product of ADAM10 图 1 猪PK15细胞中ADAM10基因编码区的扩增 Figure 1 Amplification of ADAM10 coding sequence in PK15 cells |
将猪ADAM10基因编码区扩增产物进行切胶回收后连接到pMD18-T载体,转化大肠杆菌DH5α感受态细胞,涂布于培养皿进行培养,利用菌液PCR初步确定阳性菌。最后通过测序获得全长CDS(完整型)序列长度为2 247 bp,剪接型全长CDS序列长度为1 344 bp。将剪接体CDS序列提交到GenBank,登录号为KY693666。
为了验证ADAM10基因的剪接型确实在机体中存在,用RT-PCR检测其在猪组织中的表达情况,结果显示,ADAM10的剪接型mRNA在猪的肺中特异高表达、而完整型mRNA在脾中高表达(图 2)。
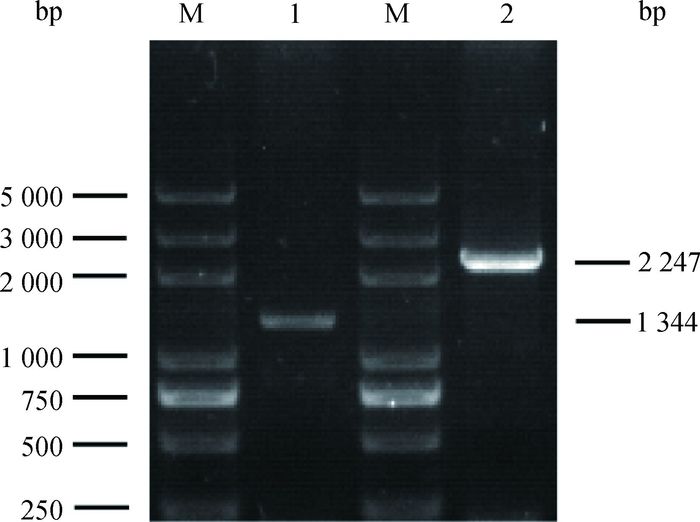
|
1.肺;2.脾;M. DNA相对分子质量标准 1. Lung; 2. Spleen; M. DL5000 marker 图 2 猪ADAM10基因选择性剪接的检测 Figure 2 Detection of alternative splicing of porcine ADAM10 |
猪ADAM10完整型的CDS长2 247 bp,编码748个氨基酸;剪接型的CDS长1 344 bp,编码447个氨基酸。利用Ensembl genome browser数据库中猪ADAM10 cDNA序列(ID: ENSSSCG00000004595) 和UCSC Genome Browser数据库对猪ADAM10基因的外显子进行组装拼接,并经序列比对分析,猪ADAM10的完整型cDNA由16个外显子组成;剪接型的ADAM10基因缺失外显子2、3、4、5、6、7和部分外显子8(总长903 bp),没有发生移码突变,和完整型CDS相比,共缺失301个氨基酸(图 3)。

|
A.猪ADAM10基因完整型和剪接型CDS比对;B.猪ADAM10基因完整型和剪接型基因结构图:方框代表外显子,其中含有斜线的方框表示剪接体缺失整个或部分外显子。ADAM10-1.完整型;ADAM10-2.剪接型,下同 A. CDS sequence alignments of alternative splicing of porcine ADAM10 gene; B. The schematic diagram for ADAM10 gene structure of alternative splicing:The boxes indicate the exons. The boxes containing diagonal lines indicate that the variant does not include the partial sequence of an exon or the whole exon. ADAM10-1. Complete type; ADAM10-2. Splicing type, the same as below 图 3 猪ADAM10基因完整型和剪接型cDNA比对结果和基因结构图 Figure 3 cDNA sequence alignments and schematic diagram for the gene structure of alternative splicing of porcine ADAM10 |
BLAST同源性比对结果显示,猪与人(NM_001110.3)、小鼠(NM_007399.4)、大鼠(NM_019254.1)、原鸡(NM_204261.2)、山羊(XM_018054271.1)、非洲爪蟾(NM_001044404.1) 和斑马鱼(XM_005168897.3)ADAM10基因的编码区核苷酸同源性分别为94%、90%、90%、84%、95%、76%、和72%;氨基酸序列同源性分别为96%、94%、94%、89%、96%、82%、和75%,结果表明,在不同的物种中ADAM10蛋白具有较高的保守性。猪ADAM10氨基酸序列和其他物种比对结果见图 4。

|
氨基酸序列一致部分由黑色标记;氨基酸序列出现替换的地方由灰色或白色标记 Identical amino acid sequences are marked in black, and amino acid sequences with substitutions are marked in grey or white 图 4 猪ADAM10氨基酸序列与其他物种的同源性比较 Figure 4 Identical alignments of amino acid sequences of porcine ADAM10 with other species |
利用ProtParam在线软件对猪ADAM10蛋白进行理化性质分析,结果显示,ADAM10完整型蛋白的分子量为84.3 ku,理论等电点pI为8.12,含有88个带负电荷的氨基酸残基(Asp+Glu)和94个带正电荷的氨基酸残基(Arg+Lys),体外半衰期为30 h,不稳定系数为37.52,说明该蛋白比较稳定,总平均亲水性为-0.619,表明该蛋白具有较强的亲水性。通过在线程序ProtScale对猪ADAM10完整型和剪接型蛋白进行疏水性预测,结果显示,完整型和剪接型蛋白的N端都为疏水性,C端都为亲水性,完整型蛋白最小疏水值为-3.2,位于该蛋白的第734位氨基酸,最大疏水值为3.1,位于第685位氨基酸(图 5A);剪接型蛋白最小疏水值为-3.2,位于第434位氨基酸,最大疏水值为3.1,位于第384位氨基酸(图 5B)。说明ADAM10完整型和剪接型蛋白都具有一定的疏水性。
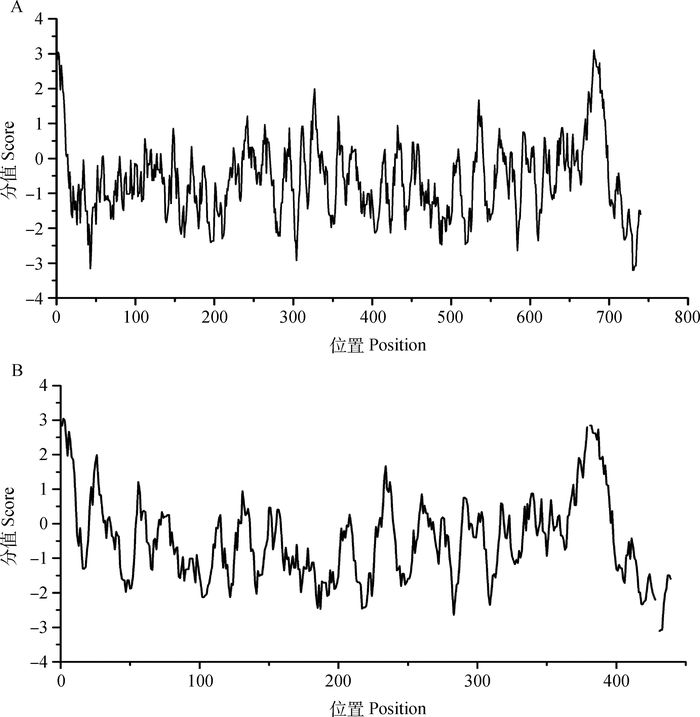
|
得分>0为疏水氨基酸;得分<0为亲水氨基酸。A.猪ADAM10完整型蛋白疏水性预测;B.猪ADAM10剪接型蛋白疏水性预测 Score > 0 indicate hydrophobic amino acids; Score < 0 indicate hydrophilic amino acids. A. The hydrophobicity of porcine ADAM10 intact protein; B. The hydrophobicity of porcine ADAM10 splicing protein 图 5 猪ADAM10完整型和剪接型蛋白疏水性分析 Figure 5 The hydrophobicity of porcine ADAM10 intact and splicing proteins |
通过在线软件SignalP-4.1分别对猪ADAM10完整型和剪接型蛋白信号肽进行预测,结果显示,2种蛋白都存在相同的信号肽序列(图 6),其中信号肽切割位点位于第18和第19位氨基酸残基之间。说明完整型和剪接型ADAM10蛋白都是分泌型蛋白。
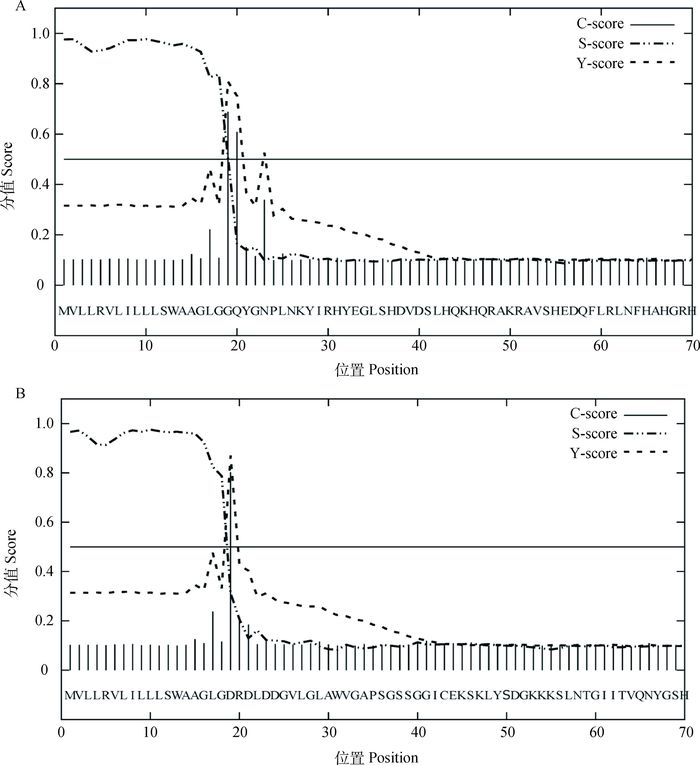
|
A.猪ADAM10完整型蛋白信号肽预测;B.猪ADAM10剪接型蛋白信号肽预测 A. The predicted result of signal peptide of porcine ADAM10 intact protein; B. The predicted result of signal peptide of porcine ADAM10 splicing protein 图 6 猪ADAM10蛋白信号肽预测 Figure 6 The predicted result of signal peptide of porcine ADAM10 |
利用TMHMM在线软件分别对猪ADAM10完整型和剪接型蛋白的跨膜结构进行预测,结果表明,ADAM10完整型和剪接型蛋白都存在跨膜结构,跨膜结构位于完整型第674~696位氨基酸残基,剪接型的第373~395位氨基酸残基,其中完整型蛋白的第1~673位氨基酸残基位于细胞膜外,第697~748位氨基酸处于细胞膜内;剪接型蛋白的第1~372位氨基酸残基位于细胞膜外,第396~447位氨基酸残基处于细胞膜内(图 7)。
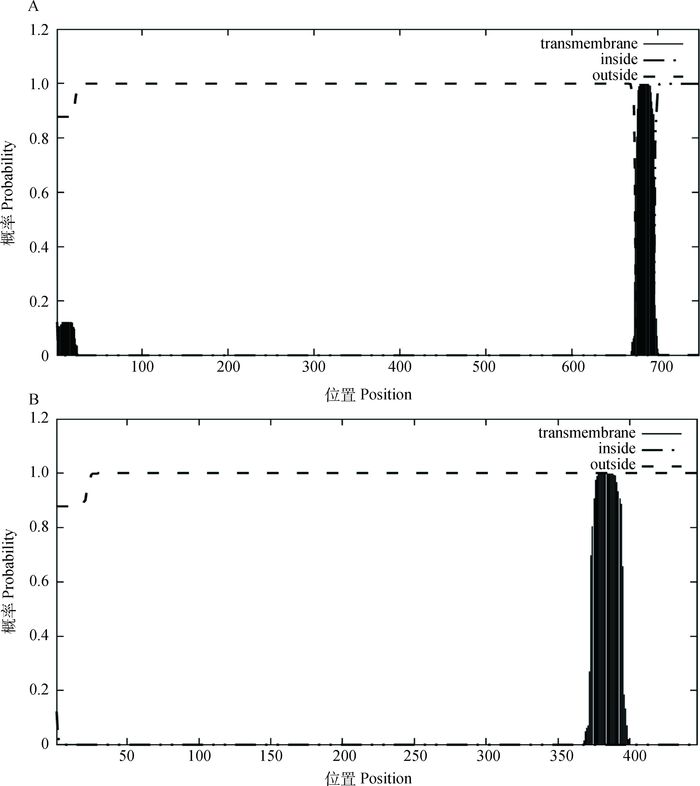
|
A.猪ADAM10完整型蛋白跨膜结构预测;B.猪ADAM10剪接型蛋白跨膜结构预测 A. The predicted result of transmembrane domain of porcine ADAM10 intact protein; B. The predicted result of transmembrane domain of porcine ADAM10 splicing protein 图 7 猪ADAM10蛋白跨膜结构预测 Figure 7 The predicted result of transmembrane domain of porcine ADAM10 |
利用NCBI的CDD在线软件分析蛋白保守结构域。结果显示,猪ADAM10完整型蛋白符合解整合素-金属蛋白酶家族结构特点,220~459位氨基酸残基存在锌依赖性金属蛋白酶结构域,46~146位氨基酸残基是一段先导肽段结构域,466~546位是解聚素结构域;而ADAM10剪接型蛋白缺失先导肽段结构域,但包含了部分锌依赖性金属蛋白酶结构域和完整的解聚素结构域(图 8)。
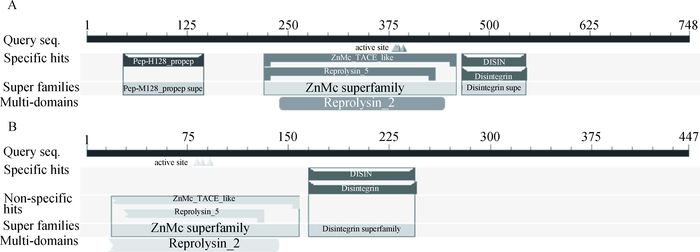
|
A.猪ADAM10完整型蛋白结构域;B.猪ADAM10剪接型蛋白结构域 A. Porcine ADAM10 intact protein domain; B. Porcine ADAM10 splicing protein domain 图 8 猪ADAM10蛋白保守结构域预测 Figure 8 The predicted result of conserved domains of porcine ADAM10 |
使用蛋白分析软件SOPMA对ADAM10蛋白的二级结构分析,结果显示,构成α-螺旋的氨基酸数量为183个,占总体的24.47%;构成β转角的氨基酸数量为66个,占总体的8.32%;构成无规则卷曲的氨基酸数目为357个,占总体的44.7%;构成延展链的氨基酸数目为142个,占总体的18.98%。通过Phyre2在线软件构建ADAM10完整型蛋白的高级结构,结果见图 9,分析发现,其与VAP1(Vascular apoptosis-inducing protein 1) 蛋白具有28%的相似性,具有100%的可信度。同时发现猪ADAM10剪接型蛋白的高级结构和完整型一致,推测完整型和剪接型具有相似的生物学功能。
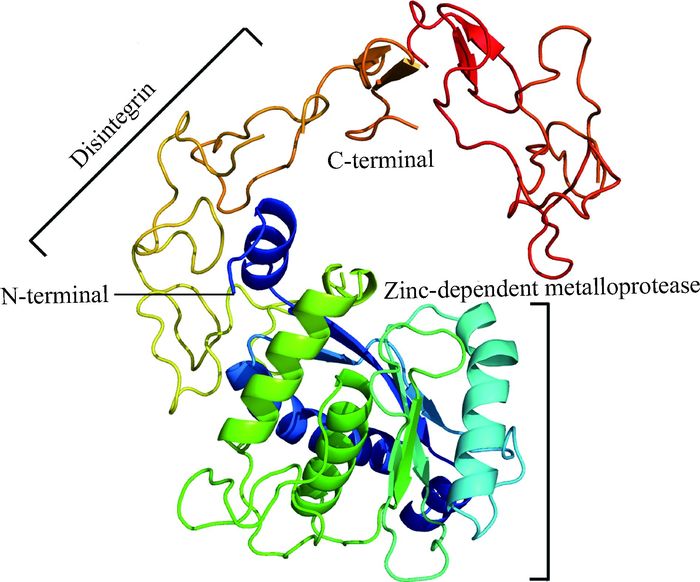
|
图 9 猪ADAM10蛋白三级结构预测 Figure 9 The prediction of tertiary structure of porcine ADAM10 |
根据猪ADAM10蛋白和其他物种的氨基酸序列,利用MEGA 5.0构建系统发育树,从进化树上可以看出,猪与山羊的ADAM10分子亲缘关系最近,其次是人、大鼠和小鼠,而与斑马鱼的分子亲缘关系较远(图 10)。
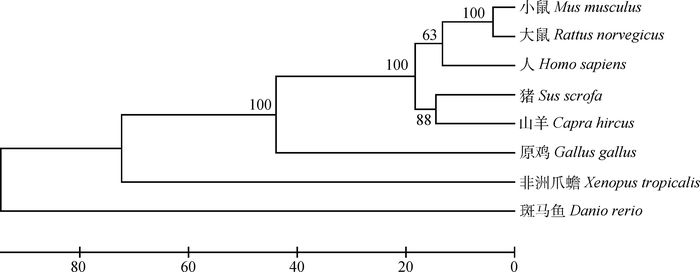
|
图 10 不同物种ADAM10蛋白的系统进化树 Figure 10 The phylogenetic tree of porcine ADAM10 protein among different species |
对猪12个组织提取RNA,利用特异性引物,采用半定量PCR方法检测猪ADAM10基因及其剪接体在不同组织中的表达情况。结果显示,猪ADAM10基因在不同组织中的表达和分布明显不同(图 11)。ADAM10基因完整型mRNA在脾中表达量较高,在肾、乳腺、腿肌、输卵管、卵巢、子宫、小肠等组织中低水平表达。ADAM10基因剪接体在肺中表达量较高,在脾和胃中也有低水平表达。
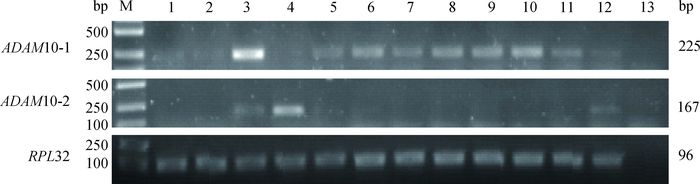
|
1.心;2.肝;3.脾;4.肺;5.肾;6.乳腺;7.腿肌;8.输卵管;9.卵巢;10.子宫;11.小肠;12.胃;13. H2O;M. DNA相对分子质量标准 1. Heart; 2. Liver; 3. Spleen; 4. Lung; 5. Kidney; 6. Mammary gland; 7. Leg muscle; 8. Fallopian tube; 9. Ovary; 10. Uterus; 11. Small intestine; 12. Stomach; 13. H2O; M. DNA marker 图 11 猪ADAM10基因及其剪接体的组织表达谱 Figure 11 The tissue expression profile of porcine ADAM10 gene and its splicing variant |
选择性剪接是真核生物调控基因表达的一种重要机制,生物体通过选择性剪接可以使得有限的基因表达出大量的基因终产物-蛋白质,其概念是指通过对前体mRNA选择不同的剪接位点组合进行剪接加工,产生成熟mRNA剪接异构体的过程,使得最终的蛋白质产物表现出结构和功能不同[14-16]。作为ADAM家族的一个重要成员ADAM10蛋白可以通过切割底物蛋白启动多种信号通路,其中底物蛋白包括IL-1、TNF-α和表皮生长因子等[13, 17-18]。关于人ADAM10蛋白的结构和功能已有较多的研究,但是当前对动物特别是猪的研究仍有许多不足。因此,本研究克隆了猪ADAM10基因编码区及其剪接体的序列。并利用分子生物信息学方法分别对其编码的蛋白结构进行分析。研究结果显示,猪ADAM10基因和其剪接体序列相差903 bp,其中完整型的ADAM10基因CDS长2 247 bp,编码748个氨基酸,剪接型ADAM10因为缺失2~7外显子全部序列和外显子8的5′端长129 bp的序列,其长度共1 344 bp,编码447个氨基酸。ADAM10剪接体并没有发生移码突变,相对于完整型氨基酸序列,其缺失19~319位氨基酸共301个氨基酸序列。研究发现,人ADAM10基因也存在选择性剪接,其剪接体长度(NP_001101.1) 和完整型(NP_001307499.1) 相比只是少了31个氨基酸序列,和完整型ADAM10相比较则只缺失了外显子7序列。而猪ADAM10基因如此大段的序列缺失,其生物学功能需要深入的研究。
通过对猪ADAM10基因同其他物种进行同源比对,发现此基因在不同物种中具有广泛的同源性,这也说明此基因作为一个具有重要功能的家族基因可能在不同物种中存具有广泛的结构和功能的一致性。蛋白保守功能域预测发现,此基因具有此家族蛋白的所有结构特点,其中包括几个重要的结构域,如前引导序列、金属蛋白酶域和解聚素结构域;而剪接体蛋白缺失前引导序列,但具有金属蛋白酶域和解聚素结构域2个重要的功能域。有研究发现,前引导序列作为一种分子伴侣能够帮助此蛋白特别是金属蛋白酶域进行正确折叠。在人肾上皮细胞系293细胞中过表达前引导序列缺失的ADAM10基因,其表达的蛋白并没有活性,而重新加入表达前引导序列的载体后,蛋白活性恢复正常[5, 19]。而一旦ADAM10蛋白折叠完成,需要在高尔基复合体中去除前引导序列后,金属蛋白酶域才能产生活性,切割蛋白底物[20-21]。猪AMAD10剪接体蛋白缺失整个前引导序列,因此推测其单独存在时并没有生物学功能,而和猪ADAM10完整型蛋白同时存在时,剪接体可能会借助完整型蛋白的前引导序列活性使得其正确折叠,从而使得其获得金属蛋白酶活性。信号肽和跨膜结构域预测结果发现,猪ADAM10完整型和其剪接体都存在完整的信号肽和一个跨膜结构域,说明剪接体蛋白同样可以在信号肽的引导下进入分泌途径,最终锚定在细胞膜中。总之,ADAM10剪接型蛋白在机体中的具体生物学功能尚不清楚,有待做进一步的分析和研究。
基因在机体内的组织表达差异和其行使的功能密切相关。为了研究猪ADAM10基因及其剪接体在猪各个组织中的表达情况,设计针对猪ADAM10基因完整型和剪接型的特异性引物,利用半定量PCR方法分析其在猪组织中的表达差异。结果显示,猪完整型ADAM10 mRNA在脾中表达量较高,而脾是机体最大的免疫器官,同时在脾中ADAM10基因可以切割TNFα前体生成成熟的TNFα[22],完整型mRNA在乳腺、输卵管、卵巢、子宫等繁殖器官中也有低水平表达,推测ADAM10基因与免疫应答和繁殖性能相关。ADAM10基因剪接体在肺中高表达,在脾和胃也有低水平表达。关于ADAM10基因剪接体和肺的关系以及其具体的功能还不是很清楚,需要进一步深入研究。
4 结论本研究克隆了猪ADAM10基因的编码区序列,发现一个剪接体。对猪ADAM10基因完整型及其剪接体蛋白进行了生物信息学分析。通过半定量PCR分析发现,ADAM10完整型在脾中特异高表达,而剪接体在肺中特异高表达。本研究为进一步深入研究猪ADAM10基因的分子特征和生物学功能奠定了理论基础。
| [1] | DREYMUELLER D, PRUESSMEYER J, GROTH E, et al. The role of ADAM-mediated shedding in vascular biology[J]. Eur J Cell Biol, 2012, 91(6-7): 472–485. DOI: 10.1016/j.ejcb.2011.09.003 |
| [2] | KWON J, JEONG S M, CHOI I, et al. ADAM10 is involved in cell junction assembly in early porcine embryo development[J]. PLoS One, 2016, 11(4): e0152921. DOI: 10.1371/journal.pone.0152921 |
| [3] | CHANTRY A, GREGSON N A, GLYNN P. e0152921[J]. J Biol Chem, 1989, 264(36): 21603–21607. |
| [4] | FOLKESSON M, LI C, FREBELIUS S, et al. Proteolytically active ADAM10 and ADAM17 carried on membrane microvesicles in human abdominal aortic aneurysms[J]. Thromb Haemost, 2015, 114(6): 1165–1174. DOI: 10.1160/TH14-10-0899 |
| [5] | SEALS D F, COURTNEIDGE S A. The ADAMs family of metalloproteases:multidomain proteins with multiple functions[J]. Genes Dev, 2003, 17(1): 7–30. DOI: 10.1101/gad.1039703 |
| [6] | ZHUANG J L, WEI Q L, LIN Z H, et al. Effects of ADAM10 deletion on Notch-1 signaling pathway and neuronal maintenance in adult mouse brain[J]. Gene, 2015, 555(2): 150–158. DOI: 10.1016/j.gene.2014.10.056 |
| [7] | FENG L Z, WANG Y J, CAI H, et al. ADAM10-Notch signaling governs the recruitment of ovarian pregranulosa cells and controls folliculogenesis in mice[J]. J Cell Sci, 2016, 129(11): 2202–2212. DOI: 10.1242/jcs.184267 |
| [8] | MURAI T, MIYAUCHI T, YANAGIDA T, et al. Epidermal growth factor-regulated activation of Rac GTPase enhances CD44 cleavage by metalloproteinase disintegrin ADAM10[J]. Biochem J, 2006, 395(1): 65–71. DOI: 10.1042/BJ20050582 |
| [9] | PAN Y, HAN C, WANG C L, et al. ADAM10 promotes pituitary adenoma cell migration by regulating cleavage of CD44 and L1[J]. J Mol Endocrinol, 2012, 49(1): 21–33. DOI: 10.1530/JME-11-0174 |
| [10] | ANDEREGG U, EICHENBERG T, PARTHAUNE T, et al. ADAM10 is the constitutive functional sheddase of CD44 in human melanoma cells[J]. J Invest Dermatol, 2009, 129(6): 1471–1482. DOI: 10.1038/jid.2008.323 |
| [11] | YANG K, LU L, LIU Y, et al. Increase of ADAM10 level in coronary artery in-stent restenosis segments in diabetic minipigs:high ADAM10 expression promoting growth and migration in human vascular smooth muscle cells via Notch 1 and 3[J]. PLoS One, 2013, 8(12): e83853. DOI: 10.1371/journal.pone.0083853 |
| [12] | KANDA K, KOMEKADO H, SAWABU T, et al. Nardilysin and ADAM proteases promote gastric cancer cell growth by activating intrinsic cytokine signalling via enhanced ectodomain shedding of TNF-α[J]. EMBO Mol Med, 2012, 4(5): 396–411. DOI: 10.1002/emmm.201200216 |
| [13] | LOREY M B, ROSSI K, EKLUND K K, et al. Global characterization of protein secretion from human macrophages following non-canonical caspase-4/5 inflammasome activation[J]. Mol Cell Proteomics, 2017, 16(4 Suppl 1): S187–S199. DOI: 10.1074/mcp.M116.064840 |
| [14] | BLACK D L. Mechanisms of alternative pre-messenger RNA splicing[J]. Annu Rev Biochem, 2003, 72: 291–336. DOI: 10.1146/annurev.biochem.72.121801.161720 |
| [15] |
万敬员, 张力, 叶笃筠. 选择性剪接调控机制的研究进展[J]. 国外医学分子生物学分册, 2003, 25(6): 342–345.
WAN J Y, ZHANG L, YE D Y. Research advances on mechanisms of alternative splicing regulation[J]. Journal of Medical Molecular Biology, 2003, 25(6): 342–345. (in Chinese) |
| [16] | VUONG C K, BLACK D L, ZHENG S K. The neurogenetics of alternative splicing[J]. Nat Rev Neurosci, 2016, 17(5): 265–281. DOI: 10.1038/nrn.2016.27 |
| [17] | ARDUISE C, ABACHE T, LI L, et al. Tetraspanins regulate ADAM10-mediated cleavage of TNF-α and epidermal growth factor[J]. J Immunol, 2008, 181(10): 7002–7013. DOI: 10.4049/jimmunol.181.10.7002 |
| [18] | VINCENT B. Regulation of the α-secretase ADAM10 at transcriptional, translational and post-translational levels[J]. Brain Res Bull, 2016, 126(Pt 2): 154–169. |
| [19] | ANDERS A, GILBERT S, GARTEN W, et al. Regulation of the α-secretase ADAM10 by its prodomain and proprotein convertases[J]. FASEB J, 2001, 15(10): 1837–1839. |
| [20] | MOCHIZUKI S, OKADA Y. ADAMs in cancer cell proliferation and progression[J]. Cancer Sci, 2007, 98(5): 621–628. DOI: 10.1111/cas.2007.98.issue-5 |
| [21] |
潘源, 丁学华. 抗癌治疗新靶标——去整合素金属蛋白酶10[J]. 国际肿瘤学杂志, 2011, 38(7): 492–495.
PAN Y, DING X H. A new anti-cancer target:ADAM10[J]. Journal of International Oncology, 2011, 38(7): 492–495. (in Chinese) |
| [22] | LUNN C A, FAN X D, DALIE B, et al. Purification of ADAM 10 from bovine spleen as a TNFα convertase[J]. FEBS Lett, 1997, 400(3): 333–335. DOI: 10.1016/S0014-5793(96)01410-X |



