副猪嗜血杆菌(Haemophilus parasuis)是一种革兰阴性短杆菌,常常存在于猪上呼吸道,能侵入机体并引起以纤维素性多发性浆膜炎、关节炎和脑膜炎为主要特征的格拉泽病[1-2]。该病普遍存在于世界各地,对养猪业造成巨大的经济损失,严重威胁着全世界养猪业的健康发展。
H. parasuis的毒力因子和致病机制一直是动物细菌疾病方面的研究热点,其毒力因子非常复杂。过去10年里,国内外研究者对H. parasuis的致病机制开展了广泛深入研究,发现了部分与H. parasuis致病性有关的毒力因子,但有关H. parasuis的毒力因子及其功能至今尚未完全解析,给该病理论研究及临床预防和控制造成了困难。
群体感应(quorum sensing,QS)系统是细菌之间信息交流的重要机制,当细胞数量到达一定密度时,在自诱导分子(autoinducer,AI)的作用下,相关基因通过该系统调控表达,从而导致细菌产生相关的代谢反应,如生物膜形成、毒力因子产生、耐药性产生等[3]。双组分调控系统(two component regulatory system,TCS)是广泛存在于细菌中介导细菌信息交流和基因调控的系统。QseBC双组分调节系统广泛存在于革兰阴性杆菌中,是细菌群体感应系统重要组成部分,组氨酸蛋白激酶(histidine protein kinase,HK)QseC通过感应外界的自诱导分子,经过一系列反应使调节蛋白QseB得到激活,激活的QseB可以调节细菌相关基因的表达,进而调控细菌的毒力、鞭毛和生物膜的形成[4-5]。而关于qseC基因在H. parasuis中作用如何,目前国内外尚未有相关的研究报道。
笔者构建了H. parasuis的qseC基因缺失株,并研究该qseC突变体的生物学特性,以期为进一步研究该基因在H. parasuis中的作用提供理论基础。
1 材料与方法 1.1 菌株及培养条件菌株:H. parasuis SC096为华南农业大学兽医学院禽病实验室保存的野生菌株;所构建的ΔqseC为H. parasuis qseC基因缺失突变体。
培养条件:含5%牛血清和0.02% NAD的TSA(胰蛋白胨大豆琼脂,Difco)和TSB(胰蛋白胨大豆肉汤,Difco)培养基。
1.2 试剂新生牛血清(Gibco),TSA(胰蛋白胨大豆琼脂,Difco),TSB(胰蛋白胨大豆肉汤,Difco);酵母提取物(Yeast extract,Oxoid),辅酶Ⅰ(NAD,购自上海生工生物工程有限公司)。新生牛血清购自北京鼎国生物技术有限公司;DNA相对分子质量标准、pMD19-T载体购自宝生物工程(大连)有限公司;氨苄青霉素(AMP)、卡那霉素(Kan)和庆大霉素(Gm)购自上海生工生物工程有限公司,其他化学试剂均为分析纯试剂。
1.3 引物分析H. parasuis的qseC基因序列,设计并合成相应引物(表 1)。
|
|
表 1 本研究所用的引物 Table 1 Primers used in this study |
首先用P1/P2和P3/P4引物扩增qseC上下游序列,用P237和P238引物以载体p34s-Gm作为模板扩增庆大霉素抗性基因Gmr片段,以上述三个片段作为模板,用引物P1和P4进行重叠PCR扩增,所获得PCR产物纯化后克隆到pMD19-T载体,经过PCR及测序确认序列正确后提取质粒,得到敲除载体pSF017。自然转化方法:将受体菌接种于TSA平板,在含5% CO2培养箱37 ℃培养24 h,然后用TSB把菌体从平板中冲洗下来,用TSB培养基把菌体浓度调整为109 cfu·mL-1。取20 μL菌液置于TSA平板上,加入10 μL上述qseC敲除载体,用接种环混合后置于培养箱中37 ℃培养5 h,然后用TSB再次把菌体洗下,离心,去上清后加入100 μL TSB培养基悬浮菌体,然后把菌液涂布在含有庆大霉素的TSA平板上,37 ℃培养2 d,同时用TE buffer(Tris-EDTA缓冲液)做阴性对照。抗性平板筛选,获得带有庆大霉素抗性的阳性菌落,用庆大霉素抗性基因Gmr检测引物对突变体进行PCR及测序鉴定。
1.5 SC096与ΔqseC生长曲线测定取H. parasuis SC096与ΔqseC的单个菌落于TSB液体培养基中,37 ℃恒温振荡培养至二者的OD600 nm值均为0.5,各取0.5 mL加入新鲜的TSB液体培养基中,37 ℃恒温振荡培养,每间隔1 h测取OD600 nm值,重复试验3次,取平均值,并绘制生长曲线。
1.6 生物被膜检测 1.6.1 玻璃试管生物被膜检测试验玻璃试管生物被膜检测参照J. B. Kaplan等的方法[6]。准备硅硼玻璃管在培养基中加入过夜培养物,在37 ℃静置培养24 h后,弃掉培养液,自然风干后加入甲醇固定15 min,吸出甲醇晾干后加入结晶紫溶液染色,室温放置5 min,用流水冲洗,除去结晶紫溶液,待完全干燥后,观察结果。每株菌株设三个重复,同时设培养基和空白对照。
1.6.2 微孔板生物被膜检测试验通过微孔板定量检测H. parasuis生物被膜。平底的96孔微孔板中每孔中加入100 μL新鲜培养液,培养液中接种10 μL菌液(OD600 nm值为1),盖上微孔板盖后37 ℃孵育36 h;然后,用注射器将菌液吸去,再加入200 μL无菌的磷酸盐缓冲液(PBS)清洗3次除去浮游菌体,100 μL甲醇固定15 min;风干后,加入100 μL 1%结晶紫染液染色10 min;最后,进行清洗、干燥;待完全干燥后,每孔加入100 μL的33%乙酸溶液溶解结晶紫,通过酶标仪测量OD630 nm值。副猪嗜血杆菌SC096和ΔqseC均做8个复孔,每次试验重复3次,结果以平均值表示。未接种菌液孔的OD值作为阴性对照,对比菌株的光密度情况。各组数据采用SPSS软件进行统计学分析,计量资料用“x±s”表示,采用t检验,P < 0.05为差异有统计学意义。
1.6.3 扫描电镜观察H. parasuis菌株在Millipore滤膜培养36 h,将滤膜轻轻浸入2.5%戊二醛中固定30 min,然后用0.1 mol·L-1 PBS清洗3~4次(每次15 min),经1% OsO4固定,再用0.1 mol·L-1 PBS清洗3~4次(每次15 min)。常规酒精逐级脱水,干燥后扫描电镜观察。
2 结果 2.1 缺失株鉴定对阳性克隆进行PCR鉴定,结果如图 1所示,用Gmr检测引物对突变体进行鉴定,只有qseC突变株能扩增出约750 bp的Gmr基因条带,而野生菌株SC096扩增不出任何条带,证明qseC突变体带有Gmr基因,同时,用qseC敲除盒的上游片段的正向引物和Gmr基因的下游引物对突变体进行确认,qseC敲除盒的上游片段大小约500 bp,Gmr抗性基因的片段大小约700 bp,理论上突变体应该能扩增出1 200 bp左右的特征条带,从图中可以看到只有qseC突变株能扩增出1 200 bp左右的敲除盒条带,而野生菌株不能扩增出相应的特征条带,说明qseC基因被抗性基因插入失活,对PCR产物进行测序,进一步确认qseC突变体构建成功。
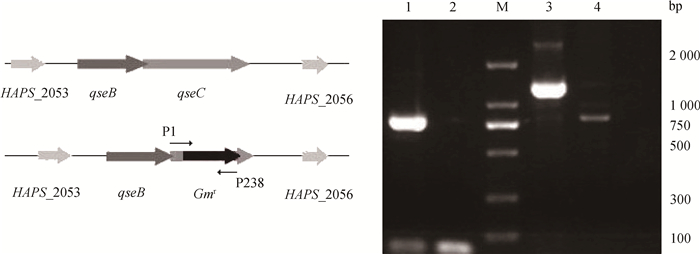
|
1. qseC突变株庆大霉素抗性基因Gmr扩增;2.野生菌株SC096庆大霉素抗性基因Gmr扩增;M. DNA相对分子质量标准;3.用引物P1与P238从qseC突变株扩增突变株特征片段;4.用引物P1与P238对野生菌株SC096进行PCR扩增 M. DL2000 DNA marker; Primers P237 and P238 were used to amplify Gmr gene from qseC mutant (Lane 1) and wild-type strain (Lane 2), and primers P1 and P238 to amplify signature sequence from qseC mutant (Lane 3) and wild-type strain (Lane 4) 图 1 H. parasuis qseC突变株基因缺失示意图(左)及PCR鉴定(右) Figure 1 The sketch map of qseC gene deletion (left) and identification of qseC mutant H. parasuis by PCR (right) |
以TSB培养基中各时刻所测定的细菌密度作图,从图 2可以看出,SC096和ΔqseC的生长速度没有明显的变化。说明在TSB这样丰富的培养基中,亲本株和qseC突变体在生长方面没有明显差异。
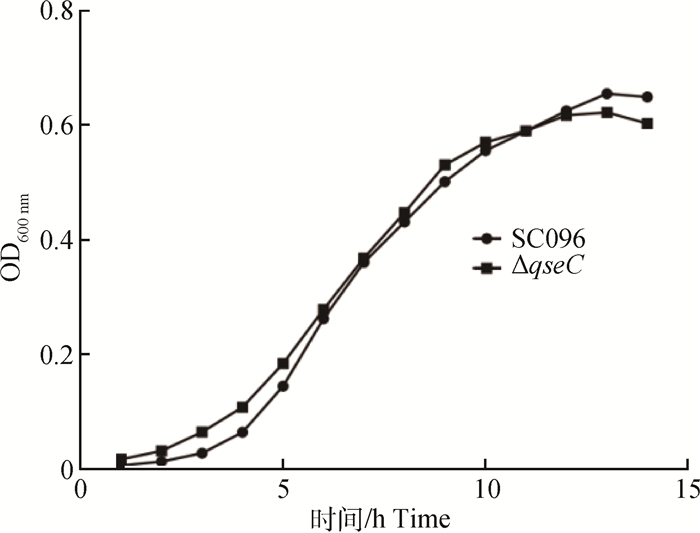
|
图 2 H. parasuis SC096和ΔqseC在TSB培养基中的生长曲线 Figure 2 Growth curves of H. parasuis SC096 and ΔqseC in TSB medium |
硅硼玻璃管检测H. parasuis SC096和ΔqseC形成的生物被膜,从图 3可以看出SC096和ΔqseC都具有生物被膜形成能力,qseC突变体的生物被膜比亲本株的生物被膜弱。

|
图 3 H. parasuis SC096和ΔqseC在玻璃试管中形成的生物被膜 Figure 3 The biofilm of H. parasuiss SC096 and ΔqseC in the glass tube |
结晶紫染色法检测H. parasuis SC096和ΔqseC的生物被膜,结果表明,SC096和ΔqseC的生物被膜定量OD630 nm值分别为0.26± 0.02和0.13±0.01,突变体ΔqseC形成的生物被膜的量明显少于亲本株SC096(P < 0.05,图 4)。
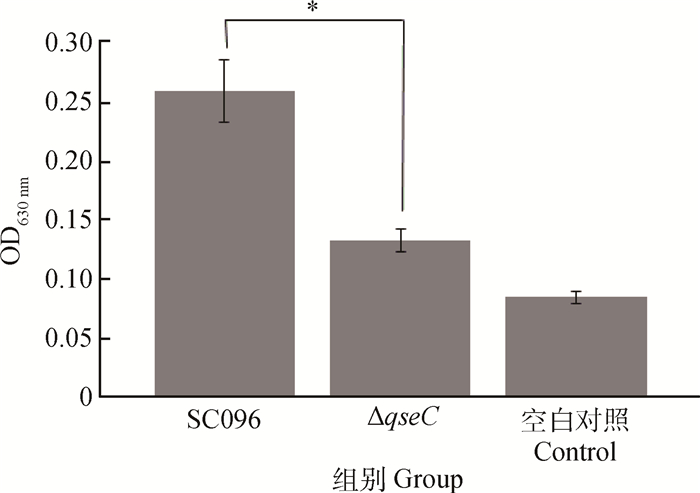
|
*. P < 0.05 图 4 H. parasuis SC096和ΔqseC在聚苯乙烯板中形成的生物被膜 Figure 4 The biofilm of H. parasuis SC096 and ΔqseC in the microplate |
H. parasuis在滤膜表面生长后通过扫描电镜观察,菌体呈短杆状,长短不一,浓厚致密的液样物质紧密包绕菌体周围,其间存在物质交换的孔道结构。图 5中A与B分别为SC096和ΔqseC的扫描电镜结果,图中显示亲本株SC096在滤膜表面能形成更浓密的生物被膜结构;而突变株ΔqseC表面明显缺乏液样物质包绕,结构较稀松,形成的生物被膜较弱。

|
A. SC096;B. ΔqseC 图 5 H. parasuis SC096和ΔqseC扫描电镜观察 Figure 5 Observation of H. parasuis SC096 and ΔqseC by scanning electron microscope |
QseBC双组分调控系统通过受体蛋白感应环境中的信号分子调控毒力相关基因的表达,进而实现细菌之间、细菌与宿主之间的通信[7-8]。许多研究发现,该双组分系统中的qseC基因对菌毛和鞭毛相关基因的表达有影响,从而影响细菌的运动力、生长能力、细胞侵袭力等。例如:鼠伤寒沙门菌缺失qseC基因导致其体外运动能力、在猪肠道中的定植能力及对小鼠的感染力均减弱[9-11]。此外,土拉杆菌(Francisella tularensis)缺失qseC基因会导致其生长能力降低[12],而胸膜肺炎放线杆菌(Actinobacillus pleuropneumoniae)缺失qseBC基因后,突变体的生长能力与野生株相比无明显差异[13],说明双组分系统QseBC对细菌生长能力的影响因细菌种属而异。本研究中,H. parasuis亲本株SC096和突变株ΔqseC生长曲线基本一致,说明缺失qseC基因后,H. parasuis生长能力不受影响。
细菌生物被膜是由于细菌中某些基因的激活,促进细菌分泌形成多糖蛋白复合物,该结构对抗生素和宿主免疫系统具有很强的抗性,从而导致严重的临床问题,常常造成慢性感染和反复发作[14-15]。细菌生物被膜的形成是一个复杂过程,受到多种信号分子的调控,其中QS系统中QseBC系统是主要的调控系统之一。研究发现qseC基因缺失严重影响了流感嗜血杆菌生物膜的形成[16]。伴放线放线杆菌(Actinobacillus actinomycetemcomitans)与侵袭性牙周炎密切相关[17]。生物被膜对伴放线放线杆菌的致病性有重要作用,由于QseBC双组分调控系统参与伴放线放线杆菌生物被膜形成的调控,与野生菌株相比,qseC基因缺失后的伴放线放线杆菌形成的生物被膜减少[18]。
QS系统依赖信号分子进行细胞间通信,随着信号分子浓度的增加,达到一定阈值,激活细菌体内某些基因的表达,产生生物被膜,所以QS对细菌生物被膜形成具有至关重要的作用[19-20]。现在认为细菌生物被膜的形成过程包括三个阶段:黏附、发展、成熟,细菌生物被膜的形成过程中,始终伴随着QS调控[21]。本研究中H. parasuis缺失qseC基因后,形成的生物被膜明显弱于野生菌株,其原因可能是H. parasuis缺失qseC基因后,影响H. parasuis生物被膜形成的三个阶段中细胞间通信的过程,进而影响H. parasuis的生物被膜形成能力。
综上所述,本研究观察到H. parasuis缺失qseC基因后,缺失株ΔqseC与野生株SC096的生长基本一致,同时发现缺失qseC基因后H. parasuis的生物被膜形成能力减弱,提示qseC基因对H. parasuis生物被膜的形成具有调控作用。通过鉴定H. parasuis生物被膜形成相关基因,可以以其作为靶标,制备相关生物制品阻止H. parasuis生物被膜形成,这对于治疗猪H. parasuis慢性感染具有重要的理论和实践意义。
4 结论通过比较H. parasuis野生菌株SC096株及其qseC基因缺失突变株生物被膜形成能力,发现qseC基因缺失后H. parasuis生物被膜形成能力减弱。二者生长速度基本一致。qseC基因对H. parasuis生物被膜的形成有重要的调控作用。
| [1] | AMANO H, SHIBATA M, KAJIO N, et al. Pathologic observations of pigs intranasally inoculated with serovar 1, 4 and 5 of Haemophilus parasuis using immunoperoxidase method[J]. J Vet Med Sci, 1994, 56(4): 639–644. DOI: 10.1292/jvms.56.639 |
| [2] | OLIVEIRA S, PIJOAN C. Computer-based analysis of Haemophilus parasuis protein fingerprints[J]. Can J Vet Res, 2004, 68(1): 71–75. |
| [3] |
张永良, 侯小平, 关宏坤, 等. 军事坑道内空气微生物污染调查及其消毒[J]. 军事医学科学院院刊, 2008, 32(1): 39–41.
ZHANG Y L, HOU X P, GUAN H K, et al. Investigation and disinfection of air microbial contamination in martial tunnels[J]. Bulletin of the Academy of Military Medical Sciences, 2008, 32(1): 39–41. (in Chinese) |
| [4] |
朱浴晖, 陶然, 莫浩联, 等. 科氏葡萄球菌引发院内感染事件调查分析[J]. 中国卫生工程学, 2001, 10(3): 106–108.
ZHU Y H, TAO R, MO H L, et al. The investigation of infected incident caused by Staphylococcus cohnii in hospital[J]. Chinese Journal of Public Health Engineering, 2001, 10(3): 106–108. (in Chinese) |
| [5] |
YAMASHITAS, YONEMURAK, SUGIMOTOR, 等. 科氏葡萄球菌可导致Weber-Christian病患者多发性脑脓肿[J]. 世界核心医学期刊文摘(神经病学分册), 2006(3): 19.
YAMASHITA S, YONEMURA K, SUGIMOTO R, et al. Staphylococcus cohnii as a cause of multiple brain abscesses in Weber-Christian disease[J]. Digest of the World Core Medical Journals:Clinical Neurology, 2006(3): 19. (in Chinese) |
| [6] | KAPLAN J B, MULKS M H. Biofilm formation is prevalent among field isolates of Actinobacillus pleuropneumoniae[J]. Vet Microbiol, 2005, 108(1-2): 89–94. DOI: 10.1016/j.vetmic.2005.02.011 |
| [7] | CLARKE M B, HUGHES D T, ZHU C R, et al. The qseC sensor kinase:a bacterial adrenergic receptor[J]. Proc Natl Acad Sci U S A, 2006, 103(27): 10420–10425. DOI: 10.1073/pnas.0604343103 |
| [8] | HUGHES D T, CLARKE M B, YAMAMOTO K, et al. The qseC adrenergic signaling cascade in enterohemorrhagic E. coli (EHEC)[J]. PLoS Pathog, 2009, 5(8): e1000553. DOI: 10.1371/journal.ppat.1000553 |
| [9] | BEARSON B L, BEARSON S M D, LEE I S, et al. The Salmonella enterica serovar Typhimurium QseB response regulator negatively regulates bacterial motility and swine colonization in the absence of the qseC sensor kinase[J]. Microb Pathog, 2010, 48(6): 214–219. DOI: 10.1016/j.micpath.2010.03.005 |
| [10] | BEARSON B L, BEARSON S M D. The role of the qseC quorum-sensing sensor kinase in colonization and norepinephrine-enhanced motility of Salmonella enterica serovar typhimurium[J]. Microb Pathog, 2008, 44(4): 271–278. DOI: 10.1016/j.micpath.2007.10.001 |
| [11] | MOREIRA C G, WEINSHENKER D, SPERANDIO V. qseC mediates Salmonella enterica serovar typhimurium virulence in vitro and in vivo[J]. Infect Immun, 2010, 78(8): 3647. DOI: 10.1128/IAI.00602-10 |
| [12] | DURHAM-COLLERAN M W, VERHOEVEN A B, VAN HOEK M L. Francisella novicida forms in vitro biofilms mediated by an orphan response regulator[J]. Microb Ecol, 2010, 59(3): 457–465. DOI: 10.1007/s00248-009-9586-9 |
| [13] | LIU J L, HU L L, XU Z F, et al. Actinobacillus pleuropneumoniae two-component system QseB/qseC regulates the transcription of PilM, an important determinant of bacterial adherence and virulence[J]. Vet Microbiol, 2015, 177(1-2): 184–192. DOI: 10.1016/j.vetmic.2015.02.033 |
| [14] | ISHAK N, TIKHOMIROVA A, BENT S J, et al. There is a specific response to pH by isolates of Haemophilus influenzae and this has a direct influence on biofilm formation[J]. BMC Microbiol, 2014, 14: 47. DOI: 10.1186/1471-2180-14-47 |
| [15] | BELLO-ORTÍ B, DESLANDES V, TREMBLAY Y D N, et al. Biofilm formation by virulent and non-virulent strains of Haemophilus parasuis[J]. Vet Res, 2014, 45: 104. DOI: 10.1186/s13567-014-0104-9 |
| [16] | ÜNALC M, SINGHB, FLEURYC, 等. qseC controls biofilm formation of non-typeable Haemophilus influenzae in addition to an AI-2-dependent mechanism[J]. Int J Med Microbiol, 2012, 302(6): 261–269. |
| [17] |
冯向辉, 徐莉, 孟焕新, 等. 慢性牙周炎患者抗伴放线放线杆菌IgG滴度改变的临床意义[J]. 北京大学学报:医学版, 2010, 42(2): 207–210.
FENG X H, XU L, MENG H X, et al. Clinical significance of the change in serum IgG titers respond to Aggregatibacter actinomycetemcomitans in chronic periodontitis patients[J]. Journal of Peking University:Health Sciences, 2010, 42(2): 207–210. (in Chinese) |
| [18] | NOVAK E A, SHAO H J, DAEP C A, et al. Autoinducer-2 and qseC control biofilm formation and in vivo virulence of Aggregatibacter actinomycetemcomitans[J]. Infect Immun, 2010, 78(7): 2919–2926. DOI: 10.1128/IAI.01376-09 |
| [19] | SHROUT J D, TOLKER-NIELSEN T, GIVSKOV M, et al. The contribution of cell-cell signaling and motility to bacterial biofilm formation[J]. MRS Bull, 2011, 36(5): 367–373. DOI: 10.1557/mrs.2011.67 |
| [20] | KUMAR J S, UMESHA S, PRASAD K S, et al. Detection of quorum sensing molecules and biofilm formation in Ralstonia solanacearum[J]. Curr Microbiol, 2016, 72(3): 297–305. |
| [21] | PARSEK M R, GREENBERG E P. Sociomicrobiology:the connections between quorum sensing and biofilms[J]. Trends Microbiol, 2005, 13(1): 27–33. DOI: 10.1016/j.tim.2004.11.007 |



