长链非编码RNAs (lncRNAs)是一类近年来新发现的长度大于200个核苷酸,并且无编码能力的RNA分子。根据lncRNAs在基因组上的位置,将其分为基因间区的lncRNAs、内含子中的lncRNAs和反义lncRNAs[1-2]。lncRNAs在转录水平、转录后水平通过信号分子、捕获分子、导向分子、支架分子等4种方式调控基因的表达,在生长发育、细胞分化、细胞周期调控、癌症发生中发挥作用,因此在恶性肿瘤的治疗和诊断中有着重要的应用前景[3]。近年的研究表明,lncRNAs的数量远远大于蛋白编码基因的数量[4],通过调节基因表达发挥作用。lncRNA和转录因子结合影响基因调控,除了蛋白质以外,lncRNA还可以和其他RNA相互作用, 包括mRNA、lncRNA和miRNA等;甚至lncRNA能够进入细胞核直接和DNA作用, 同时染色体和部分印记基因的转录能力也会受到lncRNA的顺式调控作用[5]。由此可见,lncRNA的作用机制是复杂和多样化的,因此是生物代谢调控网络的重要组成部分[6]。
J.L.Rinn等[7]首次在人成纤维细胞中发现了HOX转录反义RNA (HOX transcript antisense RNA, HOTAIR)。HOTAIR定位于人类染色体12q13.13区域HOX基因家族HOX11基因的反义链[8-9]。研究表明,HOTAIR的5′和3′端序列可以分别与PRC2和组蛋白去甲基化酶LSD1/CoREST/REST复合物结合作用于特定的基因位点,沉默目的基因[8-9]。HOTAIR的敲除和过表达研究的结果表明,HOTAIR对PRC2和LSD1的相互作用是必不可少的,并且进一步通过染色质共沉淀技术,证实了HOTAIR能作为PRC2和LSD1这2个复合体的支架,介导这2种复合体结合到特异性基因位点[10]。
近年来研究表明,HOTAIR在多种肿瘤中异常表达,针对其发挥的生物学功能及具体分子机制已有较深入的研究,其在肿瘤中的重要作用逐渐被发现。多个研究表明,HOTAIR与多种癌症(黑色素瘤[10]、肝癌[11]、结肠癌[12]、胰肠癌[13]、胃肠间质瘤[14]、喉癌[15]、乳腺癌[16]等)的转移、进展及预测有关。高表达的HOTAIR能够抑制癌基因的表达,促进肿瘤复发转移,而下调的HOTAIR表达则降低肿瘤细胞生物转移侵袭能力。本研究利用一些数据库和软件对长链非编码RNA HOTAIR进行分析,并预测其在黑色素瘤致病过程中的互作分子,结合HOTAIR靶基因的GO和KEGG分析,以及一些转录因子对HOTAIR的调控,为相关肿瘤的研究提供一定的理论基础。
1 材料与方法 1.1 相关数据库及软件UCSC Genome Browser (http://genome.UCSC.Edu/)为基因组浏览工具。
lncRNAtor (http://lncrnator.ewha.ac.kr/index.htm)[17]数据库相互作用的蛋白具有集成序列的综合处理,进化的分数和编码潜能,数据集是从TCGA、GEO、ENCODE和modENCODE收集而来(物种包括人、小鼠、果蝇、线虫和酵母)。
MEGA4.0[18]用于构建系统发育树,进行序列比对等。
LncRNA Disease database[19](http://202.38.126.151/hmdd/html/tools/lncrnadisease)是人类lncRNA相关的疾病数据库。
NONCODE (http://noncode.org/) [20]由中国科学院计算技术研究所构建,是lncRNA领域航母级别的数据库,也是目前为止数据最为全面的非编码RNA数据库,包括lncRNA在不同组织中的特异性表达。
Annolnc (http://annolnc.cbi.pku.edu.cn/)[21]数据库系统地解释了人类新型lncRNA的功能,包括转录因子的预测等。
starbaseV2.0(http://starbase.sysu.edu.cn/)[22]数据库收录的物种包括Homo sapiens、Mus musculus和C.elegans,该数据库是CLIP-Seq和mRNA降解组测序等高通量试验数据支持的miRNA靶标数据库,包括与lncRNA互作的miRNA。
Co-lncRNA (http://www.bio-bigdata.com/Co-LncRNA/)[23]数据库系统地分析了lncRNA的GO功能注释和KEGG通路的富集。
MiTranscriptome (http://www.mitranscriptome.org/)[24]数据库系统地展示了不同的lncRNA在不同疾病中的表达水平,以及与正常组织表达量的比较。数据来源于6 500多个样品的高通量RNA测序(RNA-Seq)。
TargetScan (http://www.targetscan.org/vert_71/)[25]可以预测与某个特定基因相互作用的miRNA。
1.2 分析方法运用UCSC基因组浏览工具查询HOTAIR在人类基因组中的定位及其在不同物种中的基因序列。运用MEGA4.0软件构建同源系统进化树。
在NONCODE数据库中分析总结HOTAIR在人各个组织的特异性表达。运用Annolnc在线软件分析HOTAIR的转录调控因子。运用starbaseV2.0预测与HOTAIR结合的miRNA家族。通过lncRNAtor在线数据库查找已经通过免疫共沉淀测序验证的与HOTAIR互作的蛋白质。
Co-lncRNA (GSE31614) 数据库中统计分析HOTAIR靶基因的GO功能注释和KEGG通路的富集。
运用LncRNA Disease database在线分析查找与HOTAIR相关的疾病。并在MiTranscriptome数据库检索HOTAIR在一些癌症中的表达量,以及一些癌症组织和正常组织表达量的对比。
通过TargetScan软件在线预测与黑色素瘤紧密相关的一些基因的靶标miRNA。
用Cytoscape3.2.0软件绘制miRNA-HOTAIR-黑色素瘤相关蛋白的分子调控网络图。
用IBM SPSS statistics 22进行统计学分析,本试验以P=0.05作为各项结果的差异显著性判断标准。
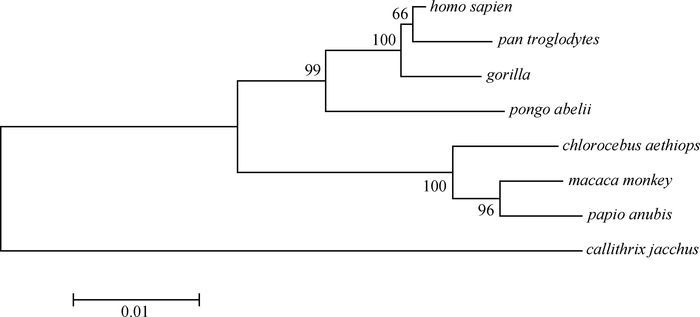
2 结果 2.1 HOTAIR在人类基因组的定位及进化分析运用UCSC基因组工具找到HOTAIR在人类基因组的定位。HOTAIR定位于人类12q13.13染色体上HOXC11与HOXC12基因的54 356 092~54 368 740 nt位点之间,包括5个短外显子和1个长外显子,其RNA全长约2 158 nt。由UCSC数据库下载人(Homo sapien)、黑猩猩(Pan troglodytes)、大猩猩(Gorilla gorilla)、苏门答腊猩猩(Pongo abelii)、绿猴(Chlorocebus aethiops)、猕猴(Macaca monkey)、狒狒(Papio anubis)和狨猴(Callithrix jacchus)8个物种的HOTAIR序列,运用MEGA4.0软件构建系统进化树(图 1),计算方法为领位相连法(Neighbor-joining)和自展内部分枝法(Bootstrapping)。
|
分枝上的数值为靴带可行度,左下角为标尺 Numbers above branches are bootstrap percentage values by replication, the bottom left corner is a scale 图 1 HOTAIR系统进化树 Figure 1 Phylogenetic tree of HOTAIR |
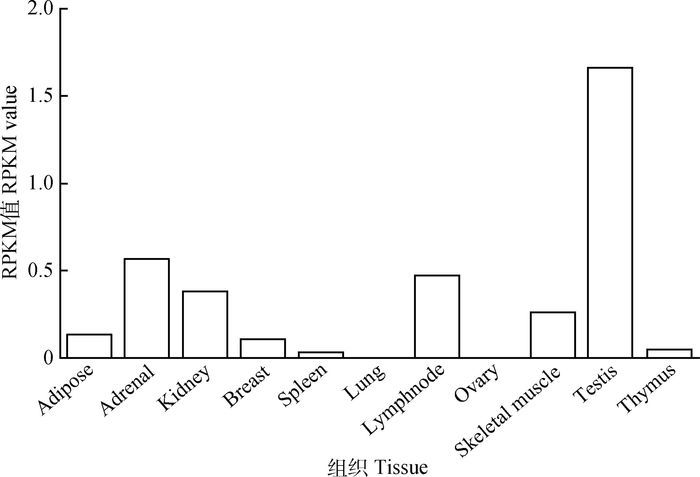
在NONCODE数据库中分析总结HOTAIR在人的11种器官和组织的特异性表达。包括脂肪组织(Adipose)、肾上腺(Adrenal)、肾(Kidney)、乳腺(Breast)、脾(Spleen)、肺(Lung)、淋巴结(Lymphnode)、卵巢(Ovary)、骨骼肌(Skeletal muscle)、睾丸(Testis)和胸腺(Thymus),其中在NONCODE数据库中统计到在肺和卵巢中的RPKM值分别为3.029E-08和2.282E-07,在睾丸中的表达量为1.659(图 2)。
|
Y轴为RPKM (Reads Per kilo bases per million reads)值, 代表每百万reads中来自于某基因每千碱基长度的reads数,图 4同。X轴为组织和器官 The Y-axis means RPKM (Reads per kilo bases per million reads) values, representing the number of reads per kilo bases per million reads from the length of a gene, the same as Figure 4. X-axis means the tissues and organisms 图 2 HOTAIR在组织和器官的特异性表达 Figure 2 The special expression of HOTAIR in different tissues and organisms |
运用Annolnc在线软件分析HOTAIR的转录调控因子,HOTAIR在H1、GM12/78等18种细胞中被27个转录调控因子调控(表 1)。
|
|
表 1 HOTAIR在不同细胞中的转录调控因子 Table 1 The transcription factors of HOTAIR in different cells |
运用starbaseV2.0生物信息学软件预测得到与HOTAIR结合的miRNA家族共28个,包括miR-217, miR-17/17-5p/20ab/20b-5p/93/106ab/427/518a-3p/519d, miR-375, miR-19ab, miR-30abcdef/30abe-5p/384-5p, miR-221/222/222ab/1928, miR-204/204b/211, miR-1ab/206/613, miR-9/9ab, miR-148ab-3p/152, miR-194, miR-129-5P/129ab-5p, miR-203, miR-216b/216b-5p, miR-124/124ab/506, miR-216a, miR-21/590-5p, miR-133abc, miR-103a/107/107ab, miR-214/761/3619-5p, miR-150/5127, miR-129-5p/129ab-5p, miR-33ab/33-5p, miR-130ac/301ab/301b/301b-3p/454/721/4295/3666, miR-208ab/208ab-3p, miR-499-5p, miR-34ac/34bc-5p/449abc/449c-5p, miR-193/193b/193a-3p, 其中miR-217和miR-17已经通过免疫共沉淀证明与HOTAIR具有结合位点。
2.5 HOTAIR与蛋白质的互作通过lncRNAtor在线数据库统计分析已经经免疫共沉淀测序验证的与HOTAIR互作的蛋白质共有15个,并且预测能够与HOTAIR互作,但未经试验证明的蛋白质有31个(评分≥90,P<0.05) (表 2)。
|
|
表 2 与HOTAIR互作的蛋白 Table 2 The proteins interacted with HOTAIR |
在Co-lncRNA (GSE31614) 数据库中统计分析发现,HOTAIR的靶基因在生物学过程(Biological process, BP)、细胞组成(Cellular component, CC)、分子功能(Molecular function, MF)3个层面的Gene Ontology注释层次分类及富集。HOTAIR的靶基因富集在负调控细胞分化、细胞周期、负调控生长、细胞发育、神经发育和细胞凋亡等多个生物学过程中,作用位置在细胞核内和细胞质中,发挥着调控作用。其生物学过程的DNA复制、转录调控与小分子调控作用会对癌症细胞的增殖、分化产生影响。通过Fsiher Exact Test计算P值,以P<0.05为显著性阈值得到的基因相对于背景具有统计学意义的GO条目,并对P值进行校正,标准是Benjamini<0.05(表 3、表 4、表 5)。
|
|
表 3 HOTAIR靶基因在分子功能层面富集 Table 3 Function enrichment of HOTAIR predicted target genes in molecular function |
|
|
表 4 HOTAIR靶基因在生物过程层面富集 Table 4 Function enrichment of HOTAIR predicted target genes in biological process |
|
|
表 5 HOTAIR靶基因在细胞组分层面富集 Table 5 Function enrichment of HOTAIR predicted target genes in celluar component |
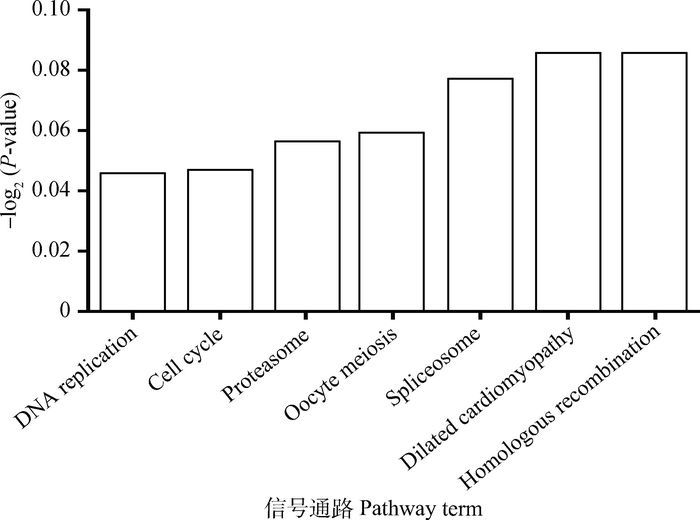
在Co-lncRNA数据库中统计分析KEGG的生物通路富集,HOTAIR的靶基因显著富集于DNA复制、细胞周期、同源重组等信号通路。通过Fsiher Exact Test计算P值,以P<0.05为显著性阈值得到的基因相对于背景具有统计学意义的GO条目,并对P值进行校正,标准是Benjamini<0.05(图 3)。
|
Y轴为-log2(P-value),X轴为富集到的信号通路条目(P<0.05, Benjamini<0.05) The Y-axis means -log2(P-value), X-axis means the pathway terms enriched (P < 0.05, Benjamini < 0.05) 图 3 HOTAIR靶基因的KEGG通路分析 Figure 3 KEGG enrichment of HOTAIR predicted target genes |
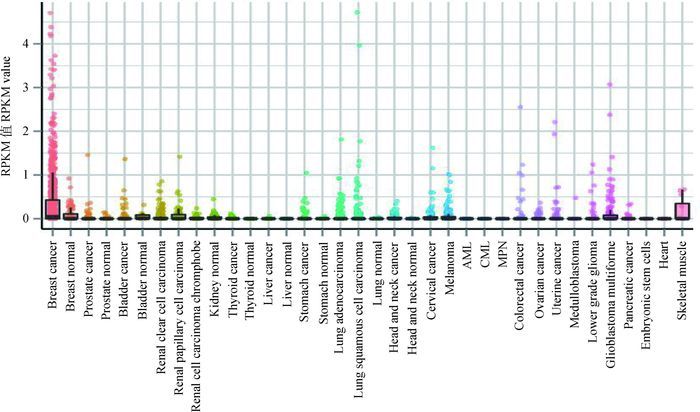
在Lnc RNA Disease数据库中检索显示,HOTAIR与乳腺癌、食管癌、肺癌、胃癌、结肠癌、黑色素瘤等多种肿瘤密切相关,并且在MiTranscriptome数据库里面检索到HOTAIR在一些癌症的表达量以及一些癌症组织和正常组织的表达量的对比,在乳腺癌中的表达量明显高于在其他癌症中的表达量(P<0.01),HOTAIR在黑色瘤中的表达量高于在正常组织中的表达量(图 4)。
|
X轴为癌症和正常组织 X-axis means the cancers and the normal tissues 图 4 HOTAIR在组织中的表达 Figure 4 The expression levels of HOTAIR in tissues |
通过Annolnc在线软件分析得到与HOTAIR互作的miRNA家族有miR-217、miR-203、miR-375和miR-17/17-5p/20ab/20b-5p/93/106ab/427/518a-3p/519d等, 通过TargetScan软件在线预测一些与黑色素瘤紧密相关基因的靶标miRNA,如与黑色素瘤中黑色素形成紧密相关的小眼畸形转录因子MITF,发现miR-217、miR-375等与MITF也有互作。同样miR-203、miR-194与黑色素瘤的迁移、侵染相关的NKX3-1、CXCR4和JARID2基因也存在着一定的调控关系。
2.8.2.1预测miRNA-HOTAIR-MITF的分子调控网络对黑色素瘤的影响: MITF是黑色素瘤中控制黑色素形成的关键基因,通过Cytoscape3.2.0软件分析并绘制miRNA-HOTAIR-MITF的分子调控网络图,从图 5可以看出,与HOTAIR互作的miRNA中miR-194、miR-203、miR-375等也调控MITF的表达,由此推测,HOTAIR会与MITF竞争性的结合miRNA,从而实现对MITF的调控。
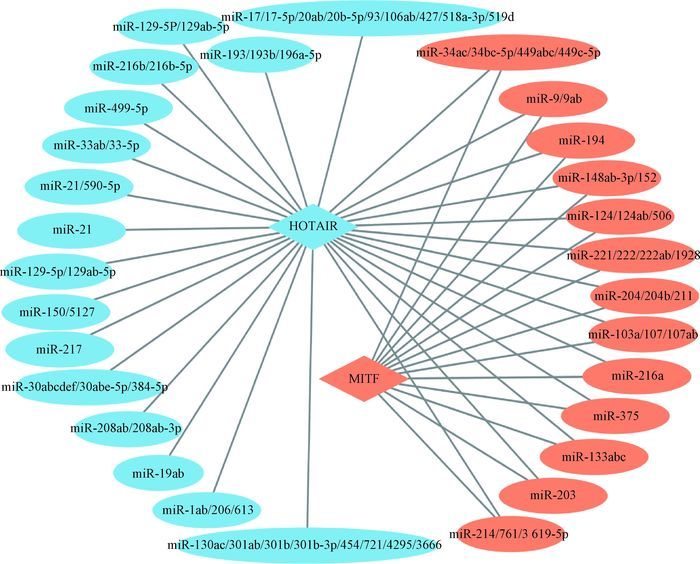
|
椭圆代表与HOTAIR互作的miRNAs,蓝色的菱形代表HOTAIR,橙色的菱形代表MITF,橙色的椭圆代表与MITF互作的miRNAs The ellipse represent the miRNAs interacted with HOTAIR, the blue diamond represents HOTAIR, the orange diamond represents MITF, and the orange ellipse represent the miRNAs interacted with MITF 图 5 miRNA-HOTAIR-MITF的分子调控网络 Figure 5 The molecular regulation network of miRNA-HOTAIR-MITF |
预测miRNA-HOTAIR-与黑色素迁移相关基因的互作网络:与黑色素瘤迁移、侵染相关的NKX3-1、CXCR4和JARID2基因,对于黑色素瘤的治疗有着至关重要的作用。用Cytoscape3.2.0软件绘制miRNA-HOTAIR-NKX3-1、miRNA-HOTAIR-CXCR4和miRNA-HOTAIR-JARID2的分子调控网络图,从图 6可以看出,一些miRNA会同时调控HOTAIR和NKX3-1、CXCR4和JARID2,由此推测,HOTAIR可与NKX3-1、CXCR4和JARID2竞争性的结合这些miRNA,从而实现对黑色素瘤迁移、侵染的调控。
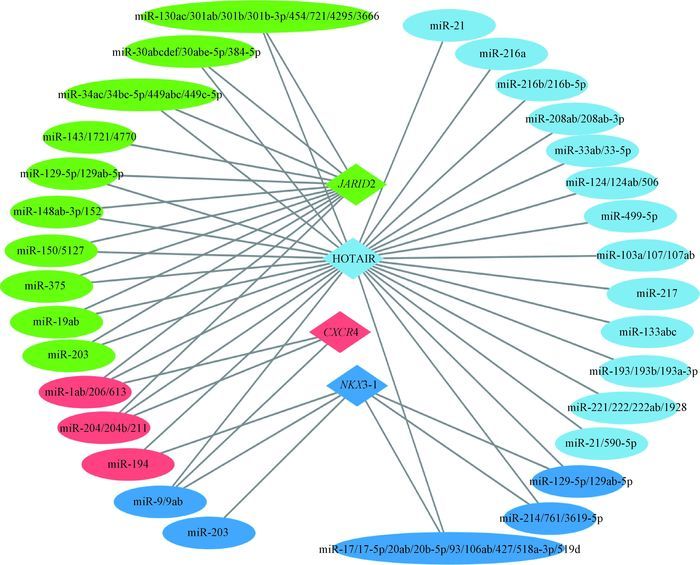
|
椭圆代表与HOTAIR互作的miRNAs,浅蓝色的菱形代表HOTAIR,红色、蓝色、绿色的菱形代表分别代表CXCR4、NKX3-1、JARID2,相应颜色的椭圆代表与其互作的miRNAs The ellipse represent the miRNAs interacted with HOTAIR, the light blue diamond represents HOTAIR, the red, blue and green diamonds represent the CXCR4, NKX3-1, JARID2, respectively, and the corresponding ellipse represent the miRNAs interacted with CXCR4, NKX3-1, JARID2 图 6 miRNA-HOTAIR-NKX3-1, miRNA-HOTAIR-CXCR4, miRNA-HOTAIR-JARID2的分子调控网络 Figure 6 The molecular regulation network of miRNA-HOTAIR-NKX3-1, miRNA-HOTAIR-CXCR4, miRNA-HOTAIR-JARID2 |
全基因组及转录组测序研究显示,根据转录本核苷酸长度,ncRNA通常被分为短链ncRNA (Small non-coding RNA, sncRNA, 18~200 nt)和长链ncRNA (Long ncRNA, lncRNA, >200 nt)[26]。最初的研究认为,lncRNA是生物体内的垃圾,不具有生物学活性,然而近些年来lncRNA成为众多研究领域的焦点,其能在转录水平、转录后水平及等多层面调控基因表达,且与诸多肿瘤及非肿瘤性疾病密切相关[27]。
本研究应用UCSC基因组数据库分析HOTAIR在人类基因组中的定位及保守性,HOTAIR位于人类、黑猩猩的第12号染色体,位于牛的第5号染色体[28]。NONCODE统计分析发现,HOTAIR在睾丸、淋巴、肾上腺以及肺等中都有表达。研究表明,HOTAIR在非小细胞肺癌组织及细胞系内均高表达,且其上调水平与进展病理分期、淋巴结转移及不良预后均密切相关[29]。HOTAIR在转移性淋巴结组织中的表达量较原发黑色素瘤组织中明显上调, 研究表明,敲除HOTAIR会抑制黑色素瘤细胞的运动性和侵蚀性,对于黑色素瘤的治疗具有非常重要的意义[30]。
大量的研究表明,HOTAIR参与调控多种肿瘤,在lncDisease数据库中发现,HOTAIR参与调控乳腺癌、食管癌、肺癌、胃癌、结肠癌、黑色素瘤等多种癌症,由MiTranscriptome数据库分析HOTAIR在乳腺癌、前列腺癌、膀胱癌和甲状腺癌的正常组织和癌症组织的表达量的对比,发现在癌症组织或细胞中HOTAIR的表达均为上调。HOTAIR的表达上调与肿瘤的发生、发展、转移和预后密切相关,干扰HOTAIR的表达后能显著抑制肿瘤细胞的增殖、侵袭和迁移能力,为肿瘤个体化治疗及解决化疗耐药性提供依据和靶点[31-32]。
HOTAIR靶基因的GO注释富集在负调控细胞周期等多个生物学过程条目中,已经有试验证明,HOTAIR会抑制黑色素瘤细胞的运动性和侵蚀性,因此,HOTAIR会对黑色素瘤产生负调控作用,从而抑制黑色素瘤细胞的迁移和侵袭。Lin28B是HOTAIR的重要靶基因,同时又是miR-126a-5p在黑素瘤中的直接靶标。Lin28B在大部分黑色素瘤患者中异常表达,并且在黑色素瘤进展过程中起着非常重要的作用。Lin28B的参与将会导致在黑色素瘤中MAPK激酶的激活[33]。MAPK信号通路参与了黑色素细胞的分化,以及黑色素的生成,并且能够促进黑色素瘤细胞的迁移和侵袭。
恶性黑色素瘤是一种恶性程度极高的皮肤肿瘤,是皮肤病中死亡率最高的疾病。侵袭和转移是治疗黑色素瘤最大的障碍[34]。关于HOTAIR对恶性黑色素瘤的侵袭及转移的作用机制尚不十分清楚。我们通过miRNA-HOTAIR-蛋白的网络互作图预测HOTAIR对黑色素瘤的调控机制,小眼畸形转录因子(MITF),在黑色素瘤中,控制黑色素非常关键的一个转录因子。通过分析发现,与HOTAIR互作的miRNAs中miR-217、miR-203、miR-375也调控MITF的表达,由此推测,HOTAIR会与MITF竞争性的结合这些miRNAs,从而实现对MITF的调控。与黑色素瘤迁移、侵染相关的NKX3-1、CXCR4和JARID2基因,对于黑色素瘤的治疗有着至关重要的作用,从图 5、图 6可以看出,一些miRNAs如miR-203、miR-194会同时调控HOTAIR和NKX3-1、CXCR4和JARID2,由此推测,HOTAIR可能会与NKX3-1、CXCR4和JARID2竞争性的结合这些miRNAs,从而实现对黑色素瘤的迁移、侵染相关的基因的调控,这一系列推导过程需要进一步试验给予验证。
本研究将与黑色素相关的且可能与HOTAIR结合的miRNA、HOTAIR及黑色素瘤相关蛋白相互关联构成HOTAIR紧密复杂的基因表达调控通路,为阐明其在黑色素瘤发生、发展中的作用机理及后续研究方向提供新思路,为更详尽研究lncRNA功能机制提供新线索。
4 结论HOTAIR在不同的细胞中受到多种调控因子的调控,在HeLa-S3细胞中受到Max、Pol2、USF等转录因子的调控。HOTAIR通过与miRNA和蛋白质的互作调控癌症和一些人类疾病的病理及生理学过程。在黑色素瘤中,HOTAIR可能会与MITF和NKX3-1等黑色瘤关键基因竞争性结合miR-217、miR-203和miR-194等,从而实现HOTAIR对黑色素瘤的调控。
| [1] | MAO Y S, SUNWOO H, ZHANG B, et al. Direct visualization of the co-transcriptional assembly of a nuclear body by noncoding RNAs[J]. Nat Cell Biol, 2010, 13(1): 95–101. |
| [2] | Affymetrix/Cold Spring Harbor Laboratory ENCODE Transcriptome Project. Post-transcriptional processing generates a diversity of 5'-modified long and short RNAs[J]. Nature, 2009, 457(7232): 1028–1032. DOI: 10.1038/nature07759 |
| [3] | MERCER T R, DINGER M E, MATTICK J S. Long non-coding RNAs:insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155–159. DOI: 10.1038/nrg2521 |
| [4] | SAXENA A, CARNINCI P. Long non-coding RNA modifies chromatin:epigenetic silencing by long non-coding RNAs[J]. Bioessays, 2011, 33(11): 830–839. DOI: 10.1002/bies.201100084 |
| [5] |
贺小云, 狄冉, 胡文萍, 等. 动物繁殖相关lncRNA的研究新进展[J]. 畜牧兽医学报, 2016, 47(9): 1749–1756.
HE X Y, DI R, HU W P, et al. New research progress in animal reproduction-related long noncoding RNAs[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(9): 1749–1756. (in Chinese) |
| [6] | GUTTMAN, M, RINN J L. Modular regulatory principles of large non-coding RNAs[J]. Nature, 2012, 482(7385): 339–346. DOI: 10.1038/nature10887 |
| [7] | RINN J L, KERTESZ M, WANG J K, et al. Functional demarcation of active and Silent chromatin domains in human HOX loci by noncoding RNAs[J]. Cell, 2007, 129(7): 1311–1323. DOI: 10.1016/j.cell.2007.05.022 |
| [8] | GRIER D G, THOMPSON A, KWASNIEWSKA A, et al. The pathophysiology of HOX genes and their role in cancer[J]. J Pathol, 2005, 205(2): 154–171. DOI: 10.1002/(ISSN)1096-9896 |
| [9] | TSAI M C, MANOR O, WAN Y, et al. Long noncoding RNA as modular scaffold of histone modification complexes[J]. Science, 2010, 329(5992): 689–693. DOI: 10.1126/science.1192002 |
| [10] | TANG L H, ZHANG W, SU B, et al. Long noncoding RNA HOTAIR is associated with motility, invasion, and metastatic potential of metastatic melanoma[J]. BioMed Res Int, 2013, 2013: 251098. |
| [11] | YANG Z, ZHOU L, WU L M, et al. Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation[J]. Ann Surg Oncol, 2011, 18(5): 1243–1250. DOI: 10.1245/s10434-011-1581-y |
| [12] | KIM K, JUTOORU I, CHADALAPAKA G, et al. HOTAIR is a negative prognostic factor and exhibits pro-oncogenic activity in pancreatic cancer[J]. Oncogene, 2013, 32(13): 1616–1625. DOI: 10.1038/onc.2012.193 |
| [13] | NⅡNUMA T, SUZUKI H, NOJIMA M, et al. Upregulation of miR-196a and HOTAIR drive malignant character in gastrointestinal stromal tumors[J]. Cancer Res, 2012, 72(5): 1126–1136. DOI: 10.1158/0008-5472.CAN-11-1803 |
| [14] | LI D D, FENG J P, WU T Y, et al. Long intergenic noncoding RNA HOTAIR is overexpressed and regulates PTEN methylation in laryngeal squamous cell carcinoma[J]. Am J Pathol, 2013, 182(1): 64–70. DOI: 10.1016/j.ajpath.2012.08.042 |
| [15] | NIE Y, LIU X, QU S H, et al. Long non-coding RNA HOTAIR is an independent prognostic marker for nasopharyngeal carcinoma progression and survival[J]. Cancer Sci, 2013, 104(4): 458–464. DOI: 10.1111/cas.2013.104.issue-4 |
| [16] | GUPTA R A, SHAH N, WANG K C, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature, 2010, 464(7291): 1071–1076. DOI: 10.1038/nature08975 |
| [17] | PARK C, YU N, CHOI I, et al. lncRNAtor:a comprehensive resource for functional investigation of long non-coding RNAs[J]. Bioinformatics, 2014, 30(17): 2480–2485. DOI: 10.1093/bioinformatics/btu325 |
| [18] | TAMURA K, DUDLEY J, NEI M, et al. MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596–1599. DOI: 10.1093/molbev/msm092 |
| [19] | CHEN G, WANG Z Y, WANG D Q, et al. LncRNADisease:a database for long-non-coding RNA-associated diseases[J]. Nucleic Acids Res, 2013, 41(D1): 983–D986. DOI: 10.1093/nar/gks1099 |
| [20] | LIU C N, BAI B Y, SKOGERBØ G, et al. NONCODE:an integrated knowledge database of non-coding RNAs[J]. Nucleic Acids Res, 2005, 33(D1): D112–D115. |
| [21] | HOU M, TANG X, TIAN F, et al. AnnoLnc:a web server for systematically annotating novel human lncRNAs[J]. BMC Genomics, 2016, 17(1): 931. DOI: 10.1186/s12864-016-3287-9 |
| [22] | LI J H, LIU S, ZHOU H, et al. starBase v2.0:decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J]. Nucleic Acids Res, 2014, 42(D1): D92–D97. DOI: 10.1093/nar/gkt1248 |
| [23] | ZHAO Z, BAI J, WU A W, et al. Co-LncRNA:investigating the lncRNA combinatorial effects in GO annotations and KEGG pathways based on human RNA-Seq data[J]. Database, 2015, 2015: bav082. |
| [24] | KACZKOWSKI B, TANAKA Y, KAWAJI H, et al. Transcriptome analysis of recurrently deregulated genes across multiple cancers identifies new pan-cancer biomarkers[J]. Cancer Res, 2016, 76(2): 216–226. DOI: 10.1158/0008-5472.CAN-15-0484 |
| [25] | ALPHONSE P Jr, HOOD N, LYN S, et al. MP-4.09:TargetScan®:a novel approach for outpatient prostate biopsy with the potential for use as an aid to focal prostate therapy[J]. Urology, 2008, 72(5S): S86–S87. |
| [26] | BIRNEY E, STAMATOYANNOPOULOS J A, DUTTA A, et al. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project[J]. Nature, 2007, 447(7146): 799–816. DOI: 10.1038/nature05874 |
| [27] | KAPRANOV P, CHENG J, DIKE S, et al. RNA maps reveal new RNA classes and a possible function for pervasive transcription[J]. Science, 2007, 316(5830): 1484–1488. DOI: 10.1126/science.1138341 |
| [28] | GAO J Z, LI J, DU J L, et al. Long non-coding RNA HOTAIR is a marker for hepatocellular carcinoma progression and tumor recurrence[J]. Oncol Lett, 2016, 11(3): 1791–1798. |
| [29] | FANG S, GAO H Y, TONG Y, et al. Long noncoding RNA-HOTAIR affects chemoresistance by regulating HOXA1 methylation in small cell lung cancer cells[J]. Lab Invest, 2016, 96(1): 60–68. DOI: 10.1038/labinvest.2015.123 |
| [30] | AFTAB M N, DINGER M E, PERERA R J. The role of microRNAs and long non-coding RNAs in the pathology, diagnosis, and management of melanoma[J]. Arch Biochem Biophys, 2014, 563: 60–70. DOI: 10.1016/j.abb.2014.07.022 |
| [31] | DONG L J, HU L N. HOTAIR promotes proliferation, migration, and invasion of ovarian cancer SKOV3 cells through regulating PIK3R3[J]. Med Sci Monit, 2016, 22: 325–331. DOI: 10.12659/MSM.894913 |
| [32] | WAN L D, KONG J L, TANG J L, et al. HOTAIRM1 as a potential biomarker for diagnosis of colorectal cancer functions the role in the tumour suppressor[J]. J Cell Mol Med, 2016, 20(11): 2036–2044. DOI: 10.1111/jcmm.2016.20.issue-11 |
| [33] | YAN J, DANG Y N, LIU S Y, et al. LncRNA HOTAIR promotes cisplatin resistance in gastric cancer by targeting miR-126 to activate the PI3K/AKT/MRP1 genes[J]. Tumor Biol, 2016, 37(12): 16345–16355. DOI: 10.1007/s13277-016-5448-5 |
| [34] | NEAGU M, CONSTANTIN C, MANDA G, et al. Biomarkers of metastatic melanoma[J]. Biomark Med, 2009, 3(1): 71–89. DOI: 10.2217/17520363.3.1.71 |



