羊巴贝斯虫病(ovine babesiosis)是由媒介蜱传播的一种血液原虫病,现已报道并公认的病原主要有绵羊巴贝斯虫(Babesia ovis)、莫氏巴贝斯虫(B. motasi)和粗糙巴贝斯虫(B. crassa)。绵羊巴贝斯虫致病性最强,莫氏巴贝斯虫次之,粗糙巴贝斯虫几乎没有致病性。绵羊巴贝斯虫和莫氏巴贝斯虫的传播媒介分别为扇头蜱(Rhipicephalus)和血蜱(Haemaphysalis)属的蜱种,粗糙巴贝斯虫的传播媒介尚不清楚[1]。该病在世界范围内广泛分布,严重影响养羊业的健康持续发展[2]。
我国在19世纪80年代,于黑龙江和四川省先后报道了羊的巴贝斯虫病,此后在山西、云南、甘肃、河南等省也相继报道了该病的存在[3]。这些报道都只是病例报道,并没有分离到病原,所以无法确认引起我国羊的巴贝斯虫病的病原种类。中国农业科学院兰州兽医研究所通过动物感染试验,自我国甘肃、新疆、河北、河南、辽宁和湖北等省份前后共分离到9株羊的巴贝斯虫。通过生物学特征和分子分类研究表明感染我国羊的巴贝斯虫为两个种——莫氏巴贝斯虫和羊巴贝斯虫未定种(Babesia sp.),而且莫氏巴贝斯虫内可能存在不同的亚种[4-7]。
血小板反应蛋白相关匿名蛋白(thrombospondin-related anonymous protein,TRAP)是由微线体分泌的一种Ⅰ型跨膜蛋白,大部分顶复门原虫中都发现了TRAP家族蛋白的存在,如疟原虫(Plasmodium)的TRAP、CTRP和MTRAP(PfTRAP)、弓形虫(Toxoplasma)的TgMIC2、隐孢子虫(Cryptosporidium)的TRAP-C1、新孢子虫(Neospora)的NcMIC2、艾美尔球虫(Eimeria)(EtMIC2) 和巴贝斯虫的TRAP[8]。通过对疟原虫的研究发现,TRAP与顶复门原虫运动和入侵宿主细胞关系密切[9],因此,现已将其作为研制抗顶复门寄生虫药物和疫苗的靶蛋白[10]。目前对巴贝斯虫TRAP的研究,仅报道了其基因的序列及表达产物的抗原性[11-12],尚没有对其基因功能的研究报道,该蛋白质在巴贝斯虫内的功能与疟原虫的一致与否,尚无定论。然而在开展这一工作前,首先必须要弄清羊巴贝斯虫trap基因的结构特征;而且对于我国羊巴贝斯虫分类地位的研究,虽然已获得较多的分子分类学研究数据,但尚需进行数据补充。
笔者以我国分离的2种6株羊源巴贝斯虫为研究对象,测定了其trap基因全长序列。并通过生物信息学分析,确定其基因/蛋白质结构特征,为后期研究巴贝斯虫trap基因的功能奠定基础;同时基于trap基因序列,对我国羊源巴贝斯虫的遗传进化关系做了分析,为进一步理清我国羊巴贝斯虫的种类做必要的数据补充。
1 材料与方法 1.1 试验时间、地点该试验于2016年3—10月在中国农业科学院兰州兽医研究所外寄生虫与虫媒疫病创新团队实验室完成。
1.2 主要试剂、虫株及实验动物Trans5α Chemically Competent Cell购自北京全式金生物技术有限公司;DNA聚合酶购自宝生物(大连)工程有限公司;pGEM-T Easy载体、T4 DNA连接酶购自Promega公司;DNA胶回收试剂盒、质粒提取试剂盒购自Omega Bio-Tek公司;基因组DNA提取试剂盒Gentra Puregene DNA purification kit购自QIAGEN公司;去白细胞滤器购自南京双威生物医学科技有限公司。
本试验涉及的2种6株羊源巴贝斯虫分离自我国甘肃、河北和新疆等地(表 1),由兰州兽医研究所外寄生虫与虫媒疫病创新团队提供。
|
|
表 1 本试验所涉及顶复门寄生虫trap/mic2基因序列的相关信息 Table 1 Information of trap/mic2 gene of parasites in Apcomplexa used in this study |
6月龄绵羊购自甘肃景泰县。经血涂片镜检和PCR检测,梨形虫和无浆体呈阴性的动物购入实验室,饲喂于兰州兽医研究所普通级别动物房。在实验室饲养1月后,手术切除脾,定期血涂片镜检和PCR检查(持续1月),梨形虫和无浆体呈阴性的动物用于本试验。
1.3 感染动物、裂殖子的纯化及DNA的提取对6只除脾绵羊分别静脉接种液氮冻存的羊巴贝斯虫未定种新疆株、敦煌株与莫氏巴贝斯虫临潭株、天祝株、宁县株和河北株的含虫血各10 mL。接种后每天肌肉注射1 mg地塞米松注射液并进行血涂片检查,待染虫率达到8%~10%时,静脉采集抗凝血(20%的柠檬酸钠作为抗凝剂),用于羊的巴贝斯虫裂殖子纯化。
将抗凝血转入干净的15 mL离心管中,3 000 r·min-1 4 ℃离心10 min,丢弃上清和白细胞层。然后用PBS将红细胞沉淀洗三遍,同前离心,红细胞沉淀用10倍体积的PBS重新悬浮,用去白细胞滤器去除剩余的白细胞。滤过的红细胞悬液同前离心,沉淀参照G. Q. Guan等报道的方法纯化羊的巴贝斯虫裂殖子[13],纯化的裂殖子-70 ℃保存备用。
取300 μL纯化的裂殖子,应用基因组提取试剂盒Gentra Puregene DNA purification kit进行虫体基因组DNA的提取,最后将DNA溶解于80 μL的Buffer AE缓冲液中,-20 ℃保存备用。
1.4 trap基因的克隆用本实验室应用二代测序技术测定的莫氏巴贝斯虫临潭株G7单克隆株和羊巴贝斯虫未定种新疆株G5单克隆株全基因组序列数据,建立两种巴贝斯虫全基因组序列本地数据库。然后用牛巴贝斯虫(XM001609738) 和双芽巴贝斯虫(XM_012911849)trap基因序列分别BLAST莫氏巴贝斯虫和羊巴贝斯虫未定种全基因组序列数据。以BLAST获得的scaffold中的序列片段为模板,分别设计莫氏巴贝斯虫和羊巴贝斯虫未定种trap基因扩增的PCR引物Bmtrap-S/Bmtrap-AS和Bsptrap-S/Bsptrap-AS,引物序列见表 2。
|
|
表 2 羊巴贝斯虫trap基因PCR引物 Table 2 PCR primers of ovine Babesia trap gene |
以感染我国羊的2种6株巴贝斯虫基因组DNA为模板,PCR扩增trap基因全长序列。PCR扩增产物胶回收后与pGEM-T Easy载体连接,连接产物转化Trans5α感受态细胞,37 ℃过夜培养。挑取白色菌落,接种于4 mL含50 μg·mL-1氨苄青霉素的LB培养液,37 ℃ 200 r·min-1过夜培养,提取质粒,PCR鉴定。阳性的单克隆重组菌送生工生物工程(上海)股份有限公司进行测序。
1.5 巴贝斯虫trap基因的结构分析首先用GENSCAN软件(http://genes.mit.edu/GENSCAN.html)和ORF Finder软件(www.ncbi.nlm.nih.gov/gorf)分析所获序列内含子和外显子的个数以及开放阅读框(ORF)大小,DNASTAR软件包中的EditSeq软件预测所获序列ORF编码的氨基酸序列,BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)分析核苷酸序列和其编码的氨基酸序列,确定其为trap基因的可能性;然后与牛巴贝斯虫、双芽巴贝斯虫、吉氏巴贝斯虫和田鼠巴贝斯虫trap基因序列比较(序列信息见表 1),用GSDS 2.0 (Gene Structure Display Server 2.0,http://gsds.cbi.pku.edu.cn/)分析其基因结构;最后用SignalP软件(www.cbi.dtu.dk/services/SignalP)在线预测ORF编码氨基酸序列的信号肽序列,MyHits软件(http://myhits.isb-sib.ch/cgi-bin/motif_scan)分析编码氨基酸序列保守的蛋白质结构域,用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)分析其跨膜区,并利用CLC Genomics Workbench 7进行氨基酸序列比对,注释鉴定的蛋白保守结构域区域。将所获序列及分析注释信息提交GenBank,获序列号。
1.6 trap基因序列比对及系统发生树构建用DNASTAR软件包中的MegAlign软件,采用Clustal W方法进行序列比对,分析顶复门寄生虫trap/mic2基因核苷酸和氨基酸序列相似性(序列信息见表 1),然后用MEGA 6.0软件中的Boststrap Test of Phylogeny分析核苷酸序列和氨基酸序列,分别构建的Neighbor-Joining(NJ)和Maximum Parsimany(MP)系统发生树,分析我国分离的6株羊巴贝斯虫的分类地位。
2 结果 2.1 trap基因克隆利用设计的两对引物,以感染我国羊的6株巴贝斯虫基因组DNA为模板进行PCR扩增,获得大小约为2 500 bp的基因片段(图 1),通过测序表明,自羊巴贝斯虫未定种新疆株和敦煌株扩增获得的片段大小为2 338 bp,莫氏巴贝斯虫宁县株和河北株的片段为2 715 bp,莫氏巴贝斯虫临潭株和天祝株的片段分别2 567和2 609 bp。
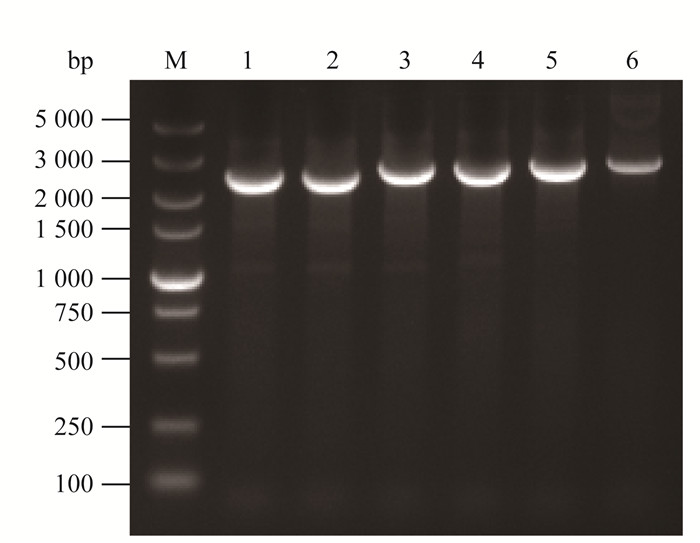
|
M. DL5000 DNA相对分子质量标准;1~2.巴贝斯虫未定种新疆株和敦煌株;3~6.莫氏巴贝斯虫临潭株、天祝株、宁县株和河北株 M. DL5000 DNA marker; 1-2. Babesia sp. Xinjiang and Dunhuang; 3-6. B. motasi Lintan, Tianzhu, Ningxian and Hebei 图 1 trap基因的PCR扩增 Figure 1 PCR amplification of trap gene |
通过与已报道的牛巴贝斯虫、双芽巴贝斯虫、吉氏巴贝斯虫和田鼠巴贝斯虫trap基因序列的比较及生物信息学分析结果表明,这些羊巴贝斯虫trap基因的内含子和外显子的数目各不相同。羊巴贝斯虫未定种新疆株和敦煌株具有5个内含子和6个外显子,莫氏巴贝斯虫临潭株和天祝株有4个内含子和5个外显子,莫氏巴贝斯虫河北株和宁县株具3个内含子和4个外显子(图 2);而且其ORF大小也存在较大差异,莫氏巴贝斯虫宁县株/河北株、莫氏巴贝斯虫天祝株、莫氏巴贝斯虫临潭株及巴贝斯虫未定种新疆株/敦煌株的ORF大小分别为2 286、2 142、2 100和1 944 bp(表 2);通过对其编码的氨基酸序列的分析结果显示,莫氏巴贝斯虫TRAP的信号肽(signal peptide)区域位于1—23位氨基酸,巴贝斯虫未定种的位于1—26位氨基酸。且其都具有TRAP蛋白家族特征性的结构域,如血管性血友病因子A样结构域(von Willebrand factor-like A domain, vWFA)、血小板反应蛋白Ⅰ型重复域(thrombospondin type-Ⅰ repeat domain, TSR)、跨膜域(transmembrane domain)和细胞质内尾部结构域(cytoplasmic tail domain,CTD)。vWFA中具有一个金属离子依赖性黏附位点(metal ion-dependent adhesion site, MIDAS)域,在MIDAS中具有可能与受体识别和细胞黏附相关的特征性的非连续的D-X-S-X-S氨基酸组成(X为任意氨基酸残基),TSR中有与细胞绑定相关的色氨酸和半胱氨酸残基,跨膜域中有菱形体蛋白酶切割位点,CTD中有末端色氨酸残基(图 3中用*标出)及肌动蛋白-肌球蛋白马达连接相关的YXXϕ域(这些TRAP蛋白的功能性结构域标示在图 3中,且重要氨基酸残基显示为灰色)。这些分析结果表明,本试验成功扩增出了6株羊的巴贝斯虫trap基因的全长。所以将该序列信息上传GenBank,获得的序列号见表 1。
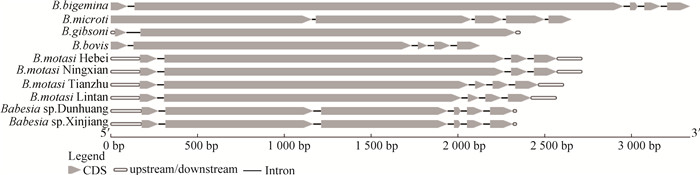
|
图 2 巴贝斯虫trap基因结构 Figure 2 Structure of Babesia trap genes |

|
图 3 巴贝斯虫TRAP蛋白结构 Figure 3 Structure of Babesia TRAP proteins |
通过对顶复门寄生虫trap基因核苷酸和蛋白质氨基酸的序列比对分析发现其相似性差异较大,核苷酸和氨基酸序列的相似性分别为30.4%~100%和8.8%~100%,而且牛巴贝斯虫和双芽巴贝斯虫与其他巴贝斯虫核苷酸和氨基酸的相似性在30.6%~57.9%和54.1%之间。6株羊的巴贝斯虫中,巴贝斯虫未定种新疆株和敦煌株氨基酸和核苷酸的相似性最高(100%),莫氏巴贝斯虫河北株和宁县株的为99.9%,莫氏巴贝斯虫临潭株和天祝株的核苷酸和氨基酸的相似性分别为99.8%和99.9%;莫氏巴贝斯虫和巴贝斯虫未定种之间核苷酸和氨基酸的相似性分别为55.4%~56.1%和43.6%~46.9%,而莫氏巴贝斯虫河北株/宁县株与莫氏巴贝斯虫临潭株/天祝株之间核苷酸和氨基酸的相似性分别为74.0%~74.6%和59.1%(表 3)。这些结果表明,6株羊的巴贝斯虫被分成3组——巴贝斯虫未定种新疆株/敦煌株、莫氏巴贝斯虫河北株/宁县株和莫氏巴贝斯虫临潭株/天祝株。
|
|
表 3 顶复门不同寄生虫虫种trap基因核苷酸序列和氨基酸序列相似性 Table 3 Similarities of trap gene nucleotide and amino acid sequences of different parasite species in Apicomplexa |
利用MEGA6软件,基于核苷酸序列和氨基酸序列分别构建了NJ和MP系统发生树,结果基本与相似性比较的结果相一致,不同种属的寄生虫各占据不同的大枝。对于巴贝斯虫,吉氏巴贝斯虫和田鼠巴贝斯虫与反刍动物的巴贝斯虫并没有分到一枝上,但反刍动物的巴贝斯虫在构建的4棵树中都被分到一大枝上。羊的6株巴贝斯虫被分为两枝——巴贝斯虫未定种和莫氏巴贝斯虫,同时莫氏巴贝斯虫又被分为两小枝——莫氏巴贝斯虫河北株/宁县株和莫氏巴贝斯虫临潭株/天祝株(图 4)。
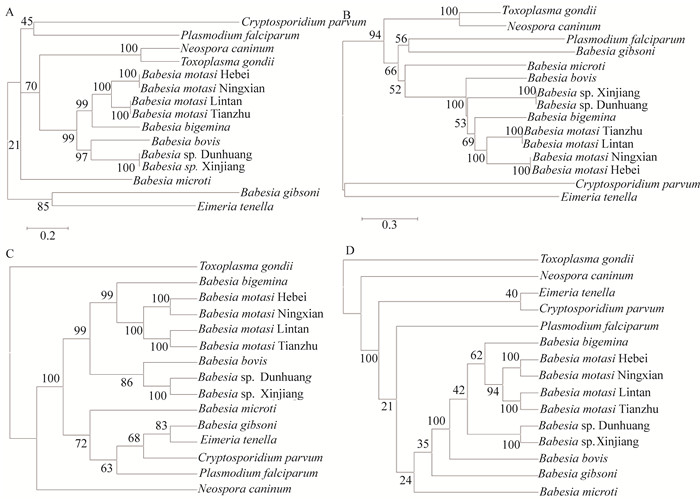
|
A、C. trap基因核苷酸序列;B、D. trap基因推导氨基酸序列;A、B和C、D系统发生树构建分别采用NJ和MP两种方法 These trees were calculated using the NJ (A/B) and MP (C/D) methods. Trees were constructed with trap nucleotide sequences (A/C) and deduced amino acid sequences (B/D) 图 4 基于顶复门寄生虫trap基因核苷酸序列和推导氨基酸序列的系统发生树 Figure 4 Phylogenetic trees based on trap nucleotide and deduced amino acid sequences of Apicomplexa parasites |
顶复门寄生虫为细胞内寄生的原虫,所以其在寄生过程中需要不断移动和入侵宿主细胞。然而其没有鞭毛和纤毛等运动器官,通过对弓形虫和疟原虫的研究表明,他们是以肌动蛋白-肌球蛋白马达为动力装置的滑行运动来移动[14]。TRAP在顶复门寄生虫滑行运动过程中,承担着“腿脚”的角色:一方面其胞外N端的TSR和vWFA结构域与细胞表面受体连接,使得虫体可以稳定黏附于细胞表面;另一方面,其胞内C端的CTD结构域与醛缩酶相互作用,将肌动蛋白-肌球蛋白马达产生的动力传输到虫体表面,从而使得虫体有动力在细胞表面产生移动。所以TRAP既是参与顶复门寄生虫运动的重要分子,又是入侵过程中黏附宿主细胞的配体[8]。由于该蛋白家族与顶复门寄生虫运动和入侵的密切关系,现已将其作为研制抗顶复门寄生虫药物和疫苗的靶蛋白[10]。然而除了在弓形虫和疟原虫中对该基因的功能进行了较为深入的研究外,顶复门的其他寄生虫中尚没有这方面的研究。是否TRAP在其他顶复门寄生虫中也具有与在弓形虫和疟原虫中相似的功能,尚不清楚。到目前为止,在巴贝斯虫中,只有对牛巴贝斯虫和吉氏巴贝斯虫TRAP核苷酸和氨基酸序列进行了分析,并对其抗原性和抗血清阻断裂殖子入侵宿主细胞的效果进行了评价[11-12]。对于田鼠巴贝斯虫和双芽巴贝斯虫,只在全基因组序列中对其做了注释。所以有必要对巴贝斯虫TRAP的功能进行研究,从而确认其在巴贝斯虫运动和入侵宿主细胞过程中的作用。
本研究以在我国普遍流行的羊源巴贝斯虫为研究对象,应用BLAST测定的全基因组序列和PCR扩增的方法,成功扩增获得6株羊的巴贝斯虫的trap基因全长序列。通过生物信息学分析和与前期研究获得的莫氏巴贝斯虫临潭株和羊巴贝斯虫未定种新疆株trap基因的cDNA序列(待发表数据)比对,确定6株羊巴贝斯虫trap基因都具有内含子,而且其内含子的数目和ORF大小在不同虫株中并不相同。羊巴贝斯虫未定种新疆株/敦煌株、莫氏巴贝斯虫临潭株/天祝株和宁县株/河北株分别具有5、4和3个内含子;莫氏巴贝斯虫宁县株/河北株、天祝株、临潭株及巴贝斯虫未定种新疆株/敦煌株的ORF大小分别为2 286、2 142、2 100和1 944 bp。这些结果与现已公布的牛巴贝斯虫、双芽巴贝斯虫、吉氏巴贝斯虫和田鼠巴贝斯虫trap基因序列的结构基本一致,从而表明巴贝斯虫trap基因都具有内含子,而且其ORF大小差异较大。这种巴贝斯虫不同种/株之间的基因差异,可能与虫株地理隔离及传播媒介蜱不同产生的进化或采用的剪接机制不同有关,而这种差异是否与巴贝斯虫不同种/株致病性差异存在相关性,尚待进一步研究。文献报道,顶复门寄生虫TRAP蛋白是一个Ⅰ型跨膜蛋白,表达储存在微线体中。当虫体入侵宿主细胞时,TRAP首先被转运到虫体前端,其胞外vWFA和TSR结构域中的MIDAS和色氨酸及半胱氨酸残基参与虫体和宿主细胞的黏附和绑定;CTD中的末端色氨酸残基和YXXϕ域与寄生虫醛缩酶相互作用,将肌动蛋白-肌球蛋白马达产生的动力传递到虫体表面,使得虫体可以在细胞表面发生位移;虫体穿入宿主细胞膜后,YXXϕ域承担起信号识别的作用,传递信号到微线体,将TRAP转运到虫体后端,菱形蛋白酶在跨膜域中的菱形体蛋白酶切割位点将TRAP蛋白胞外部分切除,从而辅助虫体完成入侵宿主细胞的过程[8-9, 14]。本研究通过对预测的TRAP蛋白核苷酸序列结构的分析表明,6株羊巴贝斯虫trap氨基酸序列中都具有TRAP蛋白家族保守的vWFA、TSR、CTD和跨膜域等重要的功能结构域。表明我们所获羊巴贝斯虫trap基因的正确性,同时推测羊巴贝斯虫TRAP的功能可能与其他顶复门寄生虫的相一致,但尚待后期通过功能研究进一步确认。另外,在进行羊巴贝斯虫trap基因PCR引物设计过程中,笔者用牛巴贝斯虫和双芽巴贝斯虫trap基因序列BLAST本实验室建立的莫氏巴贝斯虫临潭株和羊巴贝斯虫未定种新疆株的全基因组序列本地数据库时发现,这两株羊的巴贝斯虫trap基因为单拷贝基因。这些研究结果为将来开展羊巴贝斯虫trap基因功能的研究提供可靠的基因序列数据信息。
在本实验室前期对我国分离的羊巴贝斯虫生物学特性与分子分类的研究中发现,我国现分离到的羊巴贝斯虫有两个种——莫氏巴贝斯虫和羊巴贝斯虫未定种,而且在使用不同的靶基因(28S rRNA、ITS、18S rRNA、HSP90、COI、COX3、COB、RPS8和RAP-1) 作为分类标志获得的结果都是一致的。但是在使用不同的基因作为分类标志时,对于莫氏巴贝斯虫不同株的分类情况并不一致,如在使用28S rRNA和18S rRNA时所有的莫氏巴贝斯虫被分到同一枝上,且没有明显的聚类小分枝;在使用ITS、HSP90、COI、COB、COX3和RPS8时,莫氏巴贝斯虫被分成两个小分枝——河北株/宁县株和临潭株/天祝株;但是在使用RAP-1时被分成宁县株/临潭株/天祝株和河北株两个小分枝[5-6, 15-19]。本研究对测定的6株羊巴贝斯虫trap基因的核苷酸序列和推导的氨基酸序列进行了比对,并基于trap基因核苷酸序列和推导的氨基酸序列构建了NJ和MP系统发生树。结果6株羊源巴贝斯虫被分成3组——巴贝斯虫未定种新疆株/敦煌株(核苷酸和氨基酸序列相似性都为100%)、莫氏巴贝斯虫河北株/宁县株(核苷酸和氨基酸序列相似性都为99.9%)和莫氏巴贝斯虫临潭株/天祝株(核苷酸和氨基酸序列相似性分别为99.8%和99.9%)。莫氏巴贝斯虫和巴贝斯虫未定种之间核苷酸和氨基酸的相似性分别为55.4%~56.1%和43.6%~46.9%,而莫氏巴贝斯虫河北株/宁县株与临潭株/天祝株之间核苷酸和氨基酸序列的相似性分别为74.0%~74.6%和59.1%。同时在构建的系统发生树中,羊的6株巴贝斯虫被分为两枝——巴贝斯虫未定种和莫氏巴贝斯虫,同时莫氏巴贝斯虫又被分为两小枝——莫氏巴贝斯虫河北株/宁县株和莫氏巴贝斯虫临潭株/天祝株。这一结果与前期的研究结果相一致——我国分离的羊巴贝斯虫存在两个种,羊巴贝斯虫未定种和莫氏巴贝斯虫;对于莫氏巴贝斯虫种内的分类情况,基本与以ITS、HSP90、COI、COB、COX3和RPS8为标志基因的情况一致,表明莫氏巴贝斯虫可以分为两个亚种,代表株分别为河北株/宁县株和临潭株/天祝株。这一结果为理清我国羊巴贝斯虫的分类地位提供了有力的数据补充。
4 结论克隆获得了6株羊巴贝斯虫trap基因的全长序列,并通过生物信息学分析阐明了其核苷酸序列和推导的氨基酸序列的结构特征,为后期研究巴贝斯虫trap基因的功能提供了理论依据;同时以trap基因为分子标志,对我国羊巴贝斯虫的分类地位进行了分析,表明我国存在两种羊的巴贝斯虫——莫氏巴贝斯虫和巴贝斯虫未定种;而且莫氏巴贝斯虫又被分为两个群,河北株/宁县株和临潭株/天祝株群。这一研究结果为进一步阐明莫氏巴贝斯虫种内可能存在两个亚种的论断提供了数据支撑。
| [1] | UILENBERG G. Babesia-a historical overview[J]. Vet Parasitol, 2006, 138(1-2): 3–10. DOI: 10.1016/j.vetpar.2006.01.035 |
| [2] | FRIEDHOFF K T. Tick-borne diseases of sheep and goats caused by Babesia, Theileria or Anaplasma spp.[J]. Parassitologia, 1997, 39(2): 99–109. |
| [3] |
杨强, 刘爱红, 刘军龙, 等. 我国十省份羊巴贝斯虫的分子流行病学调查[J]. 中国兽医科学, 2016, 46(5): 597–601.
YANG Q, LIU A H, LIU J L, et al. Molecular epidemiological investigation of ovine Babesia spp. in 10 provinces of China[J]. Chinese Veterinary Science, 2016, 46(5): 597–601. (in Chinese) |
| [4] | GUAN G Q, MA M L, MOREAU E, et al. A new ovine Babesia species transmitted by Hyalomma anatolicum anatolicum[J]. Exp Parasitol, 2009, 122(4): 261–267. DOI: 10.1016/j.exppara.2009.05.001 |
| [5] | TIAN Z C, LIU G Y, YIN H, et al. RPS8--a new informative DNA marker for phylogeny of Babesia and Theileria parasites in China[J]. PLoS One, 2013, 8(11): e79860. DOI: 10.1371/journal.pone.0079860 |
| [6] | GUAN G Q, LIU J L, LIU A H, et al. A member of the HSP90 family from ovine Babesia in China: molecular characterization, phylogenetic analysis and antigenicity[J]. Parasitology, 2015, 142(11): 1387–1397. DOI: 10.1017/S0031182015000797 |
| [7] |
苟惠天, 薛慧文, 殷宏, 等. 基于COⅠ基因序列对我国部分巴贝斯虫分类的研究[J]. 畜牧兽医学报, 2016, 47(6): 1293–1298.
GOU H T, XUE H W, YIN H, et al. Toxonomic study of some Babesia spp. based on COⅠ gene in China[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(6): 1293–1298. (in Chinese) |
| [8] | MORAHAN B J, WANG L N, COPPEL R L. No TRAP, no invasion[J]. Trends Parasitol, 2009, 25(2): 77–84. DOI: 10.1016/j.pt.2008.11.004 |
| [9] | UCHIME O, HERRERA R, REITER K, et al. Analysis of the conformation and function of the Plasmodium falciparum merozoite proteins MTRAP and PTRAMP[J]. Eukaryot Cell, 2012, 11(5): 615–625. DOI: 10.1128/EC.00039-12 |
| [10] | BAUZA K, MALINAUSKAS T, PFANDER C, et al. Efficacy of a Plasmodium vivax malaria vaccine using ChAd63 and modified vaccinia Ankara expressing thrombospondin-related anonymous protein as assessed with transgenic Plasmodium berghei parasites[J]. Infect Immun, 2014, 82(3): 1277–1286. DOI: 10.1128/IAI.01187-13 |
| [11] | ZHOU J L, FUKUMOTO S, JIA H L, et al. Characterization of the Babesia gibsoni P18 as a homologue of thrombospondin related adhesive protein[J]. Mol Biochem Parasitol, 2006, 148(2): 190–198. DOI: 10.1016/j.molbiopara.2006.03.015 |
| [12] | GAFFAR F R, YATSUDA A P, FRANSSEN F F J, et al. A Babesia bovis merozoite protein with a domain architecture highly similar to the thrombospondin-related anonymous protein (TRAP) present in Plasmodium sporozoites[J]. Mol Biochem Parasitol, 2004, 136(1): 25–34. DOI: 10.1016/j.molbiopara.2004.02.006 |
| [13] | GUAN G Q, CHAUVIN A, LUO J X, et al. The development and evaluation of a loop-mediated isothermal amplification (LAMP) method for detection of Babesia spp. infective to sheep and goats in China[J]. Exp Parasitol, 2008, 120(1): 39–44. DOI: 10.1016/j.exppara.2008.04.012 |
| [14] | SOLDATI D, MEISSNER M. Toxoplasma as a novel system for motility[J]. Curr Opin Cell Biol, 2004, 16(1): 32–40. DOI: 10.1016/j.ceb.2003.11.013 |
| [15] | GOU H T, GUAN G Q, MA M L, et al. Phylogenetic analysis based on 28S rRNA of Babesia spp. in ruminants in China[J]. Expe Appl Acarol, 2013, 59(4): 463–472. DOI: 10.1007/s10493-012-9607-0 |
| [16] | TIAN Z C, LIU G Y, YIN H, et al. Cytochrome c oxidase subunit Ⅲ (COX3) gene, an informative marker for phylogenetic analysis and differentiation of Babesia species in China[J]. Infect Genet Evol, 2013, 18: 13–17. DOI: 10.1016/j.meegid.2013.04.002 |
| [17] | NIU Q L, LUO J X, GUAN G Q, et al. Differentiation of two ovine Babesia based on the ribosomal DNA internal transcribed spacer (ITS) sequences[J]. Exp Parasitol, 2009, 121(1): 64–68. DOI: 10.1016/j.exppara.2008.09.021 |
| [18] | TIAN Z C, LUO J, ZHENG J F, et al. Phylogenetic analysis of Babesia species in China based on cytochrome b (COB) gene[J]. Infect Genet Evol, 2013, 13: 36–40. DOI: 10.1016/j.meegid.2012.09.001 |
| [19] | NIU Q L, VALENTIN C, BONSERGENT C, et al. Strong conservation of rhoptry-associated-protein-1(RAP-1) locus organization and sequence among Babesia isolates infecting sheep from China (Babesia motasi-like phylogenetic group)[J]. Infect Genet Evol, 2014, 28: 21–32. DOI: 10.1016/j.meegid.2014.08.028 |




