2. 河南省农业科学院动物免疫学重点实验室, 郑州 450002;
3. 河南农业大学牧医工程学院, 郑州 450002;
4. 河南牧业经济学院生物工程学院, 郑州 450046
2. The Provincial Key Laboratory of Animal Immunology, Henan Academy of Agricultural Sciences, Zhengzhou 450002, China;
3. College of Veterinary Medicine, Henan Agricultural University, Zhengzhou 450002, China;
4. The Bioengineering Department, Henan University of Animal Husbandry and Economy, Zhengzhou 450046, China
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)引起以母猪繁殖障碍、仔猪和育成猪呼吸道症状为主要特征的传染病[1]。该病于1987年首次在北美发现,之后在世界范围内迅速传播,给养猪业造成了极大的经济损失[2]。PRRSV属于动脉炎病毒科,动脉炎病毒属,其同属成员还有小鼠乳酸脱氢酶病毒(LDV)、马动脉炎病毒(EAV)和猴出血热病毒(SHFV)[3]。PRRSV分为两种主要的基因型:欧洲型(Type Ⅰ)和美洲型(Type Ⅱ)。PRRSV的基因组约15.1 kb,包括10个开放阅读框ORF1a、ORF1b、ORF2a、ORF2b、ORF3、ORF4、ORF5、ORF5a、ORF6和ORF7[4]。欧洲型和美洲型的PRRSV基因组高度变异,仅有60%的核苷酸序列相同[5]。ORF1a-ORF1b占整个基因组的4/5,编码两个大的多聚蛋白(polypeptides, pp)——PP1a和PP1ab,这些蛋白质接着被加工成16个非结构蛋白(nonstructural proteins, nsps),其中14个nsps主要参与病毒基因组的复制和转录[6], 另外2个非结构蛋白nsp2TF和nsp2N为核糖体移码的产物[7]。其他的ORFs编码8个膜蛋白,ORF2~5编码病毒4种囊膜相关蛋白GP2~5及新鉴定出来的GP5a,ORF6编码非糖基化蛋白(M蛋白),ORF7编码核衣壳(N)蛋白[8]。猪肺泡巨噬细胞(porcine alveolar macrophages,PAM)是PRRSV感染和增殖的天然宿主。
定量蛋白质组学技术广泛应用于分析病毒和宿主细胞之间的相互作用。比如流感病毒[9]、人类呼吸综合征病毒[10]、圆环病毒2型[11]、口蹄疫病毒[12]、传染性法氏囊病毒[13]、乙型肝炎病毒[14]、非洲猪瘟病毒[15]等,这些研究为阐明病毒的致病机制提供理论依据。到目前为止,国内外学者曾经采用不同的定量蛋白质组学方法研究PRRSV感染。例如:H. M. Zhang等利用2-DE结合MS的方法鉴定出PRRSV感染PAM细胞后23个与代谢和应激相关的蛋白质[16]。S. Q. Xiao等利用双向荧光差异凝胶电泳分析PRRSV感染PAM细胞后的差异蛋白质[17]。Q. Lu等利用iTRAQ技术鉴定出了PRRSV感染PAM细胞后不同时期共有160个蛋白质有显著差异[18]。R. Luo等用定量蛋白质组的方法分析了PRRSV感染PAM细胞后磷酸化蛋白的变化情况,分别在12和36 hpi鉴定出了242和249个磷酸化蛋白变化显著,并揭示了PRRSV主要通过激活MAPK通路和NF-κB通路诱导炎性因子的产生[19]。
本研究采用Label-free定量技术结合liquid chromatography-tandem mass spectrometry(LC-MS/MS)研究PRRSV经典美洲株BJ-4感染PAM后细胞蛋白质的变化,是截止到目前为止相关研究中鉴定出的蛋白质覆盖率最高的报道,为进一步研究PRRSV的致病机制奠定了基础。
1 材料与方法 1.1 病毒和PAM细胞制备PRRSV BJ-4株由中国农业大学杨汉春教授惠赠。病毒在PAM细胞上增殖,滴度是108.56 TCID50·mL-1。PAM细胞采自4~6周无特定病原体仔猪(PRRSV、PCV2、PRV、CSFV、PPV、SFV等抗原、抗体检测均为阴性)。PAM细胞用RPMI 1640培养基培养,含有100单位的青霉素和100 μg·mL-1的链霉素及10%的胎牛血清。
1.2 病毒感染PAM细胞取出后在5% CO2浓度的培养箱,10%胎牛血清的1640培养基中培养16 h。未贴壁的PAM细胞在病毒接种前用1640培养基轻轻洗去。之后,将MOI=1的PRRSV BJ-4株病毒接种PAM细胞,同时设置未感染对照,于感染24 h后收样。无论是感染组还是未感染对照组都设三个独立的生物学重复。病毒的增殖用间接免疫荧光法确认。
1.3 蛋白质的提取及酶解病毒处理后的细胞用预冷的PBS洗涤1遍,胰蛋白酶消化后,300 g离心10 min。将裂解液(Lysis Buffer)加入到每个细胞样品中,裂解液配方:8 mol·L-1尿素, 2 mol·L-1硫尿, 4%CHAPS, 20 mmol·L-1 Trisbase, 30 mmol·L-1 DTT, 0.5% Bio-lyte(pH 3.5~10)。冰上裂解30 min,超声处理细胞后,将裂解液转移至EP管中,14 000 g离心20 min。按照1:3比例加入预冷丙酮,涡旋振荡后冰上沉淀30 min,14 000 g离心10 min,弃上清。EP管开口使丙酮尽量挥发,每个样品中加入5 mol·L-1尿素,使蛋白质进一步溶解完全。然后按照尿素体积1:4加入40 mmol·L-1 NH4HCO3,再分别加入DTT,4 ℃放置1 h及5倍DTT体积的100 mmol·L-1 IAA 4 ℃避光放置1 h。样品中加入胰蛋白酶37 ℃酶解过夜。次日,各样品中加入1 μL甲酸终止酶切。4 ℃ 14 000 g离心20 min后,取上清用真空离心浓缩仪抽干,再用1‰甲酸溶解,待上质谱。
1.4 LC-MS/MS分析用1%甲酸溶解干燥好的样品,涡旋振荡后,4 ℃ 14 000 g离心20 min,上清转移入上样管中,避免产生气泡。本研究使用的是Easy-nLC 1000和Q Executive(Thermo fisher Scientific, USA)液质联用的串联质谱;流动相A:1‰甲酸水,流动相B:1‰甲酸乙腈水溶液,每一针上样体积是8 μL,每个样品三个重复。
1.5 数据分析将质谱结果导入PEAKS 7.0软件,使用猪的非冗余数据库(2016年7月3日从NCBI上下载)进行检索。数据库检索参数设置如下:母离子质量数误差范围(parent mass error tolerance):2.00×10-5, 碎片离子误差范围(fragment mass error tolerance): 0.05 u, 酶为经TPCK处理的胰蛋白酶(Enzyme: Trypsin), 肽段非特异性断裂数为0(allow non-specific cleavage at: none), 每条肽段最多有两个漏切位点(maximum missed clravages per peptide:2),固定修饰选Carbamidomethyl,可变修饰选Oxidation,每条肽段最多有3种翻译后修饰类型。搜库结束后用假阳性率FDR<1%和鉴定到的蛋白中特有肽段(unique peptide)≥1这两个条件对搜库结果进行筛选。
1.6 蛋白质的定量分析运用PEAKS 7.0的PEAKS Q功能对数据库检索结果进行定量分析。PEAKS Q根据峰面积进行特征检测并计算肽段比率。条件设置如下:保留时间(retention time shift tolerance):1 min;质量误差范围(mass error tolerance):1.5×10-5;特有肽段(unique peptide)≥1;电荷范围(charge between)2~8;蛋白质和肽段都是差异倍数(fold change)≥1.5,P≤0.05(significant≥13)。
1.7 生物信息学分析运用ClueGO(v2.1.7) 对蛋白质代谢通路富集,ClueGO是Cytoscape的一个插件,能全面的揭示蛋白质参与各种生物信号通路的情况及蛋白质之间的相互作用,进而有效提高对大数据的解析能力。物种选择Sus Scrofa;猪Biological process数据库于2016年7月12日下载,猪KEGG数据库于2016年7月7日下载,输入的是蛋白质的gi号,统计检验采用超几何富集分析(statistical test used=enrichment Right-sided hypergeometric test);校正方法是多重比较(correction method used=bonferroni step down),P<0.05;GO(gene ontology)水平3~8,Kappa得分的阈值设为0.4,每个group至少有一个term (initial group size=1),共享组的比例根据输入的数据设置(sharing group percentage)。蛋白质相互作用网络用在线软件OMICSBEAN进行分析(http://www.omicsbean.cn/)。
1.8 RNA的提取和荧光定量PCR用TRIzol reagent处理PRRSV感染后的PAM细胞而提取总RNA,再用Prime Script TM RT reagent Kit with gDNA Eraser反转录试剂盒将RNA反转录成cDNA,用BIO-RAD CFX ConnectTM荧光定量PCR检测系统检测,使用试剂Power SYBR® Green PCR Master Mix (Applied Biosystems® Cat: 4367659)。每个cDNA的扩增值有三个重复,每个基因的扩增引物见表 1,数据用相对定量的ΔΔCt方法进行分析。
|
|
表 1 荧光定量PCR所扩增的基因名称及引物序列 Table 1 The sequences of primers used in this study for real-time RT-PCR |
用病毒感染后24 h的细胞及未感染的对照,用RIPA裂解后进行SDS-PAGE。之后,蛋白质被电转至0.45 μm的PVDF膜上,5%脱脂奶室温下封闭2 h,分别用兔抗MX1多抗、兔抗IFIT3多抗4 ℃孵育1 h,用PBST洗涤3次,然后用HRP标记的山羊抗兔IgG抗体作用2 h,PBST洗膜三次,用北京普利莱基因技术公司的超敏发光液进行检测。内参检测GAPDH,按照试剂使用说明进行。
2 结果 2.1 LC-MS/MS检测对未感染的细胞样品和感染后24 h样品进行了Label Free LC-MS/MS检测,每个样品三个重复。分别鉴定出3 779和4 156个蛋白质,并通过PEAKS 7.0软件进行了定量分析。PRRSV感染后共鉴定出差异显著蛋白质430个,其中有171个上调蛋白质和259个下调蛋白质,表 2、3列出了部分与我们研究相关的蛋白质。
|
|
表 2 Label-free鉴定出的PRRSV感染后显著上调表达的部分蛋白质 Table 2 Part of significant up-regulated proteins identified by label-free analysis of PRRSV infected PAM |
|
|
表 3 Label-free鉴定出的PRRSV感染后显著下调表达的部分蛋白质 Table 3 Part of significant down-regulated proteins identified by label-free analysis of PRRSV infected PAM |
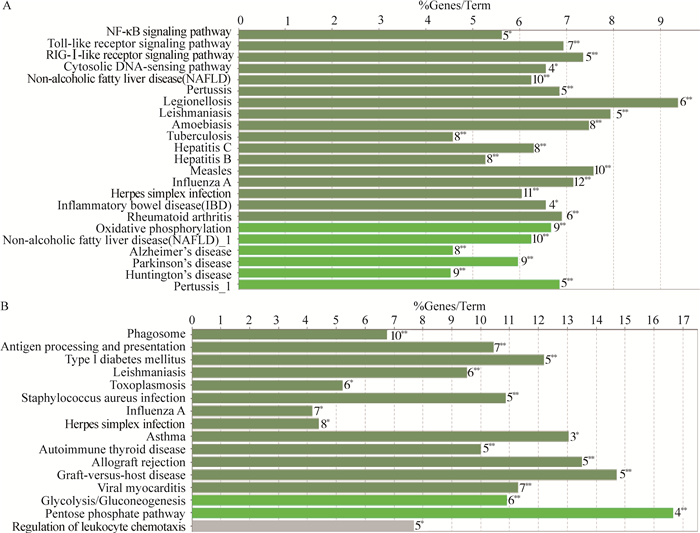
为了明确这些差异表达蛋白质的生物学功能及是否在信号通路上发挥主要作用,笔者使用ClueGo v 2.1.7进行KEGG pathway的富集,在上调蛋白质中,KEGG pathway显著富集到的Term有Toll-like receptor signaling pathway (P=7.25×10-4)、NF-κB signaling pathway (P=1.9×10-2)、RIG-I-like receptor signaling pathway(P=0.63×10-3)等(图 1A)。在下调蛋白质中,Antigen processing and presentation signaling pathway(P=1.82×10-4)等被显著富集(图 1B)。
|
A.上调蛋白质富集的通路;B.下调蛋白质富集的通路。相同颜色的条状图代表蛋白质富集到了相同的功能分组(**. P<0.01; *. P<0.05) A. Enriched GO KEGG pathway terms of upregulate proteins; B. Enriched GO KEGG pathway terms of down regulated proteins. The same color represents terms belonging to the same functional group (**. P < 0.01; *. P < 0.05) 图 1 ClueGO对PRRSV感染PAMs后显著差异蛋白质的信号通路富集 Figure 1 GO KEGG pathway terms of the differentially expressed proteins in PRRSV-infected PAMs by ClueGO |
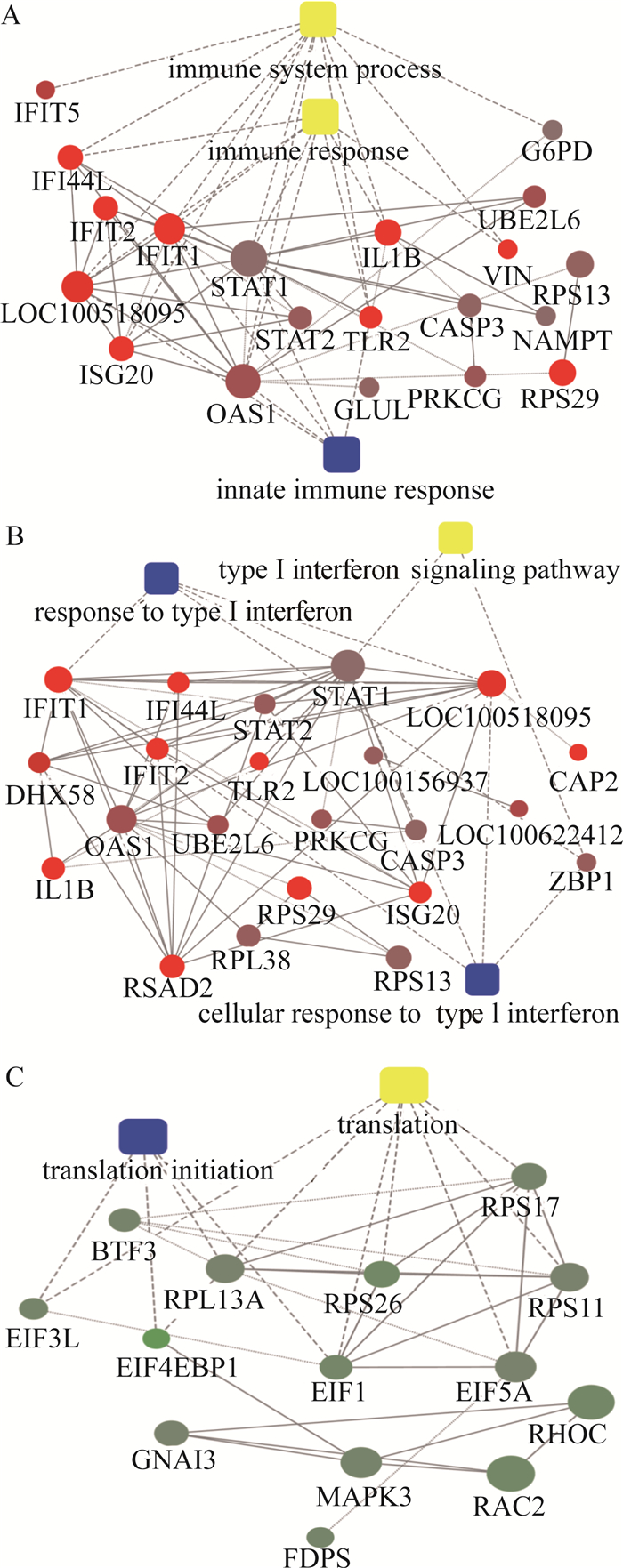
笔者使用OMICSBEAN在线分析软件(网址:http://www.omicsbean.cn/)对PRRSV BJ-4感染PAMs后显著差异表达蛋白质进行蛋白质互作网络分析,图 2列出了3个与我们研究密切相关的蛋白质互作网络图。图 2A是与免疫反应相关的蛋白质互作网络;图 2B是与Ⅰ型干扰素信号通路相关的蛋白质互作网络;图 2C是与翻译相关的蛋白质互作网络。图 2A、B中显示的是感染后上调表达蛋白质,用红色表示,图 2C显示的是感染后下调表达蛋白质,用绿色表示,颜色的深浅表示上调或者下调的程度。
|
图A、B为上调蛋白质的互作信息; 图C为下调表达蛋白质的互作信息。数据使用在线分析网站http://www.omicsbean.cn分析。上调蛋白质和下调蛋白质分别用红色和绿色来表示。方块代表了不同生物学进程中不同的Term, 圆形代表了不同的蛋白质。颜色深浅代表了上调和下调的程度。实线代表了已经证实的相互作用,虚线代表了预测的相互作用 The online analysis software http://www.omicsbean.cn was used to analysis the data. Fig 2 A, B was protein-protein interaction network of upregulated proteins; Fig 2C was protein-protein interaction network of downregulated proteins. The upregulated proteins are in red color. The downregulated proteins are in green color. The color depth indicates the magnitude of the change in protein expression. The square shape are indicative of terms in biological process, the round shape are indicative of proteins. The full lines indicate the molecular relationships had already been reported. The imaginary line indicated the molecular relationships were predicted 图 2 PRRSV感染PAMs后上调和下调蛋白质的互作信息 Figure 2 Specific network analysis of proteins significantly changed in PRRSV-infected PAMs |
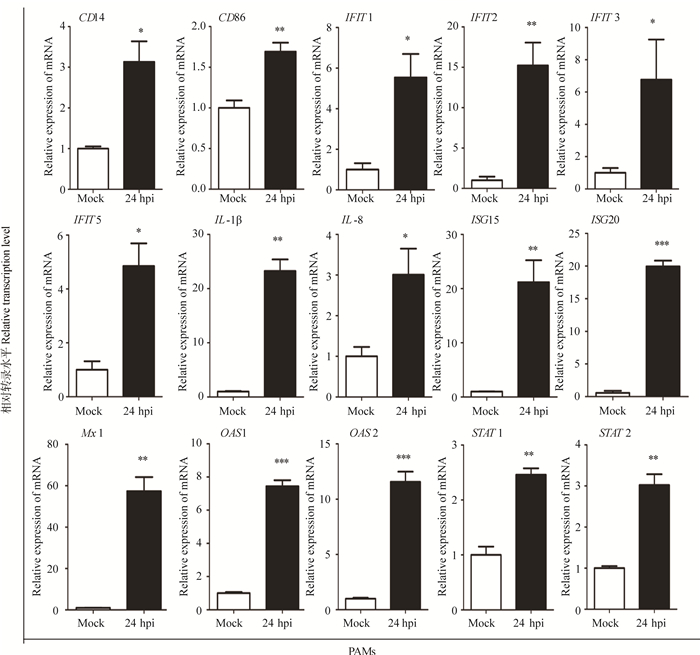
选取Label-free质谱鉴定部分差异表达的蛋白质进行Real-time PCR分析,这些差异蛋白质与炎症、天然免疫及Ⅰ型干扰素介导的信号通路密切相关。结果如图 3所示,与未感染的细胞对照相比,PRRSV感染PAMs 24 h后,CD14、CD86、IL-1β、IL-8,9个干扰素刺激基因[(interferon-stimulated gene,ISGs),包括ISG15、ISG20、interferon-induced protein with tetratricopeptiderepeats 1 (IFIT1)、IFIT2、IFIT3、IFIT5、Mx1、2′-5′-oligo adenylate synthetase 1 (OAS1) 和OAS2],以及信号转导及转录激活因子(the signal transducers and activators of transcription,STATs)STAT1和STAT2,这些基因的转录水平在PRRSV BJ-4株感染后显著上调,与质谱鉴定结果相一致。
|
PAMs用MOI=1的PRRSV感染,同时用未感染的PAMs作为空白对照. PRRSV感染24 h后同时收集两种样品,用荧光定量PCR分析CD14、CD86、IFIT1、IFIT2、IFIT3、IFIT5、ISG15、ISG20、OAS1、OAS2、STAT1、STAT2、IL-8、IL-1β和Mx1 mRNA水平的表达情况。*.P<0.05;**.P<0.01;***.P<0.001 PAMs were infected with PRRSV at MOI = 1 while the uninfected PAMs was the control. The cells were both collected at 24 hpi for real-time PCR detection to analyze the relative expression of CD14, CD86, IFIT1, IFIT2, IFIT3, IFIT5, ISG15, ISG20, OAS1, OAS2, STAT1, STAT2, IL-8, IL-1β and Mx1 mRNAs.*.P < 0.05;**.P < 0.01;***.P < 0.001 图 3 用荧光定量PCR对未感染PAMs及PRRSV感染的PAMs部分差异表达蛋白质进行转录水平分析 Figure 3 Real-time PCR analysis of the differential expression proteins in PRRSV-infected PAMs and uninfected PAMs |
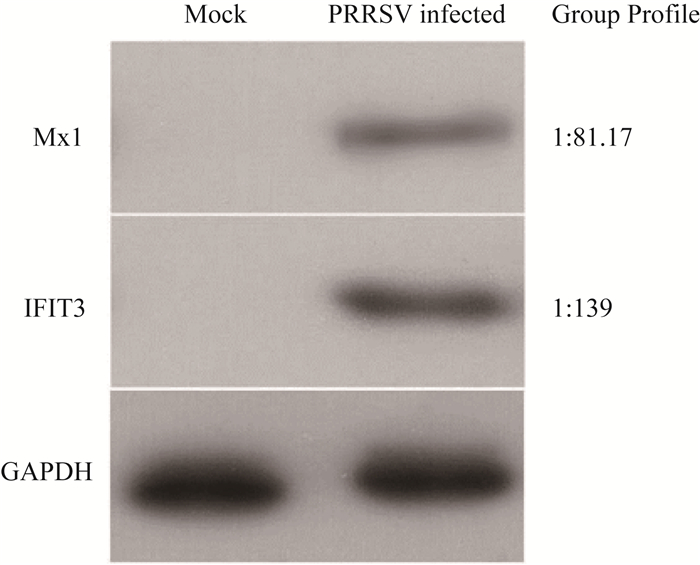
为了进一步证实LC-MS/MS数据的可靠性,对2个蛋白质Mx1和IFIT3进行Western blot分析。如图 4所示,这2个蛋白质的表达变化与LC-MS/MS结果一致。
|
免疫印迹验证PRRSV感染PAMs后IFIT3、Mx1两个蛋白质的表达情况,以GAPDH为内参。Label-free检测的比值在右侧。PAM细胞用MOI=1的PRRSV病毒感染,同时设置未感染的细胞作为对照,病毒感染后24 h收样 Immunoblotting analysis of GAPDH, IFIT3, Mx1 in PRRSV-infected PAMs and Uninfected PAMs. Label-free ratios (infection/control) are shown on the right side. PAMs were infected with PRRSV at MOI=1. Both infected and uninfected PAMs were collected at 24 hpi for immunoblotting analysis 图 4 免疫印迹验证PRRSV感染PAMs后差异表达蛋白质 Figure 4 Validation of differentially expressed proteins with immunoblotting analysis |
定量蛋白质组学技术广泛用于研究病毒和宿主的相互作用,本研究中笔者利用Label Free LC-MS/MS鉴定了PRRSV感染PAM细胞过程中差异表达的蛋白质。分别在空白对照和感染后24 h样品中检测到了3 794和3 577个蛋白质及29 854和25 597个肽段。430个蛋白质在PRRSV BJ-4株感染PAM细胞后显著差异表达。其中的两个蛋白质IFIT3、Mx1用免疫印迹技术证实在PRRSV BJ-4株感染PAM细胞后上调表达,这与Label-free LC-MS/MS鉴定结果一致。由于缺乏特异性抗体,笔者对12个基因用荧光定量PCR检测其mRNA转录水平,结果证实转录水平的变化趋势与Label-free LC-MS/MS鉴定的蛋白质水平表达趋势一致,证实我们本次采用的Label-free LC-MS/MS方法检测到的蛋白质表达差异结果可靠。
NF-κB蛋白家族包括的五个成员分别是NF-κB、IκB、RelA/p65、c-Rel和RelB,它们控制着天然免疫和炎症等诸多生物学进程。在静息状态下,NF-κB蛋白二聚体和抑制因子IκB以三聚体的形式稳定存在于细胞质中,当细胞受到外界信号(如LPS、病原菌、促炎因子等)的刺激后,在IκB激酶IKK蛋白的促进下IκB蛋白发生磷酸化并降解,NF-κB蛋白的亚基被释放,转移入核激活下游炎症信号通路,这是经典的NF-κB活化途径[20]。本研究中,我们鉴定出NF-κB1 p50/p105、NF-κB2 p52/p100和CD14三个蛋白在PRRSV BJ-4株感染后显著上调(表 2),说明NF-κB信号通路在病毒感染后被显著激活。下游的核内转录因子AP-1在PRRSV感染后变化显著,细胞因子IL-1β和趋化因子IL-8蛋白表达量在病毒感染后显著上调(表 2),推测在AP-1活化后启动了下游炎症相关的靶基因的转录。
在PRRSV BJ-4株感染PAM细胞显著上调表达的171个蛋白质中,有9个是ISGs,包括:ISG15、ISG20、interferon-induced protein with tetratricopeptiderepeats 1 (IFIT1)、IFIT2、IFIT3、IFIT5、Mx1、2′-5′-oligo adenylate synthetase 1 (OAS1) 和OAS2。此外,干扰素信号通路相关的STAT1、STAT2及Mx1基因在病毒感染后也显著上调。Ⅰ型干扰素(IFN-α和IFN-β)和IFN-γ一样对抗病毒天然免疫十分关键,对获得性免疫的调节也十分重要。激活Ⅰ型干扰素信号通路,能够介导宿主的抗病毒效应。研究表明ISG基因的表达是由干扰素通过JAK-STAT信号通路所介导:Ⅰ型干扰素信号通路的起始发生于IFN-α和IFN-β结合到细胞表面干扰素受体IFNAR上,进而激活与干扰素受体IFNAR相连接的Janus kinase (JAK)和TYK2,招募STAT1、STAT2蛋白并使其发生磷酸化。磷酸化的STAT1、STAT2蛋白再和interferon regulatory factor 9 (IRF9) 形成异源三聚体干扰素刺激因子3(ISGF3),转移至细胞核内引发ISG相关基因的转录,从而起到抗病毒作用[21]。因此,STAT蛋白的磷酸化和核转位是激活经典JAK-STAT信号通路的两大特征。PRRSV是否能够诱导干扰素的产生和抗病毒效应一直存在争议。2010年,低致病性PRRS疫苗株MLV被证实感染PAMs能够激活干扰素刺激基因的转录及抗病毒蛋白的表达[22]。S.Genini等报道了PRRSV能够诱导PAM细胞产生IFN-β[23]。但也有大量报道证实PRRSV抑制了Ⅰ型干扰素(IFN-α和IFN-β)和IFN-γ的表达和先天免疫信号通路。本研究中鉴定出的9个ISG相关蛋白及STAT1、Mx1蛋白在PRRSV感染后mRNA水平显著上调,Western blot验证Mx1和IFIT3在PRRSV感染后也显著上调表达,与LC-MS/MS鉴定结果一致,这些与JAK-STAT通路被激活的特征相符合,推测PRRSV经典毒株激活了JAK-STAT天然免疫信号通路,但PRRSV感染后是否介导了STAT蛋白的磷酸化和核转位需要进一步验证。
当有凋亡信号刺激时,细胞色素C就会从线粒体中释放出来,细胞色素C绑定到Apaf-1单体上,促进了Apaf-1的延伸,激活的Apaf-1单体装配形成了异源的凋亡体。Caspase-9在凋亡体装配前被招募并绑定到Apaf-1上,使其催化位点激活。接着,活化的Caspase-9分裂并激活Caspase-3二聚体。Caspase-3进而介导了下游分子效应,使肌动蛋白等细胞组分水解,而导致细胞的死亡[24]。本研究中PAM细胞在感染PRRSV BJ-4毒株后细胞色素C与Caspase-3蛋白表达水平较空白对照组显著升高(表 2),推测PRRSV感染PAM细胞后由Caspase-9介导的细胞凋亡信号通路被激活,引起了细胞组分的水解和细胞的凋亡。
与跨细胞膜相关的细胞骨架蛋白的改变与病毒的增殖密切相关。比如:微丝和微管相关蛋白,在传染性法氏囊病毒和非典型性肺炎病毒感染后显著变化[13]。大部分细胞骨架相关蛋白在PRRSV BJ-4株感染后下调表达(表 3),这与之前已经报道过的IBDV及PCV2感染后细胞骨架蛋白变化趋势一致[11]。在本研究中,septin-6、WAS/WASL-interacting protein family member 1两个蛋白与肌动蛋白的组成和装配有关,NDRG1、Dynein heavy/light chain、Dynamin-1、Dynamin-2与微管的产生、装配和调节有关。Golgi apparatus protein 1、Golgin subfamily B member 1参与细胞中高尔基体的形成。这些蛋白在PRRSV感染PAMs后显著下调表达,我们推测宿主细胞中的微丝、微管等细胞骨架网络结构在病毒感染后分散和崩解导致了不稳定的细胞骨架结构,从而促进了病毒从感染细胞中的释放。Zinc finger FYVE domain-containing protein 26参与同源重组,与DNA的损伤后修复有关,它的下调表达说明病毒感染细胞后抑制了细胞的正常分裂和生长机制。
在病毒感染后,一些与氧化还原反应相关的蛋白质发生了改变,比如superoxide dismutase [Mn] (SOD)蛋白在PRRSV感染后显著上调(表 2)。SOD是铁/锰超氧化物歧化酶的家族成员,它能结合到氧化磷酸化后过氧化物的副产物上,并使他们转化为过氧化氢和两个氧原子,以避免氧化引起的细胞凋亡。病毒感染介导的氧化应激与吞噬作用的激活和反应氧的释放有关,这种氧化应激正向激活了免疫系统并消除了由病毒感染所引起的细胞损伤。此外,需要注意的是Hsp70 (heat shock protein 70) 和heat shock protein beta-1在PRRSV感染PAMs后显著下调。热休克蛋白的激活是病毒感染过程中的一个特殊功能,它保证病毒粒子的正常装配,对病毒的复制、增殖起重要作用。Hsp70已证实与PRRSV的Nsp12蛋白有相互作用,在病毒感染的过程中Hsp70能够被Nsp12招募,保证了病毒在细胞上的稳定复制和增殖[25]。PRRSV CH-1a株在体外感染Mac-145细胞时,Hsp70及Hsp90的表达增加对病毒的复制有时间依赖性,并能够通过抑制Hsp70蛋白或Hsp90的表达来减少病毒的复制和增殖[26]。Hsp27蛋白在细胞感染H9N2亚型禽流感病毒[27]、非洲猪瘟病毒[15]后显著上调,但在经典猪瘟病毒[28]和PCV2感染后显著下调[29]。本研究中两个热休克蛋白在PRRSV BJ-4株感染PAMs后24 h显著下调表达,其作用机制需要进一步探讨。
MHC分子能够介导TLR所引起的天然免疫炎症相反的信号通路,研究发现MHCⅠ类分子能够降低TLR所触发的天然免疫炎症反应,在TLR激活后MHCⅠ分子发生磷酸化,从而导致Fps活性增强及招募磷酸化的SHP-2,干扰了由TRAF6介导的TLR信号通路[30]。猪MHCⅠ类分子由轻链(β2M)和重链(SLA-IHC)组成,研究发现HP-PRRSV感染后,β2M和SLA-IHC表达量均降低,非结构蛋白NSP4能够抑制β2M的表达,从而影响MHC Ⅰ类分子的形成[31]。本研究中MHC Ⅰ类分子与MHC Ⅱ类分子在PRRSV感染PAM细胞后表达量均降低,与前人研究结果一致。
4 结论经典PRRSV美洲株感染PAM细胞后共鉴定出差异显著蛋白质430个,其中有171个上调蛋白质和259个下调蛋白质,这些蛋白质与细胞抗病毒天然免疫及抗原递呈等相关。许多上调表达蛋白质都富集在与天然免疫相关的NF-κB、MAPK及RIG-Ⅰ等信号通路上;下调蛋白质主要富集在MHCⅠ和MHCⅡ分子相关信号通路上。这为我们深入探索PRRSV的感染机制及提出新的抗病毒策略提供理论依据。
| [1] | COLLINS J E, BENFIELD D A, CHRISTIANSON W T, et al. Isolation of swine infertility and respiratory syndrome virus (isolate ATCC VR-2332) in North America and experimental reproduction of the disease in gnotobiotic pigs[J]. J Vet Diagn Invest, 1992, 4(2): 117–126. DOI: 10.1177/104063879200400201 |
| [2] | NEUMANN E J, KLIEBENSTEIN J B, JOHNSON C D, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States[J]. J Am Vet Med Assoc, 2005, 227(3): 385–392. DOI: 10.2460/javma.2005.227.issue-3 |
| [3] | CAVANAGH D, NIDOVIRALES. Nidovirales:a new order comprising Coronaviridae and Arteriviridae[J]. Arch Virol, 1997, 142(3): 629–633. |
| [4] | SNIJDER E J, MEULENBERG J J. The molecular biology of arteriviruses[J]. J Gen Virol, 1998, 79(5): 961–979. DOI: 10.1099/0022-1317-79-5-961 |
| [5] | ALLENDE R, LEWIS T L, LU Z, et al. North American and European porcine reproductive and respiratory syndrome viruses differ in non-structural protein coding regions[J]. J Gen Virol, 1999, 80(2): 307–315. DOI: 10.1099/0022-1317-80-2-307 |
| [6] | FANG Y, SNIJDER E J. The PRRSV replicase:exploring the multifunctionality of an intriguing set of nonstructural proteins[J]. Virus Res, 2010, 154(1-2): 61–76. DOI: 10.1016/j.virusres.2010.07.030 |
| [7] | FANG Y, FANG L R, WANG Y, et al. Porcine reproductive and respiratory syndrome virus nonstructural protein 2 contributes to NF-κB activation[J]. Virol J, 2012, 9(1): 83. DOI: 10.1186/1743-422X-9-83 |
| [8] | FIRTH A E, ZEVENHOVEN-DOBBE J C, WILLS N M, et al. Discovery of a small arterivirus gene that overlaps the GP5 coding sequence and is important for virus production[J]. J Gen Virol, 2011, 92(5): 1097–1106. DOI: 10.1099/vir.0.029264-0 |
| [9] | EMMOTT E, WISE H, LOUCAIDES E M, et al. Quantitative proteomics using SILAC coupled to LC-MS/MS reveals changes in the nucleolar proteome in influenza A virus-infected cells[J]. J Proteome Res, 2010, 9(10): 5335–5345. DOI: 10.1021/pr100593g |
| [10] | MULLER U, STEINHOFF U, REIS L F, et al. Functional role of type Ⅰ and type Ⅱ interferons in antiviral defense[J]. Science, 1994, 264(5167): 1918–1921. DOI: 10.1126/science.8009221 |
| [11] | FAN H Y, YE Y, LUO Y W, et al. Quantitative proteomics using stable isotope labeling with amino acids in cell culture reveals protein and pathway regulation in porcine circovirus type 2 infected PK-15 cells[J]. J Proteome Res, 2012, 11(2): 995–1008. DOI: 10.1021/pr200755d |
| [12] | YE Y, YAN G R, LUO Y W, et al. Quantitative proteomics by amino acid labeling in foot-and-mouth disease virus (FMDV)-infected cells[J]. J Proteome Re s, 2013, 12(1): 363–377. DOI: 10.1021/pr300611e |
| [13] | ZHENG X J, HONG L L, SHI L X, et al. Proteomics analysis of host cells infected with infectious bursal disease virus[J]. Mol Cell Proteom, 2008, 7(3): 612–625. DOI: 10.1074/mcp.M700396-MCP200 |
| [14] | NARAYAN R, GANGADHARAN B, HANTZ O, et al. Proteomic analysis of HepaRG cells:a novel cell line that supports hepatitis B virus infection[J]. J Proteome Res, 2009, 8(1): 118–122. DOI: 10.1021/pr800562j |
| [15] | ALFONSO P, RIVERA J, HERNÁEZ B, et al. Identification of cellular proteins modified in response to African swine fever virus infection by proteomics[J]. Proteomics, 2004, 4(7): 2037–2046. DOI: 10.1002/(ISSN)1615-9861 |
| [16] | ZHANG H M, GUO X, GE X N, et al. Changes in the cellular proteins of pulmonary alveolar macrophage infected with porcine reproductive and respiratory syndrome virus by proteomics analysis[J]. J Proteome Res, 2009, 8(6): 3091–3097. DOI: 10.1021/pr900002f |
| [17] | XIAO S Q, WANG Q W, JIA J Y, et al. Proteome changes of lungs artificially infected with H-PRRSV and N-PRRSV by two-dimensional fluorescence difference gel electrophoresis[J]. Virol J, 2010, 7(1): 107. DOI: 10.1186/1743-422X-7-107 |
| [18] | LU Q, BAI J, ZHANG L L, et al. Two-dimensional liquid chromatography-tandem mass spectrometry coupled with isobaric tags for relative and absolute quantification (iTRAQ) labeling approach revealed first proteome profiles of pulmonary alveolar macrophages infected with porcine reproductive and respiratory syndrome virus[J]. J Proteome Res, 2012, 11(5): 2890–2903. DOI: 10.1021/pr201266z |
| [19] | LUO R, FANG L R, JIN H, et al. Label-free quantitative phosphoproteomic analysis reveals differentially regulated proteins and pathway in PRRSV-infected pulmonary alveolar macrophages[J]. J Proteome Res, 2014, 13(3): 1270–1280. DOI: 10.1021/pr400852d |
| [20] | HAYDEN M S, GHOSH S. Shared principles in NF-κB signaling[J]. Cell, 2008, 132(3): 344–362. DOI: 10.1016/j.cell.2008.01.020 |
| [21] | PLATANIAS L C. Mechanisms of type-Ⅰ-and type-Ⅱ-interferon-mediated signalling[J]. Nat Rev Immunol, 2005, 5(5): 375–386. DOI: 10.1038/nri1604 |
| [22] | PATEL D, NAN Y C, SHEN M Y, et al. Porcine reproductive and respiratory syndrome virus inhibits type Ⅰ interferon signaling by blocking STAT1/STAT2 nuclear translocation[J]. J Virol, 2010, 84(21): 11045–11055. DOI: 10.1128/JVI.00655-10 |
| [23] | GENINI S, DELPUTTE P L, MALINVERNI R, et al. Genome-wide transcriptional response of primary alveolar macrophages following infection with porcine reproductive and respiratory syndrome virus[J]. J Gen Virol, 2008, 89(10): 2550–2564. DOI: 10.1099/vir.0.2008/003244-0 |
| [24] | BRATTON S B, SALVESEN G S. Regulation of the Apaf-1-caspase-9 apoptosome[J]. J Cell Sci, 2010, 123(19): 3209–3214. DOI: 10.1242/jcs.073643 |
| [25] | DONG S, LIU L, WU W N, et al. Determination of the interactome of non-structural protein12 from highly pathogenic porcine reproductive and respiratory syndrome virus with host cellular proteins using high throughput proteomics and identification of HSP70 as a cellular factor for virus replication[J]. J Proteom, 2016, 146: 58–69. DOI: 10.1016/j.jprot.2016.06.019 |
| [26] | GAO J T, XIAO S Q, LIU X H, et al. Inhibition of HSP90 attenuates porcine reproductive and respiratory syndrome virus production in vitro[J]. Virol J, 2014, 11(1): 17. DOI: 10.1186/1743-422X-11-17 |
| [27] | LIU N, SONG W J, WANG P, et al. Proteomics analysis of differential expression of cellular proteins in response to avian H9N2 virus infection in human cells[J]. Proteomics, 2008, 8(9): 1851–1858. DOI: 10.1002/(ISSN)1615-9861 |
| [28] | SUN J, JIANG Y, SHI Z X, et al. Proteomic alteration of PK-15 cells after infection by classical swine fever virus[J]. J Proteome Res, 2008, 7(12): 5263–5269. DOI: 10.1021/pr800546m |
| [29] | ZHANG X, ZHOU J Y, WU Y P, et al. Differential proteome analysis of host cells infected with porcine circovirus type 2[J]. J Proteome Res, 2009, 8(11): 5111–5119. DOI: 10.1021/pr900488q |
| [30] | XU S, LIU X G, BAO Y, et al. Constitutive MHC class Ⅰ molecules negatively regulate TLR-triggered inflammatory responses via the Fps-SHP-2 pathway[J]. Nat Immunol, 2012, 13(6): 551–559. DOI: 10.1038/ni.2283 |
| [31] | QI P F, LIU K, WEI J C, et al. Non-structural protein 4 of porcine reproductive and respiratory syndrome virus modulates cell surface swine leukocyte antigen class i expression by down-regulating β2-microglobulin transcription[J]. J Virol, 2017, 91(5): e01755–16. |



