哺乳动物早期胚胎在体外培养的过程中受多种环境因素的影响,如温度、氧气浓度、CO2浓度等[1]。其中氧气浓度尤为重要,胚胎在体内发育和体外培养过程中氧气浓度存在较大的差异,输卵管中氧气浓度约为大气氧浓度的1/4~1/3[2-3]。降低培养箱中的氧气浓度能够提高小鼠[4]、山羊[5]和牛[6]等早期胚胎的体外发育能力。在体外培养的过程中降低氧气的浓度,有利于减少过量的活性氧自由基(Reactive oxygen species,ROS)。ROS作为细胞内的第二信使,对胚胎的发育和凋亡至关重要。适量的ROS能够促进胚胎的发育,但是体外培养过程中积累的过量ROS对胚胎发育造成损伤,降低胚胎发育率。在体外添加抗氧化剂,如谷胱甘肽(Glutathione,GSH),能显著促进牛[7-8]、猪[9]、小鼠[10]、山羊[11]等胚胎的发育能力,GSH对牛和山羊早期胚胎发育的促进作用主要发生在8~16-细胞期,这与本实验室的前期研究结果一致[8]。
随着转录组学研究的深入和高通量测序技术的广泛应用,大量的长链非编码RNA(Long non-coding RNA,lncRNA)被发现和挖掘。lncRNA是一类长度>200个核苷酸,保守性较差,且具有组织和细胞特异性的RNA分子。据估计,17%的lncRNAs位于细胞核,4%则在细胞质中富集[12]。lncRNAs能够在转录水平、转录后水平和表观遗传水平调控基因表达[13]。尽管lncRNA已经成为研究的热点,但常规RNA-seq至少需要103个细胞才能提供足量的RNA进行测序,而早期胚胎细胞数量较少,需要大量的胚胎才能满足测序对RNA含量的要求。关于早期胚胎中lncRNA的研究较少。单细胞测序技术的出现,使得在转录组水平上分析早期胚胎中lncRNA的表达谱成为可能。lncRNA对早期胚胎的发育至关重要,L.Y.Yan等[14]发现人类的植入前胚胎中存在2 700多种全新的lncRNAs,其表达具有发育阶段特异性,参与胚胎发育过程中的细胞命运决定。J. Durruthy-Durruthy等[15]对人干细胞多能性相关转录本(Human pluripotency-associated transcripts,HPAT)2、3和5(HPAT2、HPAT3和HPAT5) 在2-细胞期胚胎中进行干扰,结果胚胎发育到囊胚期时内细胞团中不表达八聚体结合转录因子4(Octamer-binding transcription factor 4,OCT4) 和性别决定基因相关转录因子2(Sex determining region Y box 2,SOX2),表明该lncRNA可能有助于内细胞团的形成和多能性的维持,首次证明了lncRNA对人体内胚胎的发育具有重要影响。N.Hamazaki等[16]对小鼠MII期卵母细胞和2-细胞期胚胎测序获得1 000多个lncRNAs,干扰其中1条的表达后胚胎死亡,可见其对胚胎的发育至关重要。
针对家畜良种快速扩繁的实际需求及其胚胎体外生产效率低的现状,本研究以牛体外受精胚胎为研究对象,在体外培养的过程中添加GSH,收集8~16-细胞期胚胎和桑椹胚,进行单细胞转录组测序分析,阐明GSH对早期胚胎的抗氧化作用机理,揭示lncRNA在牛早期胚胎发育中的功能。
1 材料与方法 1.1 主要试剂所用试剂,如无特殊说明均购自西格玛奥德里奇(上海)贸易有限公司。细胞裂解试剂盒SMARTer® UltraTM Low RNA Kit for Illumina® Sequencing及cDNA扩增试剂盒Advantage® 2 PCR kit购自美国Clontech公司,cDNA纯化试剂盒AMPure XP beads购自美国Beckman Coulter公司,荧光定量PCR试剂盒Power SYBR® Green Cells-to-CtTM Kit购自美国Life Technologies公司。
1.2 牛卵母细胞的采集、体外受精和胚胎的体外培养牛卵巢采集于当地屠宰场,置于37 ℃生理盐水中,3 h内运回实验室。用含有100 IU·mL-1青霉素和100 mg·mL-1链霉素的生理盐水洗卵巢3~5次,抽取直径为2~6 mm的卵泡中的卵母细胞。在体式显微镜下,挑选有3层以上卵丘细胞紧密包围的卵丘-卵母细胞复合体(Cumulus-oocyte complexes,COCs),在成熟培养液(TCM 199培养液、10% FBS、1 μg·mL-1 E2、0.01 IU·mL-1 FSH、10 IU·L-1 LH、10 μg·mL-1肝素、100 IU·mL-1青霉素、100 mg·mL-1链霉素)洗涤后,放入含750 μL成熟液的四孔板中,每孔50枚COCs,在38.5 ℃、5% CO2和100%湿度的CO2培养箱中培养22~24 h。
将COCs从成熟培养液中移入50 μL受精液滴(Brackett-Oliphant培养液[17]、4 mg·mL-1 BSA)中,每滴15枚。37.5 ℃水浴解冻精液,用6 mL洗精液(Brackett-Oliphant培养液、4 mg·mL-1 BSA、10 mol·L-1咖啡因)进行稀释,500 g离心8 min, 洗涤2次;用洗精液调整精子密度约为2×107个·mL-1。取50 μL精液加入含有COCs受精滴中,在38.5 ℃、5% CO2和100%湿度的CO2培养箱中孵育8 h。
用吸卵针脱去外周的卵丘细胞和黏附的精子,将受精卵在前期培养液(26.20 mol·L-1 NaHCO3、109.50 mol·L-1 NaCl、3.10 mol·L-1 KCl、0.80 mol·L-1 MgCl2·6H2O、1.19 mol·L-1 KH2PO4、0.40 mol·L-1丙酮钠、1% NEAA、6 mg·mL-1 BSA、1.50 mol·L-1葡萄糖、5 mol·L-1半乳糖酸钙、2% EAA、1 mol·L-1 L-谷氨酰胺)中洗3次后,放入100 μL的前期培养液中培养。48 h后,将胚胎移入后期培养液(109.50 mol·L-1 NaCl、3.10 mol·L-1KCl、26.20 mol·L-1NaHCO3、0.80 mol·L-1 MgCl2·6H2O、1.19 mol·L-1KH2PO4、0.40 mol·L-1丙酮钠、1.50 mol·L-1葡萄糖、5 mol·L-1半乳糖酸钙、2% EAA、1% NEAA、10% FBS、1 mol·L-1 L-谷氨酰胺)中培养,每隔48 h半量更换培养液。在受精后48和132 h分别收取8~16-细胞期胚胎和桑椹胚,用于转录组测序和荧光定量PCR检测。处理组胚胎的前期和后期培养液中均添加3 mmol·L-1 GSH,对照组则不添加GSH。
1.3 cDNA文库的构建和测序以未经GSH处理的8~16-细胞期(C8C16_C)和桑椹期(ML_C)的胚胎为对照组,经3 mmol·L-1 GSH处理的8~16-细胞期(C8C16_T)和桑椹期(ML_T)胚胎为处理组进行测序。每组收集20个胚胎,并做两个生物学重复,共构建8个cDNA文库进行上机测序。
使用SMARTer® UltraTM Low RNA Kit for Illumina® Sequencing试剂盒进行胚胎的裂解和第一条cDNA链的合成,使用AMPure XP beads试剂盒纯化后,用Advantage® 2 PCR kit试剂盒扩增第一条cDNA链,并再次纯化,最终得到双链cDNA。Qubit 2.0定量检测,合格后用Covaris系统超声打断双链cDNA短片段进行末端修复, 加A尾并连接测序接头后,纯化,选择片段大小在200 bp左右的文库进行PCR富集得到最终的cDNA文库。先使用Qubit 2.0进行文库的初步定量,稀释文库至2 ng·μL-1,随后用Agilent 2100对文库的insert size进行检测,符合预期后,使用qPCR方法对文库的有效浓度进行准确定量(文库有效浓度>2 nmol·L-1),以保证文库质量。文库检验合格后,把不同文库按照有效浓度及目标数据量的需求,进行HiSeq/MiSeq测序。
使用Tophat 2将得到的原始测序序列比对到牛参考基因组(版本号:UMD3.1) 上,用Cufflinks软件组装出高质量的转录本,用Cuffcompare将其与已注释的转录本信息进行比较,得到潜在的转录本。然后使用CNCI筛选得到长度>200 bp的潜在的非编码转录本。最后与Pfam数据库进行比对,去除能编码蛋白质的转录本,得到最终的lncRNAs。
1.4 差异表达基因的GO功能注释和KEGG通路分析Gene Ontology(GO)是国际标准化的基因功能分类体系,提供了一套标准词汇来描述生物学功能,对基因的功能进行注释[18]。采用GOseq[19]分析方法对差异表达基因(Differentially expressed genes,DEGs)进行GO注释,筛选P < 0.05的GO注释项目。并用WEGO软件对所有DEGs的GO注释项目进行分类统计。
Kyoto encyclopedia of genes and genomes(KEGG)是分析基因在细胞中的代谢途径(Pathway)的主要公共数据库,尤其适用于分析基因组测序和其他的高通量技术得到大规模的数据[20]。使用KOBAS(2.0) 对DEGs进行KEGG通路分析。
1.5 lncRNA和DEGs的共表达分析及其顺式作用靶基因的预测对每一个差异表达lncRNA(Differentially expressed lncRNA,DElncRNA),计算其与每一个DEG之间的皮尔森相关性系数(Pearson correlation coefficient, PCC),将PCC>0.7,或PCC < -0.7,且P < 0.05定义为相关。并采用Cytoscape 3.2.1软件, 构建lncRNA与其共表达基因的网络图。
在Ensemble数据库中找出距lncRNA上下游300 kb内的mRNA,作为lncRNA潜在的顺式作用靶基因,并进行GO功能注释和KEGG通路分析,进而推测lncRNA的功能。
1.6 lncRNA表达量的荧光定量PCR检测筛选8个lncRNAs(CUFF.33095.2、CUFF.52291.1、CUFF.55358.1、CUFF.152963.1、CUFF.17837.1、CUFF.10166.1、CUFF.42178.2和CUFF.38204.3) 进行荧光定量PCR反应,以GAPDH为内参基因,验证测序结果。引物序列见表 1。将收集的8~16-细胞期和桑椹期的胚胎,使用Power SYBR® Green Cells-to-CtTM Kit试剂盒进行细胞裂解,得到cDNA模板。PCR反应体系为上下游引物(10 μmol·L-1)各0.2 μL,cDNA 1 μL,Rnase-Free Water 3.6 μL,master mix 5 μL。PCR反应条件为95 ℃ 10 min,95 ℃15 s,60 ℃1 min,共40个循环,3个重复。使用7900HT system(Applied Biosystems,美国)进行检测,采用2-ΔΔCt法计算lncRNA的相对表达量。
|
|
表 1 lncRNAs引物信息 Table 1 Primers for lncRNAs |
将荧光定量PCR得到8~16-细胞期和桑椹期胚胎中lncRNA的相对表达量和测序结果进行Fold Change(FC)比较。FC的计算公式:FC=桑椹胚时期lncRNA的相对表达水平/8~16-细胞时期lncRNA的相对表达水平。采用SAS 9.2软件进行单方面分类的方差分析,P < 0.05定义为差异显著。
2 结果 2.1 lncRNA数据的来源、分布和质量分析测序共得到4 273个lncRNAs,其中62%来自于基因间区,11%来源于内含子区,4%来源于外显子区,17%来源于编码区,6%来源于反义链。lncRNA在染色体上均有分布,且没有明显的偏好性,说明其转录是全基因组范围的。同时,对鉴定得到的lncRNA与已知转录本的表达量、长度和外显子个数进行了比较。lncRNA的平均表达值为(FPKM)2.17,而已知转录本的平均表达值为(FPKM)378.925;lncRNA的平均长度为1 368.69 bp,小于已知转录本的1 708.6 bp;lncRNA的平均外显子个数为2.52,小于已知转录本的8.44。这些都与已发表的lncRNA的特征一致[21],说明lncRNA的可靠性。对lncRNA表达量的分析也说明数据重复性较好。
2.2 DElncRNAs与对照组中DEGs的共表达分析根据测序结果,筛选在对照组和处理组中有不同表达模式的lncRNA。共获得59个DElncRNAs,其中23个在对照组上调,在处理组下调(图 1);及36个在对照组下调,而在处理组上调(图 2)。
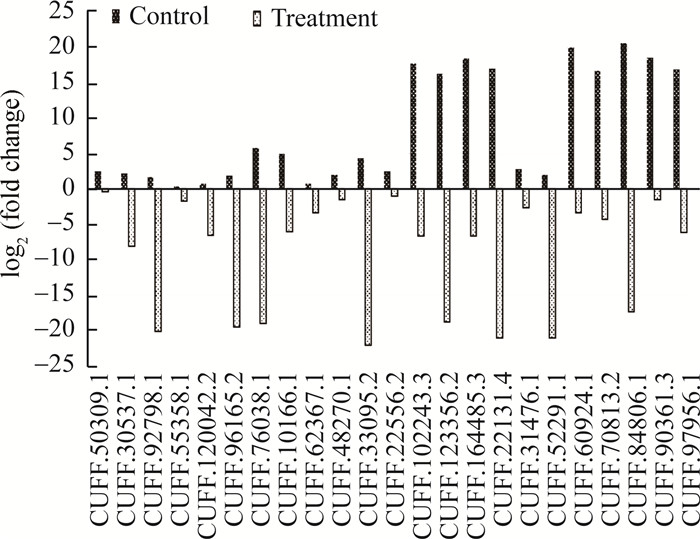
|
图 1 在对照组上调,在处理组下调的23个DElncRNAs Figure 1 Twenty-three DElncRNAs up-regulated in control group and down-regulated in GSH treatment group |
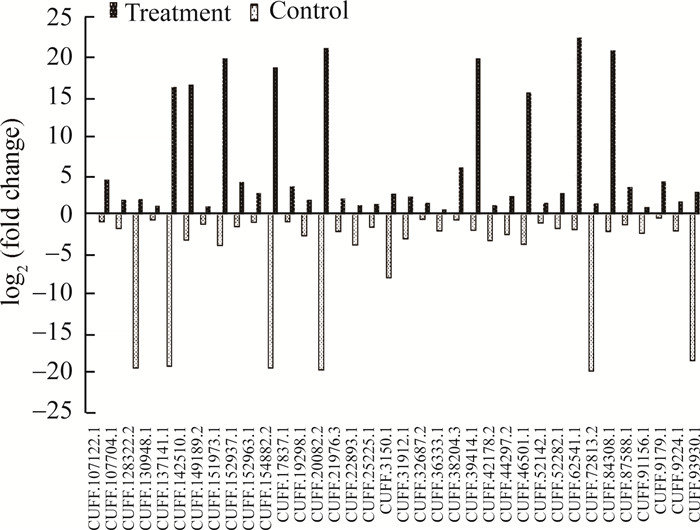
|
图 2 在对照组下调,在处理组上调的36个DElncRNAs Figure 2 Thirty-six DElncRNAs down-regulated in control group and up-regulated in GSH treatment group |
对59个DElncRNAs与对照组DEGs(885个)做共表达分析,并构建互作网络图。
对照组中,有754个DEGs与59个DElncRNAs共表达,对其进行GO注释分析(图 3),发现这些基因参与了47个功能分类。细胞组分分类中富集的条目:细胞、细胞部分、细胞器部分、胞外区部分、突触部分、突触等;生物过程分类中包括细胞组分的生物发生、发育过程、繁殖、死亡、免疫系统过程等;分子功能分类中富集的条目:酶调节剂活性、电子载流子活性、分子转导子活性、转录调节子活性、转运蛋白活性等。其中,细胞、细胞部分、结合、大分子复合物、细胞器、细胞器部分、代谢通路富集基因数较多。KEGG分析结果中,共表达基因主要参与5条代谢通路(图 4):亨廷顿舞蹈症、帕金森氏病、阿尔兹海默氏病、肥大型心肌病、氧化磷酸化。其中,亨廷顿舞蹈症和氧化磷酸化代谢通路上富集的基因较多。
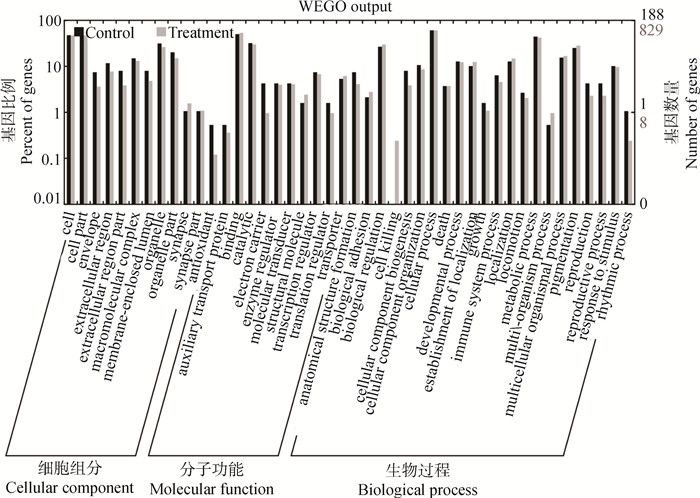
|
图 3 59个lncRNAs与对照组与处理组中共表达DEGs的GO分析 Figure 3 GO analysis of DEGs co-expressed with 59 DElncRNAs in control and GSH treatment groups |
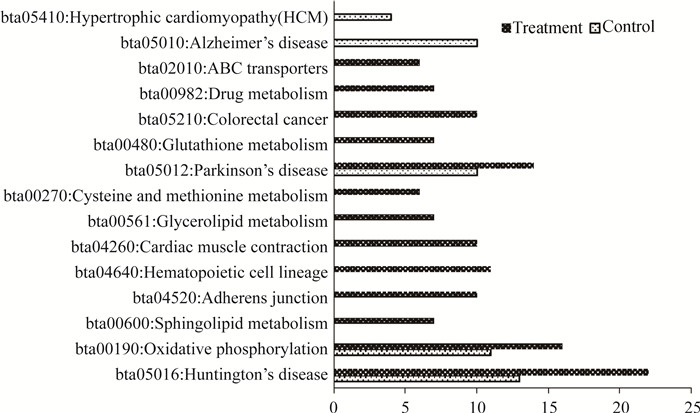
|
图 4 59个DElncRNAs在对照组与处理组中共表达DEGs的KEGG分析 Figure 4 KEGG analysis of DEGs co-expressed with 59 DElncRNAs in control and GSH treatment groups |
lncRNA与共表达DEGs的互作网络图中(图 5),有809个网络节点,6 210个连接;其中1个lncRNA最多和460个DEGs相互作用,1个DEG最多和25个lncRNAs共表达。
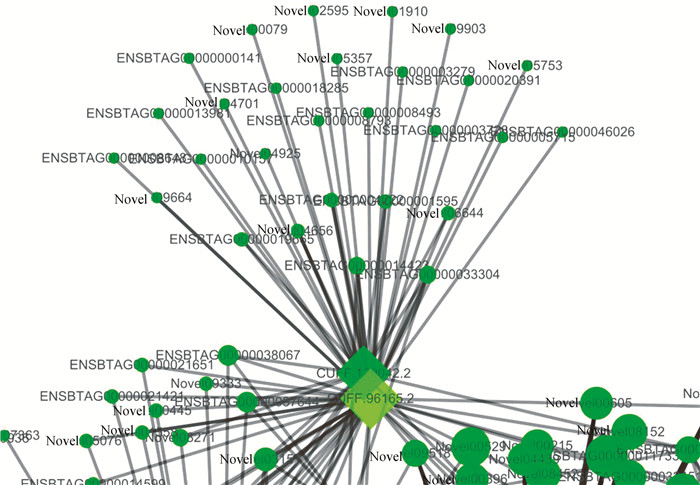
|
图 5 59个DElncRNAs与对照组共表达DEGs的互作网络图 Figure 5 Network of 59 DElncRNAs and co-expressed DEGs in control group |
对59个DElncRNAs与处理组的DEGs(3 952个)进行共表达分析,并构建互作网络图。
处理组中,59个DElncRNAs与2 782个DEGs共表达,对其进行GO注释分析(图 3),这些基因参与了48个功能分类。细胞组分分类中富集的条目:细胞部分、细胞外区、大分子复合物、细胞外区部分、包膜等;生物过程分类中富集的条目:色素沉着、生物黏附、代谢过程、发育过程、生物调节等;分子功能分类中富集的条目:催化活性、抗氧化活性、辅助转运蛋白活性、翻译调节子活性等。其中,细胞成分、细胞器、结合、新陈代谢过程、催化反应、色素形成等条目富集基因较多。KEGG分析结果,共表达DEGs主要参与13条代谢通路(图 4):亨廷顿舞蹈症、药物代谢、谷胱甘肽代谢、帕金森氏病、半胱氨酸和蛋氨酸代谢、氧化磷酸化、黏合连接、心肌收缩、造血细胞系、鞘脂类代谢、甘油酯代谢、结肠癌、ABC转运蛋白。其中帕金森氏病、氧化磷酸化、亨廷顿舞蹈症代谢通路上富集的基因较多。
互作网络图(图 6)中有2 838个网络节点,21 187个连接;其中一个lncRNA最多和2 141个DEGs相互作用,1个DEG最多和34个lncRNAs共表达。
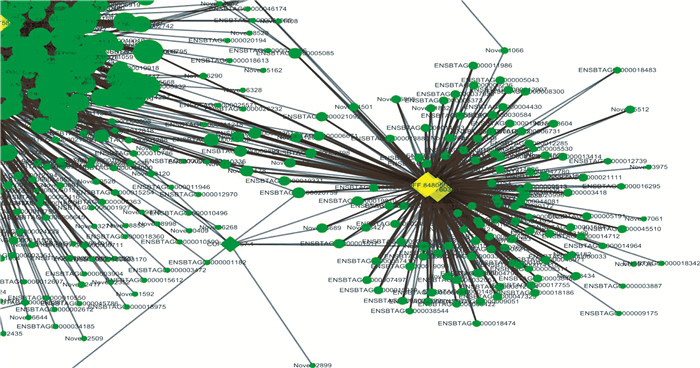
|
图 6 59个DElncRNAs与处理组共表达DEGs的互作网络图 Figure 6 Network of 59 DElncRNAs and co-expressed DEGs in GSH treatment group |
许多lncRNA顺式作用的靶基因是其坐标临近的蛋白编码基因。本研究统计结果表明,约80%的lncRNA距靶基因都在300 kb以内,因此筛选距DElncRNA 300 kb范围内的蛋白编码基因,并对其进行GO功能注释以推测lncRNA的主要功能。结果预测23个DElncRNAs的靶基因有145个蛋白编码基因,共参与35个功能分类,主要包括细胞组分中的胞外区、细胞大分子复合物、细胞器、细胞器部分等;生物过程分类中的发育过程、繁殖、细胞组分组织、死亡、生物调节等;分子功能分类中的分子转导活性、转录调节子活性、酶调节活性、催化活性等(图 7)。36个DElncRNAs的靶基因有154个蛋白编码基因,共参与43个功能分类,主要包括细胞组分中的细胞部分、膜封闭管腔、大分子复合物、细胞器等;生物过程分类中的死亡、繁殖过程、病毒繁殖、色素沉着等;分子功能分类中的结构分子活性、结合、酶调节活性、转运蛋白活性等(图 8)。
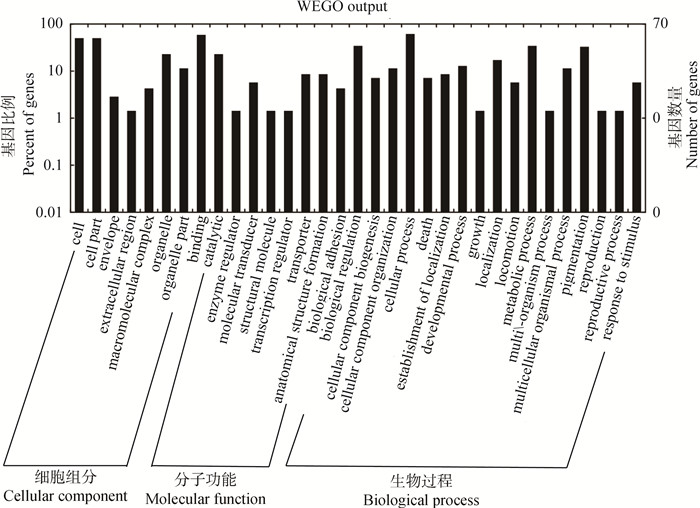
|
图 7 23个DElncRNAs顺式作用靶基因的GO分析 Figure 7 GO analysis of the target genes of 23 DElncRNAs |
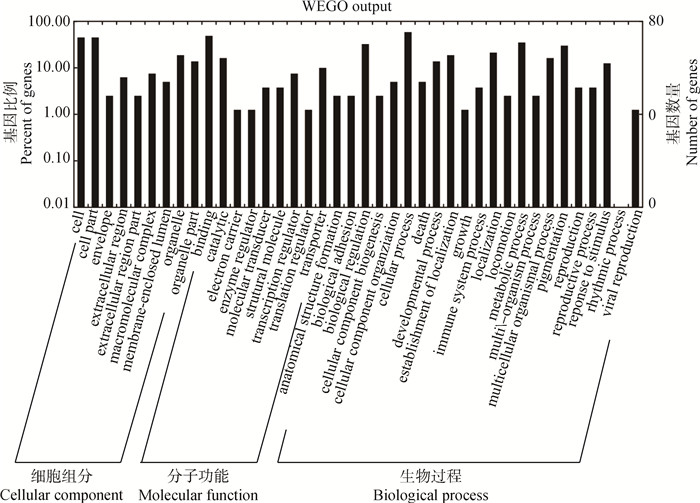
|
图 8 36个DElncRNAs顺式作用靶基因的GO分析 Figure 8 GO analysis of the target genes of 36 DElncRNAs |
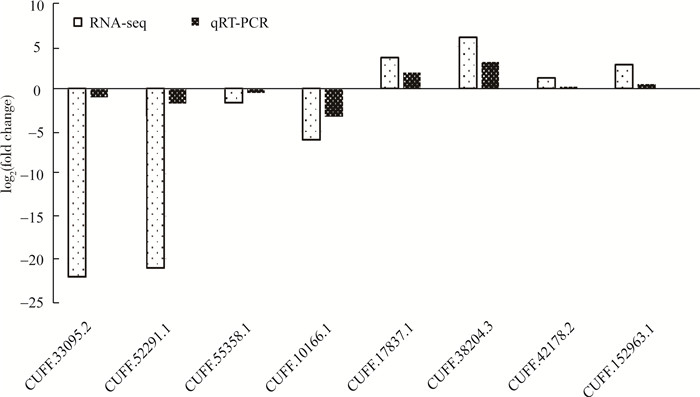
通过lncRNA表达的差异倍数来判断定量和测序结果的一致性。当lncRNA的FC值>或 < 1时,说明lncRNA在荧光定量PCR与测序结果中上调或下调,表达量增加或降低。结果表明,在CUFF.33095.2、CUFF.52291.1、CUFF.55358.1、CUFF.10166.1在荧光定量PCR和测序结果中都下调,CUFF.152963.1、CUFF.17837.1、CUFF.42178.2、CUFF.38204.3则都上调,证明荧光定量PCR检测结果与测序结果是一致的(图 9),表明,转录组测序结果的可靠性。
|
图 9 荧光定量PCR检测结果与转录组测序结果比较 Figure 9 Comparison of the results between RNA-seq and qRT-PCR |
胚胎的发育过程与基因表达控制、蛋白质的合成、翻译后修饰等物理和生化反应相协调,其中氧化还原反应对胚胎的体外发育具有重要的影响。本试验的前期研究表明,在胚胎培养液中添加外源谷胱甘肽极大地促进了牛早期胚胎的发育[8],但其分子机制还不清楚,GSH在转录水平引起的变化也研究较少,因此本研究采用单细胞测序技术对GSH处理的牛体外受精胚胎进行转录组测序,阐明GSH添加后,牛胚胎中lncRNA表达模式的变化,从lncRNA的角度揭示GSH促进牛体外早期胚胎发育的分子机制。
近年来,在小鼠[22]和人[23]的基因组上发现了大量的lncRNAs,且具有多种重要的生物学作用。尽管作为研究哺乳动物胚胎发育的模型,lncRNA在牛胚胎基因组中的功能研究还较少。因此从转录组数据中挖掘潜在lncRNA,对其进行系统的功能分析。已有的研究表明,lncRNA的平均表达量、长度、外显子个数均小于蛋白编码基因[24-26],与本研究结果一致,说明本研究测序结果的可靠性[27]。本试验共预测得到4 273个lncRNAs,从中筛选出具有不同表达模式的59个DElncRNAs,进一步对其进行共表达分析和顺式作用的靶基因预测,以探究其功能。共表达分析获得DEGs进行GO分析,结果同一条GO条目在处理组比对照组中富集的基因个数多,其中细胞杀死条目在处理组DEGs特异富集;二者共同涉及的、与生长发育相关的功能:繁殖过程、细胞组分的生物发生、发育过程、转录调节子活性、酶调节剂活性、催化活性、翻译调节子活性等,说明lncRNA的共表达基因在胚胎发育、转录调控等方面具有重要作用。
共表达基因的KEGG通路分析结果中,处理组和对照组DEGs共同富集的代谢通路有亨廷顿舞蹈症、帕金森氏病、氧化磷酸化;对照组DEGs特异富集的代谢通路是阿尔兹海默氏病和肥大型心肌病;处理组DEGs特异富集的代谢通路:药物代谢、谷胱甘肽代谢、半胱氨酸和蛋氨酸代谢、黏合连接、结肠癌、造血细胞系、鞘脂类代谢、甘油酯代谢等。蛋氨酸通过转硫途径,将硫转移给丝氨酸生成半胱氨酸[28],而半胱氨酸是构成GSH的主要成分,在体内γ-谷氨酰半胱氨酸合成酶催化L-半胱氨酸与L-谷氨酸生成L-谷氨酰半胱氨酸,进一步与甘氨酸在谷胱甘肽合成酶催化作用下生成GSH[29]。当体内半胱氨酸含量供应不足时,GSH分解产生半胱氨酸,以满足生物体生命活动的需要[30]。因此,半胱氨酸和蛋氨酸代谢与GSH代谢密不可分,共同维持机体的氧化还原平衡。越来越多的研究表明,GSH或其类似物对多种疾病的预防和治疗具有显著的促进作用,如亨廷顿舞蹈症[31]、帕金森氏病[32]、阿尔兹海默氏病[33]。本研究发现14个DElncRNAs共表达DEGs参与GSH代谢通路(表 2),说明外源GSH改变DElncRNAs的表达,进而影响胚胎发育相关基因表达,因此推测这些DElncRNAs对胚胎发育至关重要。
|
|
表 2 14个DElncRNAs共表达DEGs的KEGG分析 Table 2 KEGG analysis of co-expressed DEGs with 14 DElncRNAs participating in the glutathione metabolism pathway |
本研究表明,约80% lncRNAs的顺式作用靶基因是其DNA序列上下游300 kb以内的蛋白编码基因,这和已有的研究报道一致[34]。本研究发现lncRNA CUFF.21976.3附近有许多基因,其中质子泵V-ATPase的C2亚基(ATPase H+ transporting V1 subunit C2,ATP6V1C2)、核糖核苷还原酶M2(Ribonucleotide reductase subunit M2,RRM2)、Grainyhead样转录因子1(Grainyhead like transcription factor 1,GRHL1) 和Kruppel样因子(Kruppel-like factor 11,KLF11) 在对照组8~16-细胞期胚胎发育到桑椹胚过程中表达没有差异,但在处理组中ATP6V1C2、RRM2和GRHL1表达显著上调,而KLF11表达显著下调。其中RRM2是核糖核苷还原酶的一个亚基,其合成具有细胞周期依赖性,在S期表达量最高[35],在结肠癌、胃癌等肿瘤的发生和发展过程发挥关键作用[36-37]。且RRM2在GSH代谢通路上具有关键作用,和GSH合成密切相关[38]。此外,KLF11是生物体发育的过程中一个关键的调节因子,能通过下调Bcl-X的表达来诱导细胞死亡[39]。ATP6V1C2和GRHL1在胚胎发育、细胞周期、物质代谢方面发挥重要的调节作用[40-41]。根据以上分析推测,GSH处理可能影响lncRNA CUFF.21976.3表达,降低KLF11表达量,从而减少细胞死亡,提高胚胎发育率。因此推测lncRNA CUFF.21976.3在GSH保护胚胎发育的过程中发挥至关重要的作用,然而其具体的作用机制还需要进一步研究。
4 结论本研究采用单细胞测序技术对添加外源GSH处理的牛8~16-细胞期胚胎和桑椹胚进行转录组测序,筛选了差异表达lncRNAs,并对与其共表达的差异表达基因和顺式作用靶基因进行生物信息学分析,推测了lncRNA重要调节作用;为从lncRNA的角度揭示GSH提高胚胎抗氧化能力和促进胚胎发育的作用机制奠定了基础。近年来,lncRNA相关研究很多,但其在胚胎发育和胚胎细胞命运决定中的调控作用还需要深入地、系统地研究,为提高体外胚胎的质量和生产效率提供理论依据。
| [1] | DENNERY P A. Effects of oxidative stress on embryonic development[J]. Birth Defects Res C Embryo Today, 2007, 81(3): 155–162. DOI: 10.1002/(ISSN)1542-9768 |
| [2] | THOMPSON J G, MCNAUGHTON C, GASPARRINI B, et al. Effect of inhibitors and uncouplers of oxidative phosphorylation during compaction and blastulation of bovine embryos cultured in vitro[J]. J Reprod Fertil, 2000, 118(1): 47–55. DOI: 10.1530/reprod/118.1.47 |
| [3] | MAAS D H A, STOREY B T, MASTROIANNI L Jr. Oxygen tension in the oviduct of the rhesus monkey(Macaca mulatta)[J]. Fertil Steril, 1976, 27(11): 1312–1317. DOI: 10.1016/S0015-0282(16)42201-6 |
| [4] | QUINN P, HARLOW G M. The effect of oxygen on the development of preimplantation mouse embryos in vitro[J]. J Exp Zool, 1978, 206(1): 73–80. DOI: 10.1002/(ISSN)1097-010X |
| [5] | BATT P A, GARDNER D K, CAMERON A W. Oxygen concentration and protein source affect the development of preimplantation goat embryos in vitro[J]. Reprod Fertil Dev, 1991, 3(5): 601–607. DOI: 10.1071/RD9910601 |
| [6] | RHO G J, BALASUBRAMANIAN S, KIM D S, et al. Influence of in vitro oxygen concentrations on preimplantation embryo development, gene expression and production of Hanwoo calves following embryo transfer[J]. Mol Reprod Dev, 2007, 74(4): 486–496. DOI: 10.1002/(ISSN)1098-2795 |
| [7] | LUVONI G C, KESKINTEPE L, BRACKETT B G. Improvement in bovine embryo production in vitro by glutathione-containing culture media[J]. Mol Reprod Dev, 1996, 43(4): 437–443. DOI: 10.1002/(ISSN)1098-2795 |
| [8] | SUN W J, PANG Y W, LIU Y, et al. Exogenous glutathione supplementation in culture medium improves the bovine embryo development after in vitro fertilization[J]. Theriogenology, 2015, 84(5): 716–723. DOI: 10.1016/j.theriogenology.2015.05.001 |
| [9] | LI X X, LEE K B, LEE J H, et al. Glutathione and cysteine enhance porcine preimplantation embryo development in vitro after intracytoplasmic sperm injection[J]. Theriogenology, 2014, 81(2): 309–314. DOI: 10.1016/j.theriogenology.2013.09.030 |
| [10] | GARDINER C S, REED D J. Status of glutathione during oxidant-induced oxidative stress in the preimplantation mouse embryo[J]. Biol Reprod, 1994, 51(6): 1307–1314. DOI: 10.1095/biolreprod51.6.1307 |
| [11] | LEE C S, KOO D B, FANG N, et al. Potent and stage-specific action of glutathione on the development of goat early embryos in vitro[J]. Mol Reprod Dev, 2000, 57(1): 48–54. DOI: 10.1002/(ISSN)1098-2795 |
| [12] | DERRIEN T, JOHNSON R, BUSSOTTI G, et al. The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure, evolution, and expression[J]. Genome Res, 2012, 22(9): 1775–1789. DOI: 10.1101/gr.132159.111 |
| [13] | SHI X F, SUN M, LIU H B, et al. Long non-coding RNAs:a new frontier in the study of human diseases[J]. Cancer Lett, 2013, 339(2): 159–166. DOI: 10.1016/j.canlet.2013.06.013 |
| [14] | YAN L Y, YANG M Y, GUO H S, et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells[J]. Nat Struct Mol Biol, 2013, 20(9): 1131–1139. DOI: 10.1038/nsmb.2660 |
| [15] | DURRUTHY-DURRUTHY J, SEBASTIANO V, WOSSIDLO M, et al. The primate-specific noncoding RNA HPAT5 regulates pluripotency during human preimplantation development and nuclear reprogramming[J]. Nat Genet, 2016, 48(1): 44–52. |
| [16] | HAMAZAKI N, UESAKA M, NAKASHIMA K, et al. Gene activation-associated long noncoding RNAs function in mouse preimplantation development[J]. Development, 2015, 142(5): 910–920. DOI: 10.1242/dev.116996 |
| [17] | BRACKETT B G, OLIPHANT G. Capacitation of rabbit spermatozoa in vitro[J]. Biol Reprod, 1975, 12(2): 260–274. DOI: 10.1095/biolreprod12.2.260 |
| [18] | HARRIS M A, CLARK J, IRELAND A, et al. The Gene Ontology (GO) database and informatics resource[J]. Nucleic Acids Res, 2004, 32(D1): D258–D261. |
| [19] | YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene Ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biol, 2010, 11(2): R14. DOI: 10.1186/gb-2010-11-2-r14 |
| [20] | KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2008, 36(D1): D480–D484. |
| [21] | HUANG J L, ZHENG L, HU Y W, et al. Characteristics of long non-coding RNA and its relation to hepatocellular carcinoma[J]. Carcinogenesis, 2014, 35(3): 507–514. DOI: 10.1093/carcin/bgt405 |
| [22] | ZHANG K S, HUANG K F, LUO Y P, et al. Identification and functional analysis of long non-coding RNAs in mouse cleavage stage embryonic development based on single cell transcriptome data[J]. BMC Genomics, 2014, 15(1): 845. DOI: 10.1186/1471-2164-15-845 |
| [23] | LOEWER S, CABILI M N, GUTTMAN M, et al. Large intergenic non-coding RNA-RoR modulates reprogramming of human induced pluripotent stem cells[J]. Nat Genet, 2010, 42(12): 1113–1117. DOI: 10.1038/ng.710 |
| [24] | CABILI M N, TRAPNELL C, GOFF L, et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J]. Genes Develop, 2011, 25(18): 1915–1927. DOI: 10.1101/gad.17446611 |
| [25] | GUTTMAN M, GARBER M, LEVIN J Z, et al. Ab initio reconstruction of cell type-specific transcriptomes in mouse reveals the conserved multi-exonic structure of lincRNAs[J]. Nat Biotechnol, 2010, 28(5): 503–510. DOI: 10.1038/nbt.1633 |
| [26] | PAULI A, VALEN E, LIN M F, et al. Systematic identification of long noncoding RNAs expressed during zebrafish embryogenesis[J]. Genome Res, 2012, 22(3): 577–591. DOI: 10.1101/gr.133009.111 |
| [27] | BILLEREY C, BOUSSAHA M, ESQUERRÉ D, et al. Identification of large intergenic non-coding RNAs in bovine muscle using next-generation transcriptomic sequencing[J]. BMC Genomics, 2014, 15(1): 499. DOI: 10.1186/1471-2164-15-499 |
| [28] | RAO A M, DRAKE M R, STIPANUK M H. Role of the transsulfuration pathway and of gamma-cystathionase activity in the formation of cysteine and sulfate from methionine in rat hepatocytes[J]. J Nutr, 1990, 120(8): 837–845. |
| [29] | RIBAS V, GARCÍA-RUIZ C, FERNÁNDEZ-CHECA J C. Glutathione and mitochondria[J]. Front Pharmacol, 2014, 5: 151. |
| [30] | YIN J, REN W K, YANG G, et al. L-cysteine metabolism and its nutritional implications[J]. Mol Nutr Food Res, 2016, 60(1): 134–146. DOI: 10.1002/mnfr.201500031 |
| [31] | PEÑA-SÁNCHEZ M, RIVERÓN-FORMENT G, ZALDÍVAR-VAILLANT T, et al. Association of status redox with demographic, clinical and imaging parameters in patients with Huntington's disease[J]. Clin Biochem, 2015, 48(18): 1258–1263. DOI: 10.1016/j.clinbiochem.2015.06.014 |
| [32] | SMEYNE M, SMEYNE R J. Glutathione metabolism and Parkinson's disease[J]. Free Radic Biol Med, 2013, 62: 13–25. DOI: 10.1016/j.freeradbiomed.2013.05.001 |
| [33] | SAHARAN S, MANDAL P K. The emerging role of glutathione in Alzheimer's disease[J]. J Alzheimers Dis, 2014, 40(3): 519–529. |
| [34] | SHANG D H, ZHENG T, ZHANG J, et al. Profiling of mRNA and long non-coding RNA of urothelial cancer in recipients after renal transplantation[J]. Tumour Biol, 2016, 37(9): 12673–12684. DOI: 10.1007/s13277-016-5148-1 |
| [35] | AIRD K M, ZHANG G, LI H, et al. Suppression of nucleotide metabolism underlies the establishment and maintenance of oncogene-induced senescence[J]. Cell Rep, 2013, 3(4): 1252–1265. DOI: 10.1016/j.celrep.2013.03.004 |
| [36] | MORIKAWA T, HINO R, UOZAKI H, et al. Expression of ribonucleotide reductase M2 subunit in gastric cancer and effects of RRM2 inhibition in vitro[J]. Human Pathol, 2010, 41(12): 1742–1748. DOI: 10.1016/j.humpath.2010.06.001 |
| [37] | LIU X Y, ZHOU B S, XUE L J, et al. Ribonucleotide reductase subunits M2 and p53R2 are potential biomarkers for metastasis of colon cancer[J]. Clin Colorectal Cancer, 2007, 6(5): 374–381. DOI: 10.3816/CCC.2007.n.007 |
| [38] | GATTO F, MIESS H, SCHULZE A, et al. Flux balance analysis predicts essential genes in clear cell renal cell carcinoma metabolism[J]. Sci Rep, 2015, 5: 10738. DOI: 10.1038/srep10738 |
| [39] | WANG Z, SPITTAU B, BEHRENDT M, et al. Human TIEG2/KLF11 induces oligodendroglial cell death by downregulation of Bcl-XL expression[J]. J Neural Transm, 2007, 114(7): 867–875. DOI: 10.1007/s00702-007-0635-6 |
| [40] | SMITH A N, BORTHWICK K J, KARET F E. Molecular cloning and characterization of novel tissue-specific isoforms of the human vacuolar H+-ATPase C, G and d subunits, and their evaluation in autosomal recessive distal renal tubular acidosis[J]. Gene, 2002, 297(1-2): 169–177. DOI: 10.1016/S0378-1119(02)00884-3 |
| [41] | WILANOWSKI T, TUCKFIELD A, CERRUTI L, et al. A highly conserved novel family of mammalian developmental transcription factors related to Drosophila grainyhead[J]. Mech Develop, 2002, 114(1-2): 37–50. DOI: 10.1016/S0925-4773(02)00046-1 |



