2. 西南民族大学 青藏高原研究院, 成都 610041;
3. 金川畜牧兽医局, 金川 624100
2. Institute of Qinghai-Tibetan Plateau, Southwest Minzu University, Chengdu 610041, China;
3. Jinchuan Animal Husbandry and Veterinary Bureau, Jinchuan 624100, China
青藏高原是中国最大,世界海拔最高的高原,境内面积240万平方公里,平均海拔4 000~5 000米,有“世界屋脊”和“第三极”之称。青藏高原气候和环境非常严酷,具有低温、低氧、强紫外线等。但是生活在该地区的动物对当地极端环境有很好的适应能力[1]。牦牛是青藏高原高寒牧区的特有畜种,也是牛属动物中能适应高寒高海拔气候的珍稀畜种资源[2-3]。与黄牛相比,牦牛能很好的适应青藏高原高海拔、高寒、极度缺氧等恶劣的自然环境,并在生理、生化和形态学上形成了稳定的适应高原低氧的独特遗传学特征和机制,被认为是研究哺乳动物高原适应性的良好模式动物。
肺是呼吸系统中重要的功能器官,动物机体通过肺从外界环境摄取氧气并进一步通过交换系统为机体提供所需的氧分子。在低氧环境中肺组织通过一系列的生理变化调整使动物机体适应外界环境刺激,但是牦牛肺组织适应低氧环境的遗传机制目前尚不清楚。2012年完成的牦牛基因组数据为后续相关研究奠定了重要的基础和平台,开启了牦牛组学研究的新时代[4]。同时随着RNA-Seq测序技术的日益成熟[5-6],通过大规模转录组深度测序在RNA水平上探究物种驯化起源、分化、分类状况,疾病发生机理、育种及遗传分子机制等一系列问题已经成为研究者的主要策略之一[7]。截止目前,关于高原土著动物低氧适应性相关转录组学已有相关报道,王翔宇等通过转录组RNA-Seq技术比较、分析和探讨了藏鸡胚胎低氧适应性的遗传机制[8],姚娜[9]对辽宁绒山羊和西藏班戈绒山羊的11个不同组织包括心、呼吸道、肝、脾、骨髓、肾、皮肤毛囊、肺、大脑皮层、胆囊、骨骼肌等的RNA进行转录组测序,成功筛选出7个与高海拔环境适应性相关的基因。但涉及牦牛转录组学方面的研究很少且主要集中在生殖繁育方面,其中,兰道亮等[10]在RNA水平对牦牛繁殖相关基因进行了分析,完善并对相关新基因进行了深度挖掘和分析,钟金城等[11]对犏牛和牦牛睾丸组织的转录组进行了分析研究,曾贤彬等[12]通过对牦牛与犏牛精原细胞的转录组测序研究探讨了犏牛精子发生阻滞的原因和机制。然而,牦牛作为研究高原低氧适应性的良好模式动物,其在低氧适应机理相关的研究却只集中在单个基因层面,全基因组层面的研究尚未展开。
本研究利用最新的RNA-Seq测序技术对牦牛肺转录组进行测序,描绘出牦牛肺正常转录组图谱,并与黄牛肺转录组比较,筛选出正选择基因,为进一步从分子水平解释牦牛高原低氧适应性的独特进化过程及遗传机制奠定基础,同时也为注释和补充牦牛基因组提供了有价值的数据。
1 材料与方法 1.1 试验材料在高原屠宰场(中国四川阿坝州红原县,北纬31°51′至33°19′,东经101°51′至103°23′;平均海拔3 600 m)随机选取成年健康雄性麦洼牦牛3头(年龄3~5岁,体重300~400 kg),屠宰后立即采集肺组织样本,均放入液氮速冻,送回实验室-80 ℃保存,用于提取组织总RNA;3头黄牛肺组织样本采自成都青白江唐家寺向阳牛市屠宰场(中国四川成都,北纬30°05′至31°26′,东经102°54′至104°53′;平均海拔高度为750 m),方法同上。本研究获得西南民族大学动物伦理与福利协会批准,所有样本均采集自政府审查批准的屠宰场。
1.2 RNA提取、cDNA文库构建和Illumina测序应用TRIzol试剂(Life Technologies公司,美国)提取牦牛和黄牛肺组织的总RNA。提取后的总RNA样品均以无RNase的DNAse I(TaKaRa公司,大连)进行处理,以消除可能存在的基因组DNA污染。然后分别等量混合3头牦牛和黄牛的RNA样品,组成2个RNA池(RNA pool,各30 μg)。使用Oligotex mRNA小量提取试剂盒(Qiagen公司,德国)从RNA池中分离和纯化mRNA。使用Nanodrop ND-1000分光光度计(LabTech,美国)和Agilent 2100生物分析仪(Agilent公司,美国)对整个流程中RNA的质量和浓度进行测定。利用TruSeq RNA Sample Prep Kit试剂盒(Illumina公司,美国)构建随机片段测序文库,即用打断试剂在恒温混匀仪中对纯化的mRNA进行片段化(约200 bp),而后将其作为cDNA第一链合成的模板;随后使用RNase H、dNTP及DNA聚合酶I合成第二链cDNA;纯化和配对末端修复后,将cDNA片段连接到测序接头并通过聚合酶链反应(PCR)对其进行扩增,获得最终的双末端(Paired-end,PE)测序文库。使用Agilent2 100生物分析仪和ABI StepOnePlus实时PCR系统执行质控(QC)测试后,在Illumina HiSeq 2 500平台上对文库进行测序。
1.3 转录组数据分析对Hiseq 2 500测序产生的原始读数进行QC测试后,通过去除接头序列、空序列及低质量测序序列(≤Q20质量值),将原始读数过滤为净测序序列(Clean reads)。使用SOAPaligner/SOAP2软件,将过滤后序列与牦牛基因组(版本1.0) 进行比对,统计测序序列(reads)在参考基因组及基因序列上的分布情况及覆盖度。应用BlastGO程序将比对上的基因与GO(Geneontology)数据库(http://www.geneontology.org/)进行比对,然后应用WEGO程序对这些基因所涉及的主要生物学功能按细胞组分、分子功能和生物过程等类别进行GO分类注释。通过与KEGG(Kyoto encyclopedia of genes and genomes)数据库(http://wego.genomics.org.cn)进行比对,对这些基因所涉及的信号通路或代谢途径(Pathway)进行分析。利用cufflinks软件将比对上基因组的测序序列进行组装拼接,在过滤掉低质量序列(长度≤180 bp, Q值≤10) 后,将组装的转录本序列与牦牛基因组上基因注释信息进行比较,如与现有基因两翼有交叉重叠(≥200 bp),则可能发现组装转录本会延长已注释基因的5′-或3′-末端, 由此优化基因结构;若组装的转录本序列未与现有基因比对上, 而是位于某些基因之间的基因组上, 但又同时满足以下条件:距离现有注释基因200 bp以上,长度不短于180 bp,测序深度不小于2,则这些序列有可能为潜在的新转录本;利用CPC(Coding potential calculator)程序可预测新转录本的编码能力,而相关的注释则可通过应用BLAST程序与Nr/Nt (ftp://ftp.ncbi.nih.gov/blast/db/nt/)及SwissProt数据库比对来完成。
1.4 正选择基因检验Ka/Ks,即异义替换速率和同义替换速率,用来判断是否有选择压力作用于该基因。当Ka/Ks=1时,表明该基因受到中性选择作用,不影响蛋白质结构和功能;Ka/Ks < 1时,该基因受到纯化选择作用,表明该基因对维持蛋白质的正常结构有着重要的作用,很难在替代的情况下维持正常功能;当Ka/Ks>1时,该基因受到正选择作用,改变蛋白质的结构和功能,有利于个体的生存和繁殖。我们运用KaKs_Calculator软件计算牦牛和黄牛肺各表达基因的Ka、Ks以及Ka/Ks值,筛选牦牛肺中Ka/Ks>1的表达基因作为牦牛特殊的正选择基因[13]。同时,为了进一步研究这些正选择基因的相关功能,我们应用GO和KEGG通路富集分析(P≤0.05),对这些基因的功能进行注释。
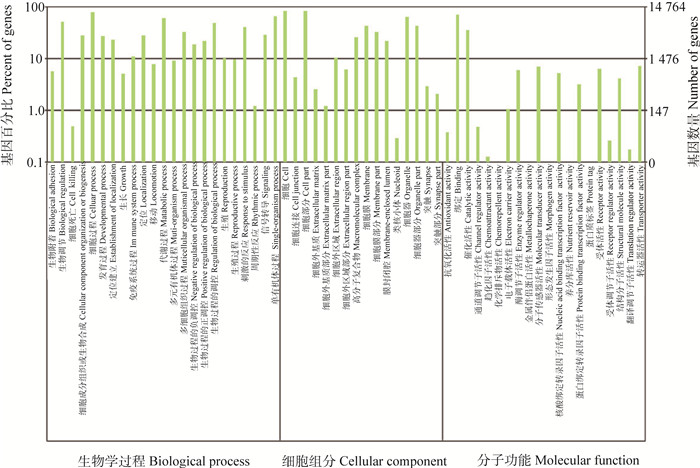
2 结果 2.1 牦牛肺组织转录组测序基因组比对分析及数据注释经Illumina HiSeq2 500深度测序后,去除接头序列、空读序列以及低质量序列后在牦牛转录测序文库中共获得51 641 282条过滤测序序列,包含4 647 715 380 bp;碱基组成和质量分析表明,原始测序数据的碱基组成中低质量碱基测序序列(≤Q20) 的比例很低,说明测序质量良好。将牦牛测序文库与牦牛基因组及相关参考基因进行比对分析,共有31 898 650条(61.77%)及18 409 161条(35.65%)测序序列分别比对上牦牛基因组及相关参考基因。基因覆盖度统计表明,牦牛转录组基因的覆盖率达90%~100%,基因数有11 221条,占所有比对上基因的67%,80%~90%的基因(1 412条),占所有比对上基因的8%,这一结果表明测序结果对基因的覆盖度良好。GO分析表明,共有14 764个基因得到分类注释,涉及细胞组分、生物学过程及分子功能3个大类59个小类(图 1)。在细胞组分分类中,最为富集的是细胞类别,其次是细胞部分类别及细胞器部分类别;在生物学过程分类中,最为富集的是细胞过程类别,其次为代谢过程类别及单有机体过程类别;在分子功能分类中,最为富集的是绑定类别,其次为催化活性类别及分子传感器功能类别。KEGG分析表明,共14 485个基因涉及258个通路,其中富集前10通路如表 1所示,代谢途径通路最为富集,其次为粘着斑通路及阿米巴病通路。
|
图 1 牦牛肺转录组GO分类注释图 Figure 1 The GO categories in transcriptome of yak lung |
|
|
表 1 牦牛肺转录组中前10位富集的KEGG通路 Table 1 Top 10 enriched KEGG pathways in transcriptome of yak lung |
在黄牛转录测序文库中共获得了54 720 968条过滤测序序列,包含4 924 882 170 bp,碱基组成和质量分析结果良好,可用于后续的研究分析。
2.2 基因结构优化及新转录本预测牦牛肺转录组基因结构优化分析表明,有8 123个基因的5′-或3′-末端在原有基础上发生了延伸,其中4 719个基因的5′-端发生了延伸,3 404个基因的3′-端发生了延伸。牦牛肺转录组中有7 059个新转录本,长度为180~14 884 bp,其中存在1~84个外显子。2 795个新转录本应该具有编码蛋白的能力。
2.3 正选择基因检验及GO功能KEGG通路显著性富集分析Ka/Ks分析显示,与黄牛相比,牦牛有39个基因的Ka/Ks>1,受到了强烈的正向选择。对正选择基因的GO分析表明它们涉及到细胞组分、生物学过程及分子功能;进一步的富集分析表明,细胞组分中有53个分类条目,TOP10(表 2)中与核糖体相关的类别所占比例最大(4/10),生物学过程中有297个分类条目,TOP10(表 3)中与免疫细胞相关的类别所占比例最大(7/10),分子功能涉及81个分类条目,TOP10(表 4)中与酶活性相关的类别所占比例最大(5/10)。KEGG注释表明它们涉及到56个通路,前10的通路(表 5)中阿尔滋海默氏病通路最为富集,其次为代谢途径及糖酵解/糖异生途径。
|
|
表 2 正选择基因GO注释细胞组分类别富集前10(TOP10) Table 2 Top 10 enriched GO categories in the cellular components of positively selected genes |
|
|
表 3 正选择基因GO注释生物学过程分类别富集前10(TOP10) Table 3 Top 10 enriched GO categories in the biological processes of positively selected genes |
|
|
表 4 正选择基因GO注释分子功能分类别富集前10(TOP10) Table 4 Top 10 enriched GO categories in the molecular functions of positively selected genes |
|
|
表 5 正选择基因KEGG通路分析富集前10(TOP10) Table 5 Top 10 enriched KEGG pathways in positively selected genes |
牦牛在长期适应青藏高原高海拔、严寒、极度缺氧等恶劣自然环境的过程中很多基因特异性的提高了相应的转录活性,成为研究高原哺乳动物低氧适应性的良好模式动物。肺是呼吸系统中重要的功能器官,在低氧环境中肺组织通过一系列的生理变化使动物机体适应外界环境刺激得以生存,但肺转录组学的研究尚属空白。本研究采用转录组高通量测序技术RNA-Seq对牦牛及普通黄牛肺组织进行转录组测序。通过对转录组数据注释,描绘出了牦牛肺的正常转录组图谱;通过与黄牛转录组的比较,筛选出了正选择基因,进一步从分子水平解释牦牛高原低氧适应性的独特的进化过程及遗传机制,与此同时该研究成果也为进一步完善牦牛基因组数据库提供了有价值的数据。
3.1 牦牛肺正常转录组轮廓及特点GO分析显示,在细胞组分分类中除细胞类别最为富集外,细胞膜类别也占了很大的比例。众所周知,细胞膜的主要功能是物质运输以及为细胞正常的生命活动提供稳定的内环境;肺是动物机体进行呼吸作用的主要场所,特别是在极度低氧的环境中,在细胞分子层面上发生的有氧及无氧呼吸链作用、涉及血红蛋白氧气运输等大量的生理活动及生物化学反应来满足机体对氧的需求,这些过程都需要细胞膜以及膜内外大量分子及功能的参与才能完成,这提示细胞膜在牦牛肺低氧环境生理活动中扮演了非常重要的角色。生物学过程类别中最为富集是细胞过程,其次是代谢过程,细胞代谢相关的生物学调控过程富集说明牦牛通过适应了青藏高原高寒、低氧的环境,形成了其特异的生理和遗传特征。分子功能类别中最为富集的是绑定分子功能,与以前报道的牦牛其他组织转录组的结果一致[10],这说明绑定在牦牛肺正常的生理活动中频繁发生且有着重要的地位。KEGG分析显示代谢途径最为富集,这符合牦牛肺生理活动的特点。
3.2 正选择基因的检验及其功能分析利用Ka/Ks统计分析在牦牛中筛选到了39个受到正选择的基因。GO分析表明,细胞组分中与核糖体相关的类别所占比例最大(4/10),核糖体是蛋白质的合成场所,而蛋白质是机体生命活动的主要承担者,有很多重要的生理功能,这说明蛋白质的合成在牦牛肺低氧环境生理活动中扮演了重要的角色。此外,在细胞组分中线粒体也占了较大的比例,线粒体被称为机体内的“能量工厂”。现有的研究表明,生活在寒冷海域的鱼类,线粒体基因受到的进化压力大于热带海域的鱼类,表明不同环境中物种对能量的需求不同而导致线粒体受到的进化压力不同,说明线粒体的功能在牦牛的低氧适应性机制中扮演了重要的角色[14]。
生物学过程中,与免疫细胞移动调控相关的类别占了很大的比例(7/10),除了单核细胞和白细胞迁移的调控,其余均与免疫细胞趋化作用有关。趋化作用是指免疫细胞在趋化因子的吸引下做定向移动,在炎症反应中有重要作用。机体在高海拔、低氧量、高紫外的恶劣环境中,容易造成肺功能性损伤,趋化作用引导免疫细胞移向感染部位,使得机体更加积极地应对疾病,发挥重要的免疫功能。研究表明,在低氧环境下,多形核白细胞趋化活性显著高于常氧情况[15],与本研究结果一致。无论是免疫细胞的迁移还是趋化,都与机体的免疫机制和抗病有关。以此推断,牦牛在长期进化中有可能已经形成了一套特有的免疫机制以抵抗高原的恶劣环境。除此之外,蛋白质亚硝基化类别在生物学过程中也占到了一定的比例(3/10),蛋白质亚硝基化是指一氧化氮对半胱氨酸巯基进行一种可逆的氧化修饰。这种蛋白质翻译后修饰,可以作为信号分子介导信号通路,也可以调控蛋白质的功能。目前的研究表明,蛋白质亚硝基化对血液输氧有着重要的影响,它可以调控血流量随着外界氧浓度不同而变化[16-17],在肺发生的蛋白质亚硝基化反应主要是为了帮助血液的氧合作用。由此可以推断,蛋白质亚硝基化在牦牛高原低氧适应机制中有着重要的作用。
在分子功能分类中,酶活性分子功能类别最为显著富集,而且其相关类别在Top10富集分类中也占据了很高比例(5/10),具体为3-磷酸甘油醛脱氢酶活性、磷酸酯水解酶活性、磷酸脂酶活性、脂肪酶活性及磷脂酰肌醇磷脂酶C活性。3-磷酸甘油醛脱氢酶是糖酵解中心环节的关键酶,它催化3-磷酸甘油醛氧化和磷酸化ADP产生ATP,它不仅与机体供能有关,而且能加强低氧情况下糖的酵解[18],为机体提供更多的能量。磷酸酯水解酶分为磷酸单酯酶和磷酸二酯酶,其中磷酸二酯酶的主要功能是水解细胞内的第二信使(环磷酸鸟苷cGMP或环磷酸腺苷cAMP),可调节细胞内多种信号通路和活动[19]。磷脂酶是一类能水解甘油磷脂的酶,分为A1、A2、B、C、D五大类,它们的功能除了水解磷脂外,也参与细胞膜结构的维护和细胞内信号传导的调节[20],其中磷脂酶A2与肺泡表明活性物质代谢有关[21]。脂肪酶是一类有着重要功能的酶,参与机体多个代谢过程,例如血清脂蛋白脂肪酶可以加快体内甘油三酯的清除以及提高脂肪酸的利用,对机体的血脂和胆固醇代谢有着重要的影响[22-23]。酶参与机体的多种生理活动过程,这些类别的富集说明牦牛在长期进化中逐渐形成了一套特有的生理机制,增加产能、调节细胞活动、提高脂肪酸的利用等等都是为了抵御高海拔、低氧量的恶劣环境。
KEGG分析显示,代谢通路、糖酵解/糖异生通路、核黄素代谢通路、叶酸的一碳代谢通路、嘧啶代谢通路、氧化磷酸化通路及二羧酸代谢通路等7个涉及代谢的通路排在前10位之内。糖异生是指非糖化合物转变为葡萄糖或糖原的过程,是草食动物体内主要的糖的来源,在饥饿或摄入糖不足时,机体通过糖异生来维持体内的血糖稳定[24-25]。糖酵解是指糖类在无氧条件下的分解代谢,可为机体提供能量,它的关键酶3-磷酸甘油醛脱氢酶在GO分析中也较为富集,糖酵解/糖异生通路的富集说明糖代谢为牦牛在低氧环境中提供更多的能量以对抗恶劣的生存环境。核黄素又称维生素B2,在机体内以黄素腺嘌呤二核苷酸(FAD)和黄素单核苷酸(FMN)两种形式参加氧化还原反应,也是一些酶的辅酶。低氧环境下,维生素B2对机体碳水化合物和脂肪的代谢有一定改善作用,可提高动物的代谢率,增加血液中的ATP[26];维生素B2还通过影响小肠NADH-FMN氧化还原酶来调节铁的代谢[27],而铁作为血红蛋白的基本元素,对血液运输氧的能力有着直接的影响,因此推断,核黄素代谢通路的富集应该是牦牛在长期进化中通过调节自身代谢而逐渐适应低氧环境的结果。嘧啶代谢及叶酸的一碳代谢与DNA的合成修复有关,嘧啶是DNA的组成部分,叶酸的一碳代谢是指氨基酸代谢过程中产生的含一个碳原子的基团必须和四氢叶酸结合而转运及参加代谢,是合成嘧啶、嘌呤的原料。如果叶酸摄入不足或代谢障碍,则会影响DNA的完整性及其修复[28-29]。高原紫外线强,而紫外线可对DNA造成损伤, 我们推断这两个通路的富集对牦牛抵御高原紫外线的损伤有着重要作用。氧化磷酸化是需氧生物体内合成ATP的重要方式,其在线粒体中进行,与GO分析中线粒体类别富集的结果相一致,牦牛在进化中通过改善机体供能来促进其适应高原的寒冷低氧环境。二羧酸代谢与脂肪代谢有关,这和GO分析中脂肪酶代谢类别富集的结果一致。
KEGG富集的前10个通路中的另外3个通路与疾病相关,分别是阿尔滋海默氏症通路、亨廷顿舞蹈症通路以及帕金森氏病通路,这3类通路看上去和牦牛肺的生理过程并没有直接联系,但这3个疾病的通路所导致的生理现象均与细胞凋亡有关。细胞凋亡对于组织的分化、器官发育及维持稳态有着重要的作用[30],机体在产生新细胞的同时通过凋亡及时清除衰老或者突变细胞,我们推断这可能与牦牛在高原环境中的机体自我保护有关。
4 结论本研究运用RNA-Seq测序技术对牦牛肺转录组进行分析,绘制了牦牛肺正常转录组图谱,并通过与黄牛肺转录组的比较,筛选出受到正选择的基因,为进一步从分子水平解释牦牛高原低氧适应性的独特进化过程及遗传机制奠定了基础,同时也为注释和补充牦牛基因组提供有价值的数据。
| [1] | MIAO F, GUO Z, XUE R, et al. Effects of grazing and precipitation on herbage biomass, herbage nutritive value, and yak performance in an alpine meadow on the Qinghai-Tibetan Plateau[J]. PLoS One, 2015, 10(6): e0127275. DOI: 10.1371/journal.pone.0127275 |
| [2] | FU M, CHEN Y B, XIONG X R, et al. Establishment of mammary gland model in vitro:culture and evaluation of a yak mammary epithelial cell line[J]. PLoS One, 2014, 9(12): e113669. DOI: 10.1371/journal.pone.0113669 |
| [3] | GUO X S, LONG R J, KREUZER M, et al. Importance of functional ingredients in yak milk-derived food on health of Tibetan nomads living under high-altitude stress:a review[J]. Crit Rev Food Sci Nutr, 2014, 54(3): 292–302. DOI: 10.1080/10408398.2011.584134 |
| [4] | QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J]. Nat Genet, 2012, 44(8): 946–949. DOI: 10.1038/ng.2343 |
| [5] | WANG Z, GERSTEIN M, SNYDER M. RNA-Seq:a revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10(1): 57–63. DOI: 10.1038/nrg2484 |
| [6] | RICHARD H, SCHULZ M H, SULTAN M, et al. Prediction of alternative isoforms from exon expression levels in RNA-Seq experiments[J]. Nucleic Acids Res, 2010, 38(10): e112. DOI: 10.1093/nar/gkq041 |
| [7] | MIAO X Y, LUO Q M. Genome-wide transcriptome analysis between small-tail Han sheep and the Surabaya fur sheep using high-throughput RNA sequencing[J]. Reproduction, 2013, 145(6): 587–596. DOI: 10.1530/REP-12-0507 |
| [8] |
王翔宇. 藏鸡胚胎低氧适应的转录组差异表达分析[D]. 北京: 中国农业大学, 2014.
WANG X Y. Transcriptomic difference analysis in Tibetan chicken embryo at hypoxia[D]. Beijing:China Agricultural University, 2014. (in Chinese) |
| [9] |
姚娜. 西藏班戈绒山羊高海拔环境适应性的遗传分析[D]. 北京: 中国农业科学院, 2014.
YAO N. Genetic analysis of high altitude adaptability of Bange cashmere goat in Tibet[D]. Beijing:Chinese Academy of Agricultural Sciences, 2014. (in Chinese) |
| [10] |
兰道亮, 熊显荣, 位艳丽, 等. 基于RNA-Seq高通量测序技术的牦牛卵巢转录组研究:进一步完善牦牛基因结构及挖掘与繁殖相关新基因[J]. 中国科学:生命科学, 2014, 44(3): 307–317.
LAN D L, XIONG X R, WEI Y L, et al. RNA-Seq analysis of yak ovary:improving yak gene structure information and mining reproduction-related genes[J]. Scientia Sinica Vitae, 2014, 44(3): 307–317. (in Chinese) |
| [11] |
钟金城, 姬秋梅, 柴志欣, 等. 基于转录组测序对犏牛和牦牛睾丸组织的分析研究[C]//中国遗传学会第九次全国会员代表大会暨学术研讨会论文集. 哈尔滨: 中国遗传学会, 2013.
ZHONG J C, JI Q M, CHAI Z X, et al. Transcriptomic analyses of testicular tissue of dzo and yak[C]//Abstracts Presented at China's Ninth National Congress of the Members of the Genetics Society and Academic Seminar Harbin:Chinese Genetics Society, 2013. (in Chinese) http://cpfd.cnki.com.cn/Article/CPFDTOTAL-ZGYL201309001225.htm |
| [12] |
曾贤彬, 柴志欣, 王永, 等. 犏牛精子发生阻滞的比较转录组研究[J]. 中国科学:生命科学, 2014, 44(6): 584–601.
ZENG X B, CHAI Z X, WANG Y, et al. Comparative transcriptome analysis of spermatogenesis arrest in cattle-yak[J]. Scientia Sinica Vitae, 2014, 44(6): 584–601. (in Chinese) |
| [13] | WANG D P, ZHANG Y B, ZHANG Z, et al. KaKs_Calculator 2.0:a toolkit incorporating Gamma-series methods and sliding window strategies[J]. Genomics Proteomics Bioinformatics, 2010, 8(1): 77–80. DOI: 10.1016/S1672-0229(10)60008-3 |
| [14] | SUN Y B, SHEN Y Y, IRWIN D M, et al. Evaluating the roles of energetic functional constraints on teleost mitochondrial-encoded protein evolution[J]. Mol Biol Evol, 2011, 28(1): 39–44. DOI: 10.1093/molbev/msq256 |
| [15] | GLADWIN M T, CRAWFORD J H, PATEL R P. The biochemistry of nitric oxide, nitrite, and hemoglobin:role in blood flow regulation[J]. Free Radic Biol Med, 2004, 36(6): 707–717. DOI: 10.1016/j.freeradbiomed.2003.11.032 |
| [16] | LIMA B, FORRESTER M T, HESS D T, et al. S-Nitrosylation in cardiovascular signaling[J]. Circul Res, 2010, 106(4): 633–646. DOI: 10.1161/CIRCRESAHA.109.207381 |
| [17] | BOLOTINA V M, NAJIBI S, PALACINO J J, et al. Nitric oxide directly activates calcium-dependent potassium channels in vascular smooth muscle[J]. Nature, 1994, 368(6474): 850–853. DOI: 10.1038/368850a0 |
| [18] | GASSMANN M, WENGER R H. HIF-1, a mediator of the molecular response to hypoxia[J]. Physiology, 1997, 12(3): 214–218. |
| [19] | RONDINA M T, WEYRICH A S. Targeting phosphodiesterases in anti-platelet therapy[M]//GRESELE P, BORN G V, PATRONO C, et al. Antiplatelet Agents. Berlin Heidelberg:Springer, 2012:225-238. |
| [20] | CASADO V, MARTIN D, TORRES C, et al. Phospholipases in food industry:a review[M]//SANDOVAL G. Lipases and Phospholipases. New York:Humana Press, 2012:495-523. https://link.springer.com/protocol/10.1007/978-1-61779-600-5_29 |
| [21] | MURAKAMI M, SHIMBARA S, KAMBE T, et al. The functions of five distinct mammalian phospholipase A2S in regulating arachidonic acid release:type ⅡA and type V secretory phospholipase A2S are functionally redundant and act in concert with cytosolic phospholipase A2[J]. J Biol Chem, 1998, 273(23): 14411–14423. DOI: 10.1074/jbc.273.23.14411 |
| [22] | GILL J M R, HERD S L, VORA V, et al. Effects of a brisk walk on lipoprotein lipase activity and plasma triglyceride concentrations in the fasted and postprandial states[J]. Eur J Appl Physiol, 2003, 89(2): 184–190. DOI: 10.1007/s00421-002-0788-9 |
| [23] | KIENS B, LITHELL H. Lipoprotein metabolism influenced by training-induced changes in human skeletal muscle[J]. J Clin Invest, 1989, 83(2): 558–564. DOI: 10.1172/JCI113918 |
| [24] | STAEHR P, HOTHER-NIELSEN O, BECK-NIELSEN H. The role of the liver in type 2 diabetes[J]. Rev Endocr Metab Disord, 2004, 5(2): 105–110. DOI: 10.1023/B:REMD.0000021431.90494.0c |
| [25] | LIBNKE P L, DEFRAIN J M, HIPPEN A R, et al. Ruminal and plasma responses in dairy cows to drenching or feeding glycerol[J]. J Dairy Sci, 2004, 87(S1): 343. |
| [26] |
陈东升, 杨家驹, 朱俊东, 等. 营养干预对动物急性缺氧能量代谢的影响[J]. 营养学报, 1997, 19(4): 423–426.
CHEN D S, YANG J J, ZHU J D, et al. The effect of nutrition intervention on the energy metabolism in acute anoxic rats[J]. Acta Nutrimenta Sinica, 1997, 19(4): 423–426. (in Chinese) |
| [27] | ADELEKAN D A, THURNHAM D J. The influence of riboflavin deficiency on absorption and liver storage of iron in the growing rat[J]. Br J Nutr, 1986, 56(1): 171–179. DOI: 10.1079/BJN19860097 |
| [28] | FENECH M. The role of folic acid and vitamin B12 in genomic stability of human cells[J]. Mutat Res, 2001, 475(1-2): 57–67. DOI: 10.1016/S0027-5107(01)00079-3 |
| [29] | KIM Y I. Folate and colorectal cancer:An evidence-based critical review[J]. Mol Nutr Food Res, 2007, 51(3): 267–292. DOI: 10.1002/(ISSN)1613-4133 |
| [30] | LIN L, ZAKERI Z F. Apoptosis in development[M]//TUAN R S, LO C W. Developmental Biology Protocols:Volume Ⅱ. Totowa, NJ:Humana Press, 2000:107-113. https://es.scribd.com/document/272520561/Hood-2006-Developmental-and-Reproductive-Toxicology |



