2. 福建农林大学动物科学学院, 福州 350002
2. College of Animal Science, Fujian Agriculture and Forestry University, Fuzhou 350002, China
畜禽的大多数经济性状都是数量性状,数量性状受多个基因控制[1]。当今学术界认同的数量性状的潜在遗传模型是寡基因模型,即数量性状由少数几个主效基因和大量微效基因共同控制。在连锁分析中,这些有效应的基因被称为数量性状位点(QTL)。在过去的10年,全基因组关联分析(GWAS)广泛地用于定位数量性状QTL,并成功找到了大量候选基因[2-3]。但是GWAS所检测到的位点只能解释复杂性状一部分的加性遗传方差,这种“缺失遗传力”的问题逐渐吸引了大量学者的关注[4]。GWAS基于“常见疾病,常见变异”的假设,主要评估常见遗传变异(最小等位基因频率(MAF)>5%的遗传变异)对表型的作用,所以被GWAS忽略的稀有变异(MAF<5%)很可能是造成“缺失遗传力”的原因[5]。其次,许多被以家族设计为基础的连锁分析鉴定并确认的区域,并没有在随后的GWAS分析中找到相关联的单核苷酸多态性位点(SNP),这一方面是由于连锁分析的假阳性所致,另一方面也可能是区域中的稀有变异作用的结果[6]。在人类基因组中等位基因的非同义突变、可变剪切、蛋白质删除等大多数都是稀有变异[7],同时人类复杂疾病的研究已成功鉴定到许多具有效应的稀有变异[8-9],因此对稀有变异的研究有助于进一步增加对性状遗传结构和生物学机理的理解。目前在畜禽上对稀有变异的研究很少,主要原因是关联分析对样本群体规模的要求及其昂贵的测序费用,但是随着测序技术的发展和成本的下降,稀有变异终将会在畜禽数量性状的研究上扮演重要角色。
1 稀有变异关联方法 1.1 折叠多个位点GWAS最常用、效果最好的方法是基于混合线性模型对标记进行逐个检验,然而由于稀有变异单个变异出现频率低,其所能提供的信息很少,所以该方法对稀有变异的检测效力较低。为了增加稀有变异的检测效力,通常将一个兴趣区域内的稀有变异折叠起来与表型进行关联。考虑如下线性模型(加粗的为向量):
| $ {\mathit{y}_\mathit{i}} = {\mathit{\alpha }_0} + \mathit{\boldsymbol{\alpha '}}{\mathit{\boldsymbol{X}}_\mathit{i}} + \mathit{\boldsymbol{\beta '}}{\mathit{\boldsymbol{G}}_\mathit{i}} + {\mathit{\varepsilon }_\mathit{i}} $ |
yi是第i个个体的表型;α0是截距;Xi是场、年、季等固定效应;Gi是兴趣区域内的基因分型;
SKAT[12]是方差组分检验的一种,考虑和上式相同的线性模型:
| $ {y_i} = {\alpha _0} + \mathit{\boldsymbol{\alpha '}}{\mathit{\boldsymbol{X}}_i} + \mathit{\boldsymbol{\beta '}}{\mathit{\boldsymbol{G}}_i} + {\varepsilon _i} $ |
SKAT假设每个β服从平均值为0,方差为wjτ的任意分布(wj是预先给定的变异j的权重,τ是方差组分)。此时对应的零假设可以转变为τ=0,可以使用方差组分score检验,score统计量构建:
| $ Q = \left( {y-\hat \mu } \right)K\left( {y-\hat \mu } \right) $ |
y是表型,
Wj是预先给定的变异j的权重,其反映了变异j对score统计量的相对贡献。SKAT从贝塔分布中抽取权重值,令
SKAT家族主要包括SKAT-O,RC-SKAT、meta-SKAT、CEP-SKAT、gSKAT和iSKAT。
1.3.1 SKAT-O兴趣区域内含有大量与表型相关且效应方向一致的标记时,负担检验将比方差组分检验更有效力。为了克服这个缺陷,S.Lee等提出了SKAT-O[14-15],用于结合负担检验和方差组分检验的优势,使结果更符合性状的潜在遗传机理。首先通过将负担检验和方差组分检验结合,公式:
| $ {Q_p} = \rho {Q_b} + \left( {1-\rho } \right){Q_s}, 0 \le \rho \le 1 $ |
Qb和Qs分别是零模型下负担检验和方差组分检验(SKAT)的score统计量。ρ代表各标记间的相关性。当完全相关时,ρ=1。此时Qp=Qb,即只会用负担检验。反之,只使用方差组分检验。不同的性状遗传结构对应不同的最优值,最优值可以通过优化检验程序[15]获得。
1.3.2 RC-SKATSKAT假设变异最小等位基因频率越小,其效应越大。使用基于贝塔分布的加权方法,增加稀有变异权重的同时降低常见变异的权重。如果常见变异对性状有很大效应时,这样的加权方法会降低关联效力。RC-SKAT[16]使用大样本理论阈值法将标记划分为稀有变异和常见变异后分开进行计算,然后适应性加权结合[17]稀有变异和常见变异的score统计量或使用Fisher法直接结合P值。大样本理论阈值法公式:
| $ T = \frac{1}{{\sqrt {2n} }} $ |
n表示样本大小。最小等位基因频率小于T的定义为稀有变异,反之定义为常见变异。
1.3.3 meta-SKAT及其他不同于传统的以回归系数为基础的单标记荟萃分析,meta-SKAT[18]提出了结合单个变异的score统计量而不是P值的一般框架。优势在于不需要重新估计标记回归系数,大大减轻计算负担。CEP-SKAT[19]将极端表型抽样与SKAT相结合,把极端表型当做病例对照模型处理。gSKAT[20]是为家庭数据关联分析而设计的。当使用混合模型校正群体家庭关系时,数据中的未测量遗传因素和共同环境因素都会对结果造成影响。gSKAT使用基于类核机器估计等式的统计量检验表型和兴趣区域的关联,可以有效解决家庭内相关性被错误指定的问题[21]。iSKAT[22]将加权线性核关联检验方法运用到检测在存在稀有变异时基因与环境的互作效应。其线性模型(加粗的为矩阵):
| $ {y_i} = {\mathit{\boldsymbol{X'}}_{i{\alpha _1}}} + {E_{i{\alpha _2}}} + {\mathit{\boldsymbol{G'}}_{i{\alpha _3}}} + {\left( {{\mathit{\boldsymbol{G}}_i}{\mathit{\boldsymbol{E}}_i}} \right)^\prime }\beta + {\rm{ \mathsf{ ε} }} $ |
X′iα1、Eiα2和G′iα3分别是协变量项、环境效应项和基因效应项。该模型零假设为β=0,即基因与环境的互作效应为0。采用SKAT一样的加权序列核关联检验方法检验零假设。
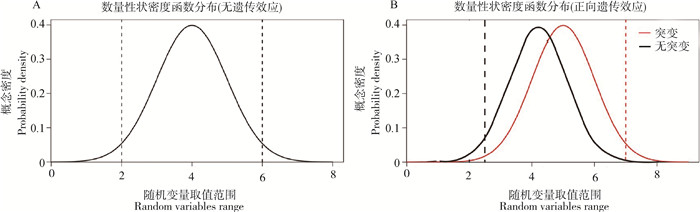
2 提高稀有变异检测效力的常用方法 2.1 极端表型抽样极端表型抽样是指抽取正态分布性状中表型值最大和最小的一部分个体。如图 1所示,当突变对表型有正向影响时,大多数位于右尾的个体会具有该变异,然而大多数位于左尾的个体没有该变异[23]。当突变对表型具有负向作用时,情况类似。因此,有效应的变异往往集中于具有极端表型的个体。为了提高样本携带稀有变异的概率,可以从群体中抽取表型值位于左右两尾的个体[24]。若样本足够大,可以从两尾各取5%的个体。极端表型抽样等同于为数量性状构建了病例-对照组,可以提高检测效力。对群体先进行表型值测量,再抽取极端表型个体进行芯片分型或测序,可以在保证相同检测效力的同时降低试验费用。在人类中,已经成功运用极端表型抽样的方法检测到与低密度脂蛋白胆固醇水平[25]、假单胞菌属初次感染时间[26]和二型糖尿病[27]等疾病相关的稀有变异。一项模拟研究也表明,运用极端表型抽样可以提高数量性状的稀有变异检测效力[28]。但是,抽样偏差会导致一类错误的增加,需要通过复杂的统计学方法去除[19, 23]。在畜禽的稀有变异关联分析中,极端表型抽样也是一种降低成本、提高效力的方法。
2.2 荟萃分析荟萃分析,是通过统计学手段将多个独立试验的信息合并分析。在全基因组关联分析中常用的Bonferroni检验太过保守,导致很多效应微小的位点没有通过显著性阈值[29],最直接的解决方法就是通过增大样本量以提高微小效应位点的检测效力。然而出于隐私等原因,绝大多数已发表论文的详细数据也不会公开。使用荟萃分析方法可以直接使用多个独立试验的结果,通过增加样本量提高关联效力并减少一类错误概率[30]。荟萃分析的优势在于只需要获得独立试验的P值或Z值,并且其检测效力与使用详细个体数据进行分析的结果差异不大[7]。
GWAS中常用的荟萃分析方法包括合并P值法、合并Z值法、固定效应模型、随机效应模型和贝叶斯法等。固定模型假设在不同独立试验中单个变异的效应大小都一致,随机效应模型允许效应大小不同,但随机模型对稀有变异的检测效果并不稳定[31]。稀有变异的荟萃分析可以简单的使用Fisher法合并P值或者Stouffer法合并Z值[32]。Fisher合并P值法和Stouffer合并Z值法分别是基于多个独立试验P值的负对数和Z值(估计效应值除以标准误)。这两个方法相似度很高,但合并Z值法的一个优势在于其允许标记在独立试验中的效应值方向不一致[30]。负担检验和方差组分检验合并Z值的方法是不同的,负担检验计算单个变异Z值的加权和,而方差组分检验计算单个变异Z值的平方加权和[33]。目前常用的稀有变异荟萃分析软件包括meta-SKAT[18]、seqMeta(https://cran.r-project.org/web/packages/seqMeta)、RAREMETAL[34]和RVTESTS[35]等。
独立稀有变异的研究往往在不同平台进行,不同平台包含不同的稀有变异覆盖率及基因分型错误率。这样的性质对结合多个不同类型的数据造成了巨大的困难。通过对数据进行合适的质控填充,再使用荟萃分析结合会是一个良好的解决办法[32]。
3 使用芯片数据研究稀有变异 3.1 基因型填充目前,制约畜禽中稀有变异研究最主要的因素就是测序成本太高。由于过去十年全基因组关联分析的迅速发展,累积了很多芯片数据。芯片是以单核苷酸多态性作为标记,主要针对常见变异而设计的,故检测稀有变异的效力很低。使用基因型填充技术将芯片数据向测序数据进行填充不仅可以充分利用现有资源,还可以降低试验成本。基因型填充技术一般是利用分型密度更高的样本和研究群体共同的单倍型块,对研究群体未分型的位点进行预测。常用的基因型填充软件包括IMPUTE[36]、Mach和Beagle。Beagle最新版本(V4.1) 运用pre-phasing填充方法[37],结合多线程并行运算以及内存优化算法,在内存消耗和计算速度上都优于另外两款软件[38]。在最近的一项对比试验中发现,运用pre-phasing填充法会在一定程度上降低填充准确性[39]。人类基因型填充的参考样本可从千人基因组计划(http://browser.1000genomes.org)和HapMap工程[40]两个平台获取参考基因组。目前在畜禽上可获得参考基因组的平台有千牛基因组计划(http://www.1000bullgenomes.com)。
实践证明,基因型填充的准确性与变异最小等位基因频率呈正相关,即会随最小等位基因频率降低而降低。这对稀有变异的填充造成了巨大的影响。针对这个问题主要有两个应对措施:其一,增加参考样本的数目以增加填充准确性;其二,通过对比亲代和子代同一位点的稀有变异,判断其填充的准确性。这两点在畜禽研究上短期内将很难实现,试验成本决定了无法增加参考样本数目,而且国内的畜禽系谱记录混乱,置信度低。
3.2 基于单倍型的稀有变异关联分析目前使用芯片数据研究稀有变异的方法主要有两种:一是利用测序数据对其进行填充后再对兴趣区域进行折叠;二是直接使用芯片数据将兴趣区域内的稀有单倍型与表型进行关联。前者已经在前文论述过了,尽管折叠方法有很大的前景,但存在缺陷:1.折叠方法使用来自二代测序的稀有多态单核苷酸,高深度测序成本太高,低深度测序可能会出现分型错误[41];2.不同的折叠区域会对折叠方法的结果造成相当大的影响;3.如何选择合适的变异进行折叠也没有标准化的定论[42];4.关联结果只能代表折叠区域与表型关联的显著性,无法对关联位点进行精细定位[43]。
稀有单倍型关联分析的优势在于,可以只使用GWAS数据,并且单倍型研究历程久,模型基础稳定[44-45];而且当基因分型密度不高或多个稀有变异呈独联体形式存在时,单倍型法比折叠方法更有效力[46]。由于存在以上优势,多个基于无关个体稀有单倍型关联分析的方法被提出:wei-SIMc-matching检验[47]、单倍型核关联检验(HKAT)[48]、贝叶斯分层广义线性模型(BhGLM)[49]、调节广义线性模型(rGLM)[50]、加权单倍型及填充为基础的检验(WHaIT)[51]、logistic贝叶斯LASSO(LBL)[52],hapKL[53]等。前3种方法可用于数量性状,这里做简单介绍。wei-SIMc-matching检验和单倍型核关联检验都使用期望最大算法推断单倍型,并且在广义线性模型框架下,将性状与单倍型进行回归。wei-SIMc-matching检验基于单倍型相似性的方法,通过构建相似性矩阵S,增加稀有单倍型相似性,降低常见单倍型相似性。单倍型核关联检验采用类似于SKAT的方法,使用B.E.Madsen等[54]提出的加权方法对稀有单倍型进行加权。以上两种方法都是通过构建score统计量进行检验,但这两种方法只能获得整个单倍型块的P值,无法获得特定单倍型的P值。BhGLM基于广义线性模型框架,假设回归系数服从独立学生t检验,使用EM算法对普通广义线性模型进行迭代加权,获得回归系数后验分布。BhGLM通过估计特定单倍型在个体内的数量,可以获得特定单倍型的效应值。
4 总结及应用展望在这篇综述中,笔者论述了稀有变异关联分析方法的研究进展及相关扩展。有一部分畜禽遗传育种工作者可能已经看到了稀有变异关联分析在畜禽上应用的可能。笔者尽力搜寻研究畜禽稀有变异关联分析的论文,目前只发现两篇:一篇研究的是奶牛复杂性状(产奶性状和繁殖性状)的缺失遗传力问题。基于3 311头荷斯坦奶牛,将50 k芯片先向777 k芯片填充再向测序数据填充并结合系谱数据区别稀有变异和填充错误。其试验结果发现,稀有变异可以解释小部分遗传变异,分别占乳脂产量、产奶量、乳蛋白产量和繁殖性状遗传变异的3%、0%、1%和14%[55]。另一篇是基于奶牛基因数据进行模拟研究,同样使用填充数据比较MONSTER[56](类似于SKAT-O,但使用家庭数据进行研究)和传统混合线性模型(GWAS中最常使用的方法)检测稀有变异的效力。结果如预期一样,传统的混合线性模型方法检测稀有变异效力较低[57]。尽管目前畜禽的稀有变异关联分析研究的数量屈指可数,但由于种种原因,在相同领域的研究,在畜禽中的研究总是滞后于人类,所以畜禽经济性状稀有变异关联分析应会有广阔的应用前景。
没有最好的模型,只有最合适的模型。只有在探索研究中不断掌握性状的遗传结构,才能设计出更符合复杂性状遗传结构的模型。现在机器学习的概念也逐渐进入畜禽遗传育种工作者的视线,人工神经网络[58-59]、核函数支持向量机[60-61]和随机森林[62-63]等研究的不断深入也为稀有变异关联分析提供可能更符合现实的非线性模型。在可以预见的未来,随着测序技术的发展与价格的下降以及更贴切模型的出现,畜禽经济性状关联分析的研究将不仅仅局限于常见变异。
| [1] |
张沅.
家畜育种学[M]. 北京: 中国农业出版社, 2001.
ZHANG Y. Animal breeding[M]. Beijing: China Agriculture Press, 2001. (in Chinese) |
| [2] | VISSCHER P M, BROWN M A, MCCARTHY M I, et al. Five years of GWAS discovery[J]. Am J Hum Genet, 2012, 90(1): 7–24. DOI: 10.1016/j.ajhg.2011.11.029 |
| [3] | SHARMA A, LEE J S, DANG C G, et al. Stories and challenges of genome wide association studies in livestock-a review[J]. Asian Austral J Anim, 2015, 28(10): 1371–1379. DOI: 10.5713/ajas.14.0715 |
| [4] | MANOLIO T A, COLLINS F S, COX N J, et al. Finding the missing heritability of complex diseases[J]. Nature, 2009, 461(7265): 747–753. DOI: 10.1038/nature08494 |
| [5] | GIBSON G. Rare and common variants:twenty arguments[J]. Nat Rev Genet, 2012, 13(2): 135–145. DOI: 10.1038/nrg3118 |
| [6] | LI Y L, FENG T, ZHU X. Detecting association with rare variants for common diseases using haplotype-based methods[J]. Stat Interface, 2011, 4(3): 273–283. DOI: 10.4310/SII.2011.v4.n3.a2 |
| [7] | LIU D J, PELOSO G M, ZHAN X W, et al. Meta-analysis of gene-level tests for rare variant association[J]. Nat Genet, 2014, 46(2): 200–204. |
| [8] | ROTH E M, MCKENNEY J M, HANOTIN C, et al. Atorvastatin with or without an antibody to PCSK9 in primary hypercholesterolemia[J]. New Engl J Med, 2012, 367(20): 1891–1900. DOI: 10.1056/NEJMoa1201832 |
| [9] | BAILÉN A R. Effect of a monoclonal antibody to PCSK9 on LDL cholesterol[J]. Rev Clin Esp, 2012, 212(7): 408–409. |
| [10] | DERING C, HEMMELMANN C, PUGH E, et al. Statistical analysis of rare sequence variants:an overview of collapsing methods[J]. Genet Epidemiol, 2011, 35(S1): S12–S17. DOI: 10.1002/gepi.v35.1s |
| [11] |
梁融, 张俊国, 卜涛, 等. 稀有变异的关联性研究统计方法[J]. 中华流行病学杂志, 2015, 36(8): 900–903.
LIANG R, ZHANG J G, BU T, et al. Review for the testing on rare-variants association with disease[J]. Chinese Journal of Epidemiology, 2015, 36(8): 900–903. (in Chinese) |
| [12] | WU M C, LEE S, CAI T X, et al. Rare-variant association testing for sequencing data with the sequence kernel association test[J]. Am J Hum Genet, 2011, 89(1): 82–93. DOI: 10.1016/j.ajhg.2011.05.029 |
| [13] | DUCHESNE P, DE MICHEAUX P L. Computing the distribution of quadratic forms:Further comparisons between the Liu-Tang-Zhang approximation and exact methods[J]. Comput Stat Data Anal, 2010, 54(4): 858–862. DOI: 10.1016/j.csda.2009.11.025 |
| [14] | LEE S, EMOND M J, BAMSHAD M J, et al. Optimal unified approach for rare-variant association testing with application to small-sample case-control whole-exome sequencing studies[J]. Am J Hum Genet, 2012, 91(2): 224–237. DOI: 10.1016/j.ajhg.2012.06.007 |
| [15] | LEE S, WU M C, LIN X H. Optimal tests for rare variant effects in sequencing association studies[J]. Biostatistics, 2012, 13(4): 762–775. DOI: 10.1093/biostatistics/kxs014 |
| [16] | IONITA-LAZA I, LEE S, MAKAROV V, et al. Sequence kernel association tests for the combined effect of rare and common variants[J]. Am J Hum Genet, 2013, 92(6): 841–853. DOI: 10.1016/j.ajhg.2013.04.015 |
| [17] | HAN F, PAN W. A data-adaptive sum test for disease association with multiple common or rare variants[J]. Hum Hered, 2010, 70(1): 42–54. DOI: 10.1159/000288704 |
| [18] | LEE S, TESLOVICH T M, BOEHNKE M, et al. General framework for meta-analysis of rare variants in sequencing association studies[J]. Am J Hum Genet, 2013, 93(1): 42–53. DOI: 10.1016/j.ajhg.2013.05.010 |
| [19] | BARNETT I J, LEE S, LIN X H. Detecting rare variant effects using extreme phenotype sampling in sequencing association studies[J]. Genet Epidemiol, 2013, 37(2): 142–151. DOI: 10.1002/gepi.21699 |
| [20] | WANG X F, LEE S, ZHU X F, et al. GEE-based SNP set association test for continuous and discrete traits in family-based association studies[J]. Genet Epidemiol, 2013, 37(8): 778–786. DOI: 10.1002/gepi.21763 |
| [21] | CHEN M H, YANG Q. GWAF:an R package for genome-wide association analyses with family data[J]. Bioinformatics, 2010, 26(4): 580–581. DOI: 10.1093/bioinformatics/btp710 |
| [22] | LIN X Y, LEE S, WU M C, et al. Test for rare variants by environment interactions in sequencing association studies[J]. Biometrics, 2016, 72(1): 156–164. DOI: 10.1111/biom.v72.1 |
| [23] | LIN D Y, ZENG D L, TANG Z Z. Quantitative trait analysis in sequencing studies under trait-dependent sampling[J]. Proc Natl Acad Sci U S A, 2013, 110(30): 12247–12252. DOI: 10.1073/pnas.1221713110 |
| [24] | ZUK O, SCHAFFNER S F, SAMOCHA K, et al. Searching for missing heritability:Designing rare variant association studies[J]. Proc Natl Acad Sci U S A, 2014, 111(4): E455–E464. DOI: 10.1073/pnas.1322563111 |
| [25] | LANGE L A, HU Y N, ZHANG H, et al. Whole-exome sequencing identifies rare and low-frequency coding variants associated with LDL cholesterol[J]. Am J Hum Genet, 2014, 94(2): 233–245. DOI: 10.1016/j.ajhg.2014.01.010 |
| [26] | EMOND M J, LOUIE T, EMERSON J, et al. Exome sequencing of extreme phenotypes identifies DCTN4 as a modifier of chronic Pseudomonas aeruginosa infection in cystic fibrosis[J]. Nat Genet, 2012, 44(8): 886–889. DOI: 10.1038/ng.2344 |
| [27] | FLANNICK J, THORLEIFSSON G, BEER N L, et al. Loss-of-function mutations in SLC30A8 protect against type 2 diabetes[J]. Nat Genet, 2014, 46(4): 357–363. DOI: 10.1038/ng.2915 |
| [28] | ZHOU Y J, WANG Y, CHEN L L. Detecting the common and individual effects of rare variants on quantitative traits by using extreme phenotype sampling[J]. Genes, 2016, 7(1): 2. |
| [29] | YANG J, BENYAMIN B, MCEVOY B P, et al. Common SNPs explain a large proportion of the heritability for human height[J]. Nat Genet, 2010, 42(7): 565–569. DOI: 10.1038/ng.608 |
| [30] | EVANGELOU E, IOANNIDIS J P A. Meta-analysis methods for genome-wide association studies and beyond[J]. Nat Rev Genet, 2013, 14(6): 379–389. DOI: 10.1038/nrg3472 |
| [31] | SHUSTER J J. Empirical versus natural weighting in random effects meta-analysis[J]. Stat Med, 2014, 33(7): 1260. DOI: 10.1002/sim.6031 |
| [32] | LEE S, ABECASIS G R, BOEHNKE M, et al. Rare-variant association analysis:study designs and statistical tests[J]. Am J Hum Genet, 2014, 95(1): 5–23. DOI: 10.1016/j.ajhg.2014.06.009 |
| [33] | PASANIUC B, PRICE A L. Dissecting the genetics of complex traits using summary association statistics[J]. Nat Rev Genet, 2017, 18(2): 117–127. |
| [34] | FENG S, LIU D J, ZHAN X W, et al. RAREMETAL:fast and powerful meta-analysis for rare variants[J]. Bioinformatics, 2014, 30(19): 2828–2829. DOI: 10.1093/bioinformatics/btu367 |
| [35] | ZHAN X W, HU Y N, LI B S, et al. RVTESTS:an efficient and comprehensive tool for rare variant association analysis using sequence data[J]. Bioinformatics, 2016, 32(9): 1423–1426. DOI: 10.1093/bioinformatics/btw079 |
| [36] | HOWIE B N, DONNELLY P, MARCHINI J. A flexible and accurate genotype imputation method for the next generation of genome-wide association studies[J]. PLoS Genet, 2009, 5(6): e1000529. DOI: 10.1371/journal.pgen.1000529 |
| [37] | HOWIE B, FUCHSBERGER C, STEPHENS M, et al. Fast and accurate genotype imputation in genome-wide association studies through pre-phasing[J]. Nat Genet, 2012, 44(8): 955–959. DOI: 10.1038/ng.2354 |
| [38] | BROWNING B L, BROWNING S R. Genotype imputation with millions of reference samples[J]. Am J Hum Genet, 2016, 98(1): 116–126. DOI: 10.1016/j.ajhg.2015.11.020 |
| [39] | ROSHYARA N R, HORN K, KIRSTEN H, et al. Comparing performance of modern genotype imputation methods in different ethnicities[J]. Sci Rep, 2016, 6: 34386. DOI: 10.1038/srep34386 |
| [40] | FRAZER K A, BALLINGER D G, COX D R, et al. A second generation human haplotype map of over 3.1 million SNPs[J]. Nature, 2007, 449(7164): 851–861. DOI: 10.1038/nature06258 |
| [41] | GOLDSTEIN D B, ALLEN A, KEEBLER J, et al. Sequencing studies in human genetics:design and interpretation[J]. Nat Rev Genet, 2013, 14(7): 460–470. DOI: 10.1038/nrg3455 |
| [42] | KOSMICKI J A, CHURCHHOUSE C L, RIVAS M A, et al. Discovery of rare variants for complex phenotypes[J]. Hum Genet, 2016, 135(6): 625–634. DOI: 10.1007/s00439-016-1679-1 |
| [43] | DO R, KATHIRESAN S, ABECASIS G R. Exome sequencing and complex disease:practical aspects of rare variant association studies[J]. Hum Mol Genet, 2012, 21(R1): R1–R9. DOI: 10.1093/hmg/dds387 |
| [44] | SCHAID D J, ROWLAND C M, TINES D E, et al. Score tests for association between traits and haplotypes when linkage phase is ambiguous[J]. Am J Hum Genet, 2002, 70(2): 425–434. DOI: 10.1086/338688 |
| [45] | SCHAID D J. Genetic epidemiology and haplotypes[J]. Genet Epidemiol, 2004, 27(4): 317–320. DOI: 10.1002/(ISSN)1098-2272 |
| [46] | WANG M, LIN S L. Detecting associations of rare variants with common diseases:collapsing or haplotyping?[J]. Brief Bioinform, 2015, 16(5): 759–768. DOI: 10.1093/bib/bbu050 |
| [47] | LIN W Y, YI N J, ZHI D G, et al. Haplotype-based methods for detecting uncommon causal variants with common SNPS[J]. Genet Epidemiol, 2012, 36(6): 572–582. DOI: 10.1002/gepi.21650 |
| [48] | LIN W Y, YI N J, LOU X Y, et al. Haplotype kernel association test as a powerful method to identify chromosomal regions harboring uncommon causal variants[J]. Genet Epidemiol, 2013, 37(6): 560–570. DOI: 10.1002/gepi.21740 |
| [49] | LI J, ZHANG K, YI N. A Bayesian hierarchical model for detecting haplotype-haplotype and haplotype-environment interactions in genetic association studies[J]. Hum Hered, 2011, 71(3): 148–160. DOI: 10.1159/000324841 |
| [50] | GUO W, LIN S L. Generalized linear modeling with regularization for detecting common disease rare haplotype association[J]. Genet Epidemiol, 2009, 33(4): 308–316. DOI: 10.1002/gepi.v33:4 |
| [51] | LI Y, BYRNES A E, LI M Y. To identify associations with rare variants, just WHaIT weighted haplotype and imputation-based tests[J]. Am J Hum Genet, 2010, 87(5): 728–735. DOI: 10.1016/j.ajhg.2010.10.014 |
| [52] | BISWAS S, LIN S L. Logistic Bayesian LASSO for identifying association with rare haplotypes and application to age-related macular degeneration[J]. Biometrics, 2012, 68(2): 587–597. DOI: 10.1111/biom.2012.68.issue-2 |
| [53] | LIN S. Kullback-Leibler divergence for detection of rare haplotype common disease association[J]. Eur J Hum Genet, 2015, 23(11): 1558–1565. DOI: 10.1038/ejhg.2015.25 |
| [54] | MADSEN B E, BROWNING S R. A groupwise association test for rare mutations using a weighted sum statistic[J]. PLoS Genet, 2009, 5(e10003842). |
| [55] | GONZALEZ-RECIO O, DAETWYLER H D, MACLEOD I M, et al. Rare variants in transcript and potential regulatory regions explain a small percentage of the missing heritability of complex traits in cattle[J]. PLoS One, 2015, 10(12): e0143945. DOI: 10.1371/journal.pone.0143945 |
| [56] | JIANG D, MCPEEK M S. Robust rare variant association testing for quantitative traits in samples with related individuals[J]. Genet Epidemiol, 2014, 38(1): 10–20. DOI: 10.1002/gepi.21775 |
| [57] | ZHANG Q Q, GULDBRANDTSEN B, CALUS M P L, et al. Comparison of gene-based rare variant association mapping methods for quantitative traits in a bovine population with complex familial relationships[J]. Genet Sel Evol, 2016, 48(1): 60. DOI: 10.1186/s12711-016-0238-5 |
| [58] | CURTIS D, NORTH B V, SHAM P C. Use of an artificial neural network to detect association between a disease and multiple marker genotypes[J]. Ann Hum Genet, 2001, 65(1): 95–107. DOI: 10.1046/j.1469-1809.2001.6510095.x |
| [59] | MOTSINGER-REIF A A, DUDEK S M, HAHN L W, et al. Comparison of approaches for machine-learning optimization of neural networks for detecting gene-gene interactions in genetic epidemiology[J]. Genet Epidemiol, 2008, 32(4): 325–340. DOI: 10.1002/(ISSN)1098-2272 |
| [60] | KARATZOGLOU A, SMOLA A, HORNIK K, et al. Kernlab-An S4 package for kernel methods in R[J]. J Stat Softw, 2004, 11(9). DOI: 10.18637/jss.v011.i09 |
| [61] | XU S Z, XU Y, GONG L, et al. Metabolomic prediction of yield in hybrid rice[J]. Plant J, 2016, 88(2): 219–227. DOI: 10.1111/tpj.2016.88.issue-2 |
| [62] | BREIMAN L. Random forests[J]. Mach Learn, 2001, 45(1): 5–32. DOI: 10.1023/A:1010933404324 |
| [63] | ACHARJEE A, KLOOSTERMAN B, DE VOS R C H, et al. Data integration and network reconstruction with-omics data using Random Forest regression in potato[J]. Anal Chim Acta, 2011, 705(1-2): 56–63. DOI: 10.1016/j.aca.2011.03.050 |