纯血马是世界现代马业最主要的赛事用马,以短距离速度快,爆发力强等特点闻名于世,其1 000 m平地赛最佳纪录为53″7,1 600 m为1′31″8,2 400 m为2′23″。然而国际例行的纯血马平地赛距离通常为1 000~3 200 m,最长不超过8 000 m,否则可能导致纯血马骨折、肺出血等无法医治的运动损伤[1]。在2005年第十届全国运动会速度赛马中,12 000 m的超长赛事造成了参赛马匹赛后1死11伤的惨剧。而4年后的第十一届全运会速度赛,参赛的16匹纯血马中有7匹中途退赛,1匹赛后死亡,仅有9匹勉强完成赛事。以上事例反映出我国马业与世界水平尚存在差距,一概西化,沿用他国标准来发展我国现代马业的思路是行不通的。
蒙古马是我国分布最广的典型地方马种[2],经长期自然和人工选择形成抗严寒、耐粗饲、抗病力强、持久性好等特性。传统蒙古族那达慕大会中赛马距离通常为30~100 km,经训练的蒙古马能一气呵成,中途不休息完成比赛,且极少有严重运动性损伤出现。只有在深入了解我国地方马种优良性能的基础上,结合现代生物学技术优化种群,才能逐渐摸索出符合我国现今国情的现代马业发展之路,加速传统马业向现代马业的转变的同时,达到保护地方良种马的目的。
1 材料与方法试验所用肌肉样品供体为6匹5岁成年雌性健康蒙古马,来自于内蒙古自治区达尔罕茂明安联合旗(110°43'N,41°7'E,平均海拔1 367 m)同一牧户,以保证样品供体遗传背景与成长环境相似。
1.1 试验用马的训练试验用马遵循少量多餐,训练结束后1 h内不给水草,饱食后2 h内不训练的原则。训练期间每日饲喂5次青干草,白天4次,夜间补饲1次,草量以半小时内吃完无剩余为准;每日饲喂1次精料,精料成分为玉米、燕麦与麸皮,按4:4:2加水搅拌后供给,隔天在精料中加入葵花籽和食用盐。自由饮水,且频繁更换以保持新鲜;马厩内长期挂盐砖和糖砖供其自由舔舐,以及时补充训练流失的微量元素。
训练在一个直径30 m的圆形调教圈中进行,为避免高负荷训练对马造成急性运动损伤,将4个月的训练期分为3个阶段,循序渐进加强训练负荷强度。当日训练内容不要求一次性不间断完成,可依据马匹体力与配合情况分段训练。后期训练负荷增大,马匹因倦怠不再奔跑快步配合完成当日的训练内容,则采用牵溜慢步的形式补充完成当日训练计划[3]。
第1阶段持续15 d,每5 d为1个周期,起始训练距离为8 km,每个周期中前4 d每日训练距离以2 km递增,即第2天10 km,第3天12 km,第4天14 km,第5天休息,下一周期起始距离回到8 km,如此循环。
第2阶段持续92 d,每4天为1个周期,起始训练距离为15 km,每个周期中前3 d每日训练距离以5 km递增,即第2天20 km,第2天25 km,第四天休息。如此循环。
第3阶段,在训练期最后的两周里,尽可能延长训练时间和训练距离,训练起始距离为20 km,每日训练距离以10 km递增,即第2天30 km,第3天40 km,第4天50 km,第5、6天休息并提高日粮中精饲料比例。该阶段结束后,静养3日,采集肌肉样品。
1.2 样本采集分别在训练前和训练后马匹静止时期采集肌肉样品。
训练前空腹状态下,保定并注射局部麻醉剂,从臀中肌采取试验所需肌肉样品[4],肌肉组织离体后,用蒸馏水反复冲洗,立即投入液氮冷冻,用于提取总RNA与后续分析和备份[5]。高负荷训练4个月,同上述方法采集样品。后用TRIzol法提取肌肉总RNA。
1.3 转录组高通量测序分析样品提取总RNA后,用Oligo(dT)[6]磁珠富集[7]mRNA,用二价阳离子结合Illumina缓冲液将mRNA打碎片段化[8],在随机六聚体[9]的作用下,以短链mRNA为模板,合成cDNA第一链,后加入buffer缓冲液、RNase H和DNA聚合酶Ⅰ合成cDNA的第二链,在加EB缓冲液洗脱之前,还要经过Qia Quick PCR试剂盒的纯化过程[10],后经过末端修复等修饰过程,再经琼脂糖凝胶电泳回收目的片段并进行PCR扩增,完成整个文库制备流程[11]。
构建好的文库用Illumina HiSeqTM 2000测序平台,采用双末端测序(Paired-end)法[12],分别对训练前后两个文库进行测序。获得原始序列后参考对比马基因组(Equus caballus Ensemble release 76)[13]进行生物信息学分析。并利用TopHat[14]、SOAP2[15]等程序对转录组数据库进行比对注释[16],利用Cuffdiff[17]计算基因或转录本的表达量FPKM (Fragments Per Kilobase of transcript per Million mapped reads)值,并检测是否存在差异表达[18]。得到差异表达基因后,用GO seq[19]软件包,以P<0.05为筛选条件,对训练前后2个数据库中表达量有显著差异的基因进行Gene Ontology富集分析,并运用KOBAS软件对差异基因进行KEGG Pathway(Kyoto Encyclopedia of Genes and Genomes)[20]信号通路富集分析。
2 结果 2.1 转录组测序数据分析将clean reads比对到马的参考基因组,在训练前后2个文库中分别有88.13%和89.34%的序列能唯一比对到基因组(表 1)。
|
|
表 1 clean Reads比对到参考基因组 Table 1 The clean Reads alignment to the reference genome |
样品中鉴定出的已知基因有15 662个,占到基因组参考序列(20 270) 的77.27%,同时发现1 064个新基因。
对于训练前后两个文库中表达量发生变化的基因,用P<0.05且|log2FC|>1作为阈值筛选差异基因。共筛选到1 102个发生显著变化的基因,其中训练后发生显著下调的基因299个,显著上调的基因有803个(图 1)。表 2列出了训练前后差异显著(P<0.05) 且表达量较高的前30个基因。
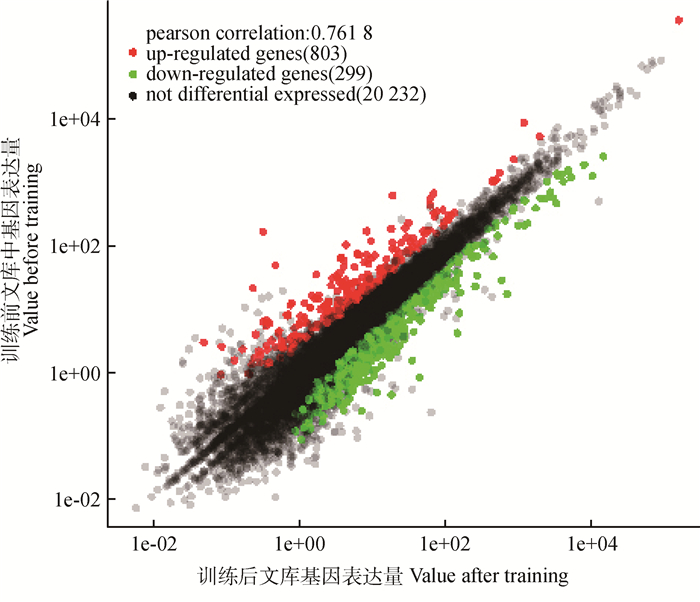
|
图中红点表示显著上调基因,绿点表示显著下调基因,黑点表示训练前后表达量差异不显著基因 The red spots mean up-regulated genes, the green spots mean down-regulated genes, the black spots mean not differentially expressed genes 图 1 差异基因统计图 Figure 1 Scatter plot of differentially expressed genes |
|
|
表 2 训练前后差异显著且表达量较高的前30个基因 Table 2 The top 30 significant differentially expressed genes before and after training |
为验证转录组试验数据的准确性,从测序结果中随机选取10个蒙古马在训练前后差异表达的基因,以β-actin作为内参基因绘制标准曲线,采用2-△△Ct定量分析对蒙古马训练前后差异表达基因的表达量进行分析。结果显示,这些差异基因的表达模式与转录组数据结果具有相同变化趋势,佐证了转录组测序结果的可靠性。
2.2 差异基因的Gene Ontology功能富集分析将差异基因向GO数据库各条目映射,确定差异表达基因所参与的代谢通路,以此了解在训练过程中各基因发挥的生物学功能[21]。1 102个差异表达基因共被注释到3 398条GO term上,其中生物学过程(Biological process)2 621条,细胞组分(Cellular component)262条,分子功能(Molecular function)515条。表 3给出了差异表达基因所参与的生物学过程GO条目。图 2列出了部分富集到基因最多的GO term。
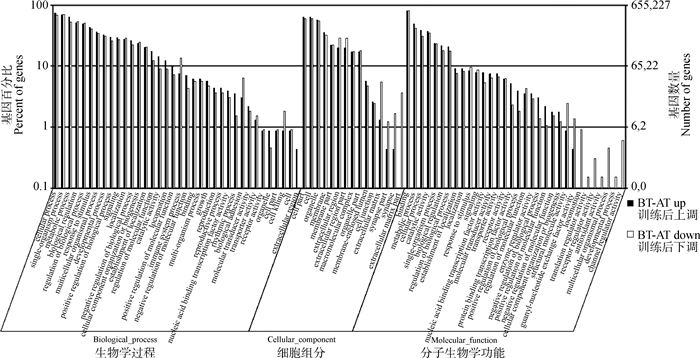
|
图 2 Gene ontology功能富集分析 Figure 2 Gene ontology enrichment analysis |
|
|
表 3 差异表达基因所参与的生物学过程GO条目 Table 3 The GO terms enriched for the differentially expressed genes in biological process |
结果显示(表 3),差异基因所参与调控的运动学过程包括:肌肉结构发育、组织发育、心血管系统发育、循环系统发育、肌肉组织发育、横纹肌发育、肌肉器官发育、血管系统发育、脉管系统发育、肌肉收缩、肌肉细胞分化等。被富集到上述过程中的差异基因可能在蒙古马适应高负荷训练的过程中起到调控作用,诸如MYL2、ANKRD2、ACTC1、CHRND、MYH11、CHRNB1、CHRNA1、MHC-1等基因也已在其他研究文献中被证实参与了骨骼肌发育、骨骼肌分化、骨骼肌收缩等生理过程,这些筛选得到的差异表达基因也成为后续深入研究蒙古马耐力性能的目标候选基因。
2.3 差异基因KEGG通路分析对1 102个差异表达基因进行KEGG通路分析,结果显示,差异表达基因被富集到扩张型心肌病、肥厚型心肌病、心肌收缩、钙信号通路、肌动蛋白骨架调控等193条与运动生理过程相关的通路上,表 4列出了前30条显著富集的KEGG通路。被富集到上述通路中的基因如:ACTC1、ATP1A1、 ATP2A2、 ATP2A3、 ATP1B2、 MYL2、 MYL3、 MYH1、 MYH3、 MYH11、 MYH13、 TPM2、 TPM3、 TNNC1、 TNNI3等已在国内外诸多研究中被证实与运动生理代谢过程显著相关。
|
|
表 4 差异表达基因所参与的KEGG通路 Table 4 The KEGG pathways enriched for the differentially expressed genes |
设计训练方案的目标是对肌肉、骨骼和心进行持续刺激,使机体产生适应性。肌细胞对刺激的反应过程包括接受刺激、疲劳、补偿、过度补偿与适应5个阶段,训练方案制定的关键在于强度的把握。首先要保证刺激足够强,使肌肉产生过度补偿和适应性代偿,同时又要避免过度刺激导致肌肉疲劳,致使肌肉进入自主保护状态,过激的刺激非但不能提高机体适应性,相反会增加因肌纤维损伤过重而受伤的风险,故而在高负荷训练中要考虑训练间隔与训练强度,提供足够的过度补偿和适应时间。科学合理的训练周期使肌纤维暂时性损伤,当机体得到充分的休息后,受损的肌纤维会代偿积累更多的能量储备,以迎接下一个训练周期,当肌肉适应了该周期的压力刺激后,再将刺激水平提高,如此反复,机体将进入循序渐进、良性积极的训练周期。
用Illumina测序技术对蒙古马高负荷运动训练前后肌肉样品文库进行构建测序结果表明,训练对某些运动生理代谢过程产生了一定影响,表现为某一基因在训练前后两文库中表达量的差异。造成表达差异的根本原因极有可能是持续数周的高负荷训练使肌细胞在转录水平积累产生了适应性代偿,致使肌细胞再次进入发育、分化的生长周期,进而诸多差异表达基因被显著富集于肌肉结构发育、肌肉组织发育、肌肉器官发育、肌肉收缩、肌肉细胞分化等运动学通路上。同时参与调控运动学相关代谢过程的基因在转录水平表达量也发生相应变化,如心血管系统发育、循环系统发育、脉管形态发育、器官发育等成为富集差异显著基因最多的代谢通路。在训练前后表达量发生显著变化的基因很多已在国内外研究中被证实与运动代谢显著相关,如:ACTC1、MYL2、MYL3、MYH1、MYH3、MYH11、MYH13是真核细胞需要控制并维持其形态及内部构造的重要物质,通过重组结构,枝状肌动蛋白网能在同样的组装动力学条件下支持不同的运动速度[22]。ATP1A1、ATP2A2、ATP2A3、ATP1B2称为三磷酸腺苷酶,是一类能将三磷酸腺苷(ATP)催化水解为二磷酸腺苷(ADP)和磷酸根离子的酶,同时伴随释放能量。在大多数情况下,能量可以通过传递而被用于驱动另一个需要能量的化学反应,这一过程被所有已知的生命形式广泛利用[23]。TNNC1与TNNI3是肌肉的主要调节蛋白质,直接参与钙所控制的肌肉收缩。钙从肌质网(Sarcoplasmic reticulum)释放出来,与肌钙蛋白结合发生结构改变,被抑制的肌动蛋白和肌球蛋白相互作用,肌肉发生收缩[24]。在KEGG通路分析结果中,基因被显著富集到与心血管疾病相关代谢通路上,如:扩张型心肌病,肥大型心肌病,病毒性心肌炎等。推测是由于本试验中蒙古马心在训练刺激后产生适应性心肌增大,心室容积增大,心壁增厚,并伴随毛细血管密度的增加,心肌增大致使心容量增加,使每次心搏出的血量增加,R.Manickam等研究获得了相似的试验结果[25],为此结果提供了佐证。
N.J.Bryant等认为长期耐力训练本身是一种非常态刺激[26],不断增强的有氧应激在长期耐力运动中可最终导致肌纤维疲劳、损伤及物理性能下降,并伴随着炎症反应和免疫系统的应答反应。故而被显著富集于运动学通路上的差异基因同时参与调控代谢疾病,如张万坡等利用细菌双杂交系统, 研究了禽流感病毒(Avian influenza virus, AIV)非结构蛋白(Nonstructurol1protein, NS1) 和鸡淋巴细胞抗原复合体(LY6E)蛋白之间的相互作用,表明AIV NS1蛋白和鸡LY6E蛋白之间存在相互作用, 为AIV致病机制的研究提供了新的资料[26];BCL9是近年来Wnt信号传导通路上发现的新致癌基因, 其在多种Wnt信号通路失调肿瘤中的异常表达表明BCL9与肿瘤的发生、发展有密切关系[27];NFATc4可增强心肌细胞核内PPARα与NFATc4之间的相互作用, 并减弱p65-NFκB与NFATc4之间的相互结合, 进而降低NFATc4在BNP启动子上的DNA结合活性, 这可能是其抑制AngⅡ诱导的心肌肥大的重要机制[28];MHC-1,主要组织相容性复合体(Major histocompatibility complex, MHC)是存在于细胞表面的一类糖蛋白, 在脊椎动物免疫反应中起着重要的作用。MHC由多基因家族所编码, 其编码产物能够识别并结合病原体或寄生虫后将它们呈递给淋巴T细胞进而引起免疫应答反应[29];以往许多研究认为, 运动训练能促使MHC亚型之间的转化, 但是结论并不一致[30];MYL2(肌球蛋白轻链2),肌球蛋白作为一种超家族蛋白质, 是细胞骨架的重要组成部分, 通过各种调节因子对其轻链磷酸化和去磷酸化的调节, 参与了几乎所有的细胞生理、病理过程, 尤其在肿瘤细胞的增殖、侵袭和迁移中发挥重要作用[31];ACTC1是先天性心脏病的候选基因, 且与人类先天性心脏病房间隔缺损有关[32-33]。这些参与调控肌肉生长、心肌发育、脉管系统运动生理学的相关基因,同时参与调控着某些疾病的免疫应答机制,这可能是蒙古马同时具备强耐力和强抗病性的原因。
4 结论本研究应用RNA-Seq技术对蒙古马高负荷运动前后进行了转录组学比对分析,共筛选出1 102个显著差异表达基因,同时对这些差异表达基因进行生物学功能分析,其结果补充了蒙古马这一强耐力中国特色马种在运动前后转录组差异研究的空白,为进一步揭示蒙古马基本运动学分子机制,以及蒙古马具备超强耐力的特性提供分子遗传学理论基础。
| [1] |
吴克亮, 吴常信. 马科学研究动态和马业发展[J]. 畜牧兽医学报, 2005, 36(4): 412–416.
WU K L, WU C X. Advanced research on equine science and horse industry[J]. Acta Veterinaria et Zootechnica Sinica, 2005, 36(4): 412–416. (in Chinese) |
| [2] |
芒来. 蒙古马的历史与展望[J]. 马业杂志, 2002(4): 14–17.
MANG L. History and outlook of the Mongolian horse[J]. Journal of Horse Industry, 2002(4): 14–17. (in Chinese) |
| [3] | MOSHER D S, QUIGNON P, BUSTAMANTE C D, et al. A mutation in the myostatin gene increases muscle mass and enhances racing performance in heterozygote dogs[J]. PLoS Genet, 2007, 3(5): e79. DOI: 10.1371/journal.pgen.0030079 |
| [4] | ZHANG X M, ZHAO L, LARSON-RABIN Z, et al. De novo sequencing and characterization of the floral transcriptome of Dendrocalamus latiflorus (Poaceae: Bambusoideae)[J]. PLoS One, 2012, 7(8): e42082. DOI: 10.1371/journal.pone.0042082 |
| [5] | VARSHNEY R K, SIGMUND R, BÖRNER A, et al. Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice[J]. Plant Sci, 2005, 168(1): 195–202. DOI: 10.1016/j.plantsci.2004.08.001 |
| [6] | CÁNOVAS A, RINCON G, ISLAS-TREJO A, et al. SNP discovery in the bovine milk transcriptome using RNA-Seq technology[J]. Mamm Genome, 2010, 21(11-12): 592–598. DOI: 10.1007/s00335-010-9297-z |
| [7] | LI X Y, SUN H Y, PEI J B, et al. De novo sequencing and comparative analysis of the blueberry transcriptome to discover putative genes related to antioxidants[J]. Gene, 2012, 511(1): 54–61. DOI: 10.1016/j.gene.2012.09.021 |
| [8] | GOECKS J, NEKRUTENKO A, TAYLOR J, et al. GALAXY: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences[J]. Genome Biol, 2010, 11(8): R86. DOI: 10.1186/gb-2010-11-8-r86 |
| [9] | JAILLON O, AURY J M, NOEL B, et al. The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla[J]. Nature, 2007, 449(7161): 463–467. DOI: 10.1038/nature06148 |
| [10] | CHEN Q M, WANG H, ZENG Y Q, et al. Developmental changes and effect on intramuscular fat content of H-FABP and A-FABP mRNA expression in pigs[J]. J Appl Genet, 2013, 54(1): 119–123. DOI: 10.1007/s13353-012-0122-0 |
| [11] | BONNET A, BEVILACQUA C, BENNE F, et al. Transcriptome profiling of sheep granulosa cells and oocytes during early follicular development obtained by Laser Capture Microdissection[J]. BMC Genomics, 2011, 12: 417. DOI: 10.1186/1471-2164-12-417 |
| [12] | MILLER J R, KOREN S, SUTTON G. Assembly algorithms for next generation sequencing data[J]. Genomics, 2010, 95(6): 315–327. DOI: 10.1016/j.ygeno.2010.03.001 |
| [13] | http://asia.ensembl.org/Equus_caballus/Info/Index. |
| [14] | ERLICH Y, MITRA P P, DELABASTIDE M, et al. Alta-cyclic: a self-optimizing base caller for next-generation sequencing[J]. Nat Methods, 2008, 5(8): 679–682. DOI: 10.1038/nmeth.1230 |
| [15] | ESTEVE-CODINA A, KOFLER R, PALMIERI N, et al. Exploring the gonad transcriptome of two extreme male pigs with RNA-seq[J]. BMC Genomics, 2011, 12: 552. DOI: 10.1186/1471-2164-12-552 |
| [16] | KANG X L, LIU G, LIU Y F, et al. Transcriptome profile at different physiological stages reveals potential mode for curly fleece in Chinese Tan sheep[J]. PLoS One, 2013, 8(8): e71763. DOI: 10.1371/journal.pone.0071763 |
| [17] | LOBJOIS V, LIAUBET L, SANCRISTOBAL M, et al. A muscle transcriptome analysis identifies positional candidate genes for a complex trait in pig[J]. Anim Genet, 2008, 39(2): 147–162. DOI: 10.1111/j.1365-2052.2007.01695.x |
| [18] | ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Genome Biol, 2010, 11(10): R106. DOI: 10.1186/gb-2010-11-10-r106 |
| [19] | NIELSEN M, HANSEN J H, HEDEGAARD J, et al. microRNA identity and abundance in porcine skeletal muscles determined by deep sequencing[J]. Anim Genet, 2010, 41(2): 159–168. DOI: 10.1111/age.2010.41.issue-2 |
| [20] | KEGG: Kyoto encyclopedia of genes and genomes[EB/OL]. http://www.genome.jp/kegg/. |
| [21] | DUTTA S, KUMAWAT G, SINGH B P, et al. Development of genic-SSR markers by deep transcriptome sequencing in pigeonpea[Cajanus cajan (L.) Millspaugh][J]. BMC Plant Biol, 2011, 11: 17. DOI: 10.1186/1471-2229-11-17 |
| [22] | ZORZATO F, FUJII J, OTSU K, et al. Molecular cloning of cDNA encoding human and rabbit forms of the Ca2+ release channel (ryanodine receptor) of skeletal muscle sarcoplasmic reticulum[J]. J Biol Chem, 1990, 265(4): 2244–2256. |
| [23] | GAJDOSIK R L. Passive extensibility of skeletal muscle: review of the literature with clinical implications[J]. Clin Biomech, 2001, 16(2): 87–101. DOI: 10.1016/S0268-0033(00)00061-9 |
| [24] | SCHRÖDER W, KLOSTERMANN A, DISTL O. Candidate genes for physical performance in the horse[J]. Vet J, 2011, 190(1): 39–48. DOI: 10.1016/j.tvjl.2010.09.029 |
| [25] | MANICKAM R, PENA R N, WHITELAW C B A. Mammary gland differentiation inversely correlates with GDF-8 expression[J]. Mol Reprod Dev, 2008, 75(12): 1783–1788. DOI: 10.1002/mrd.v75:12 |
| [26] | BRYANT N J, GOVERS R, JAMES D E. Regulated transported transport of the glucose transporter GLUT4[J]. Nat Rev Mol Cell Biol, 2002, 3(4): 267–277. DOI: 10.1038/nrm782 |
| [27] |
张万坡, 田文霞, 李建丽, 等. 禽流感病毒NS1蛋白与鸡LY6E蛋白间的相互作用[J]. 中国兽医学报, 2008, 28(6): 627–630, 645.
ZHANG W P, TIAN W X, LI J L, et al. Interaction between nonstructural (NS1) protein of avian influenza virus and LY6 E proteins of chicken[J]. Chinese Journal of Veterinary Science, 2008, 28(6): 627–630, 645. (in Chinese) |
| [28] |
吴帅, 简斌. BCL9与肿瘤及肿瘤靶点治疗研究[J]. 生命科学研究, 2013, 17(1): 86–89.
WU S, JIAN B. Research on BCL9 with tumor and tumor target treatment[J]. Life Science Research, 2013, 17(1): 86–89. (in Chinese) |
| [29] |
邹剑, 周后凤, 先志伟, 等. 激活PPARα表达对AngⅡ诱导的心肌细胞肥大及NFATc4与p65-NFκB相互作用的影响[J]. 中国病理生理杂志, 2014, 30(6): 1017–1022.
ZOU J, ZHOU H F, XIAN Z W, et al. Effects of PPARα activation on AngⅡ-induced cardiomyocyte hypertro-phy and interaction of NFATc4 with p65-NFκB[J]. Chinese Journal of Pathophysiology, 2014, 30(6): 1017–1022. (in Chinese) |
| [30] |
王永珍. 峨眉树蛙和斑腿树蛙MHCⅠ类基因的多态性及其进化[D]. 武汉: 华中师范大学, 2012.
WANG Y Z. Polymorphism and evolution of MHC class Ⅰ genes of Rhscophorus omeimontis and Rhscophorus megacephalus[D]. Wuhan: Central China Normal University, 2012. (in Chinese) (in Chinese) |
| [31] |
孟祥阁. 抗阻、耐力及混合运动对C57小鼠股四头肌MHC各亚型基因表达的影响[D]. 上海: 华东师范大学, 2010.
MENG X G. The mechanisms and the recovery process of exercise-induced fatigue[D]. Shanghai: East China Normal University, 2010. (in Chinese) (in Chinese) |
| [32] |
王丛阳, 王辉. 肌球蛋白轻链的调节及其对肿瘤细胞增殖、转移的影响[J]. 医学综述, 2010, 16(21): 3251–3254.
WANG C Y, WANG H. Regulation of myosin light chain and its effect on tumor proliferation and metastas[J]. Medical Recapitulate, 2010, 16(21): 3251–3254. DOI: 10.3969/j.issn.1006-2084.2010.21.018 (in Chinese) |
| [33] |
李航, 王彬彬, 高秉仁, 等. 一个新的ACTC1基因5'端剪切位点突变可能在先天性心脏病室间隔缺损发病中起重要作用[J]. 中国组织工程研究, 2012, 16(11): 2087–2090.
LI H, WANG B B, GAO B R, et al. A novel 5' splice site mutation in ACTC1 gene may play an important role in congenital ventricular septal defect[J]. Chinese Journal of Tissue Engineering Research, 2012, 16(11): 2087–2090. DOI: 10.3969/j.issn.1673-8225.2012.11.044 (in Chinese) |



