哺乳动物卵母细胞体外成熟是一项重要的胚胎工程技术,通过抽取卵巢有腔卵泡中的卵母细胞,体外成熟培养后,为动物克隆及转基因动物提供丰富的卵源。尽管人们在牛卵母细胞的体外成熟方面作了大量工作,但是关于卵母细胞体外成熟的分子机制方面尚有待于进一步研究。在牛的未成熟卵的体外培养研究中,普遍认为保留卵母细胞周围的卵丘细胞能明显提高卵母细胞的成熟率和受精率[1]。卵丘细胞的存在与否严重影响卵母细胞的体外成熟率及受精率[2]。牛卵母细胞体外成熟前、体外受精前、体外受精后7 h、体外受精后48 h去除卵丘细胞并进行后续培养,4种处理方式的卵母细胞成熟率依次为26%、93%、93%、96%,受精率依次为9%、58%、71%、92%,囊胚率依次为1%、6%、12%、19%,结果表明, 体外受精后48 h去除卵丘细胞的卵母细胞成熟率和囊胚率最高,由此表明,卵丘细胞在卵母细胞的体外成熟及受精过程中发挥着重要作用[3]。
miRNA是一类长约22 nt的非编码RNA,miRNA通过与靶标基因特异性的碱基互补配对,引起靶mRNA的降解或者抑制其翻译,从转录后水平调节基因的表达,从而发挥生物学功能。小鼠MⅡ期卵母细胞对应的颗粒细胞中,let-7b、let-7c、miR-27a、miR-322的表达量显著高于MⅠ颗粒细胞中的表达量,在腔前卵泡的体外培养液中,加入化学合成的miR-27a mimics,相对于对照组显著降低了卵母细胞的成熟率(P=0.042),而分别添加化学合成的let-7c、miR-27a、miR-322 inhibitors,发现与对照组比均显著提高了卵母细胞的成熟率(P<0.001,P=0.013,P=0.021),结果表明,miRNAs通过在颗粒细胞中起作用进而影响卵母细胞的成熟效率[4]。在对多囊性卵巢综合征(PCOS)患者的研究中发现,miR-509-3p在卵丘细胞中通过抑制MAP3K8基因的表达进而增加雌二醇的分泌,表明miRNAs在卵丘细胞中发挥着重要作用[5]。C.J.Li等研究发现添加脑源神经营养因子(BDNF)可能通过降低miR-205来调节卵丘扩展基因PTX3的表达水平,进而提高卵母细胞的成熟率[6]。miR-378可以抑制卵丘细胞中芳香化酶的表达,导致雌激素的表达水平显著降低,从而降低卵母细胞的成熟效率[7]。在腔前卵泡~早期卵泡的转变过程中,miR-224可能参与TGF-β1介导的卵泡颗粒细胞增殖和雌二醇的产生,且miR-224可能通过靶向于PTX3基因从而调控卵丘细胞的扩展来影响卵泡排卵和后期的胚胎发育[8]。综上表明,卵丘细胞中miRNAs的表达影响着卵母细胞的成熟及其后续胚胎的发育。
对miRNAs的研究有助于人类更好地了解miRNAs在繁殖领域中发挥的作用。研究发现miRNAs主要是通过调控其靶基因的表达来影响卵丘细胞的增殖、扩展以及相关激素的合成进而在卵母细胞的成熟过程中发挥作用,但是相关研究仅见少量报道,并没有对卵母细胞成熟过程中卵丘细胞中的miRNAs表达情况进行系统的研究。因此,本研究拟通过对水牛卵母细胞成熟过程中GV/MⅡ两个时期对应的卵丘细胞小RNA进行Solexa测序,并进行Stem-loop RT-PCR验证和生物信息学分析,挖掘两个时期卵丘细胞miRNAs的差异表达情况,为进一步研究卵母细胞成熟过程中卵丘细胞发挥的重要作用提供科学依据。
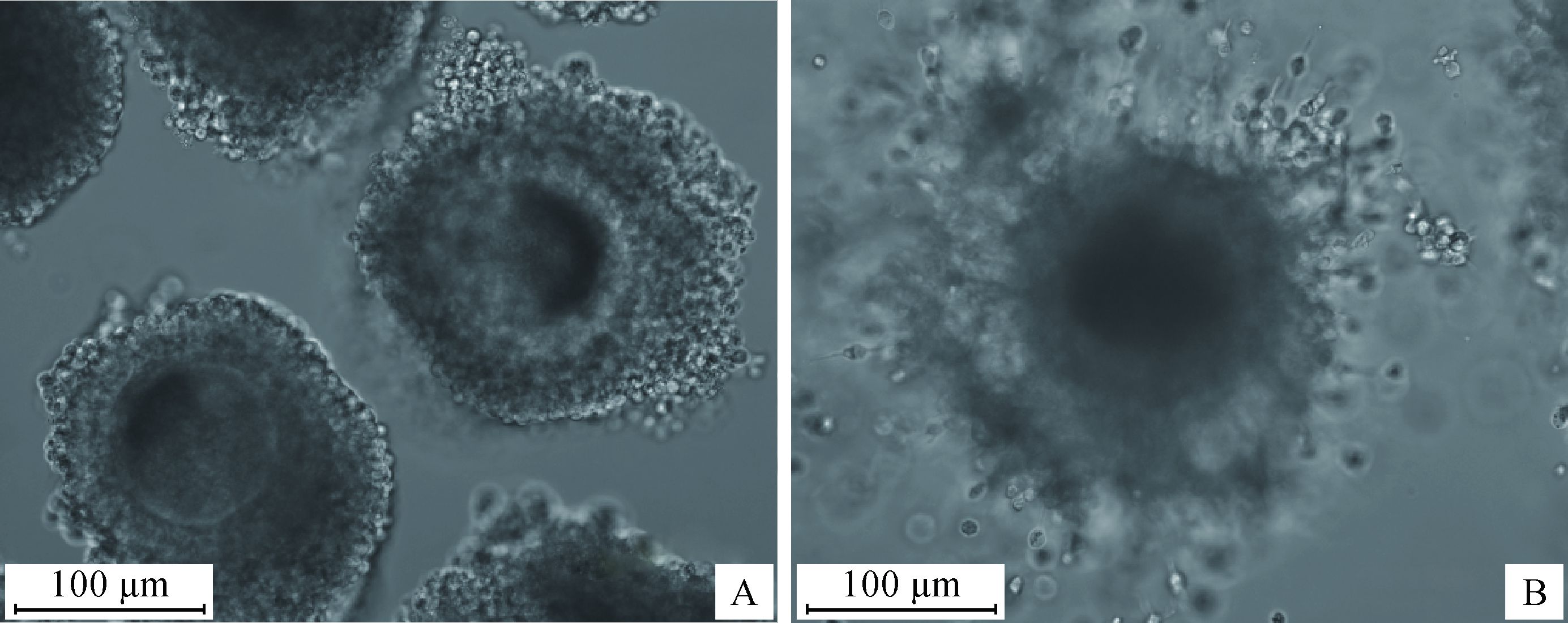
1 材料与方法 1.1 试验材料从南宁市屠宰场获得的卵巢,放入盛有37 ℃生理盐水的保温瓶中,4 h内送到实验室。用75%乙醇快速清洗卵巢表面,用37 ℃生理盐水清洗卵巢。随后用10 mL注射器选取直径为2~6 mm的卵泡抽取卵泡液和卵丘-卵母细胞复合体(Cumulus-oocyte complexs,COCs)。在体视显微镜下挑选出胞质均匀、卵丘细胞致密且至少大于3层的GV期COCs(图 1A),将挑取的COCs放入含1.5 mL成熟液的培养皿中,置于38.5 ℃,5%CO2和100%湿度的培养箱培养24 h。用200 μL移液器将卵母细胞成熟培养前后的卵丘细胞(Cumulus cells,CCs)轻轻吹打下来,在体式显微镜下观察对应的卵母细胞,成熟培养前收集的卵母细胞对应的CCs为GV期CCs,体外成熟培养后排出第一极体的COCs(图 1B)对应的CCs为MⅡ期CCs。其中GV期CCs共收集6次,来自701个GV期卵母细胞的CCs,MⅡ期CCs共收集6次,来自620个MⅡ期卵母细胞的CCs,将收集的两个时期的CCs经1 200 r·min-1离心5 min,弃上清,再用PBS重悬离心2次,弃上清,立即投入液氮冷冻,用于总RNA的提取。
|
图 1 GV(A)和MⅡ(B)期的卵丘-卵母细胞复合体 Figure 1 The cumulus-oocyte complexs of GV (A) and MⅡ(B) stage |
按照mirVanaTMmiRNA Isolation Kit操作步骤提取两个时期卵丘细胞的总RNA,利用1%琼脂糖凝胶电泳检测RNA降解程度及是否有蛋白污染,通过Agilent 2100进行RNA完整度检测,样品检测合格后,使用Small RNA Sample Pre Kit构建文库,利用Small RNA的3′及5′端特殊结构(5′端有完整的磷酸基团,3′端有羟基),以总RNA为起始样品,直接将Small RNA两端加上接头,然后反转录合成cDNA。随后经过PCR扩增,PAGE胶电泳分离目标DNA片段,切胶回收得到的即为cDNA文库,而后进行HiSeq测序。
1.3 卵丘细胞miRNA获取对测序得到的数据进行过滤,通过去掉低质量的reads、去除有5′接头污染的reads等,得到clean reads等,将用bowtie筛选后的sRNA定位到参考序列,去除其中可能的rRNA、tRNA、snRNA、snoRNA,剩余序列与miRBase21.0中Bos taurus的已知miRNAs进行比对,从而得到已知的miRNA,试验整合miREvo和miRdeep2等miRNA预测软件,进行新miRNA的预测分析。
1.4 卵丘细胞miRNAs差异筛选和富集分析对各样本中已知和新miRNA进行表达量的统计,并用TPM[9]进行表达量归一化处理,采用TMM对readcount数据进行标准化处理,之后用DEGseq进行差异分析,从差异倍数(Fold change)和校正后的显著水平(q value<0.01) 进行评估,对差异miRNAs进行筛选。对获得的差异表达的miRNAs进行靶基因预测,并对这些靶基因进行GO富集分析和KEGG富集分析。
1.5 实时定量PCR验证(qRT-PCR)随机选取GV/MⅡ期卵丘细胞中差异表达的4个上调的miRNAs和4个下调的miRNAs进行qRT-PCR检测,以U6为内参基因。由生工生物工程有限公司合成miRNA反转录引物(表 1),qRT-PCR荧光定量引物(表 2)。以miRNA反转录引物合成cDNA作为qRT-PCR模板。PCR反应体系20 μL:10 μL SYBR Premix Ex TaqTM Ⅱ(TaKaRa),8 μL DEPC水,1 μL cDNA,ROX 0.4 μL,0.6 μL的10 μmol·L-1正反向引物。PCR反应条件:95 ℃5 min;95 ℃15 s,55 ℃30 s,共35个循环;72 ℃延伸5 min。相对表达量结果采用2-△△Ct法进行计算。
|
|
表 1 miRNAs反转录引物序列 Table 1 The RT-primers of miRNAs |
|
|
表 2 qRT-PCR荧光定量引物 Table 2 The primers of qRT-PCR |
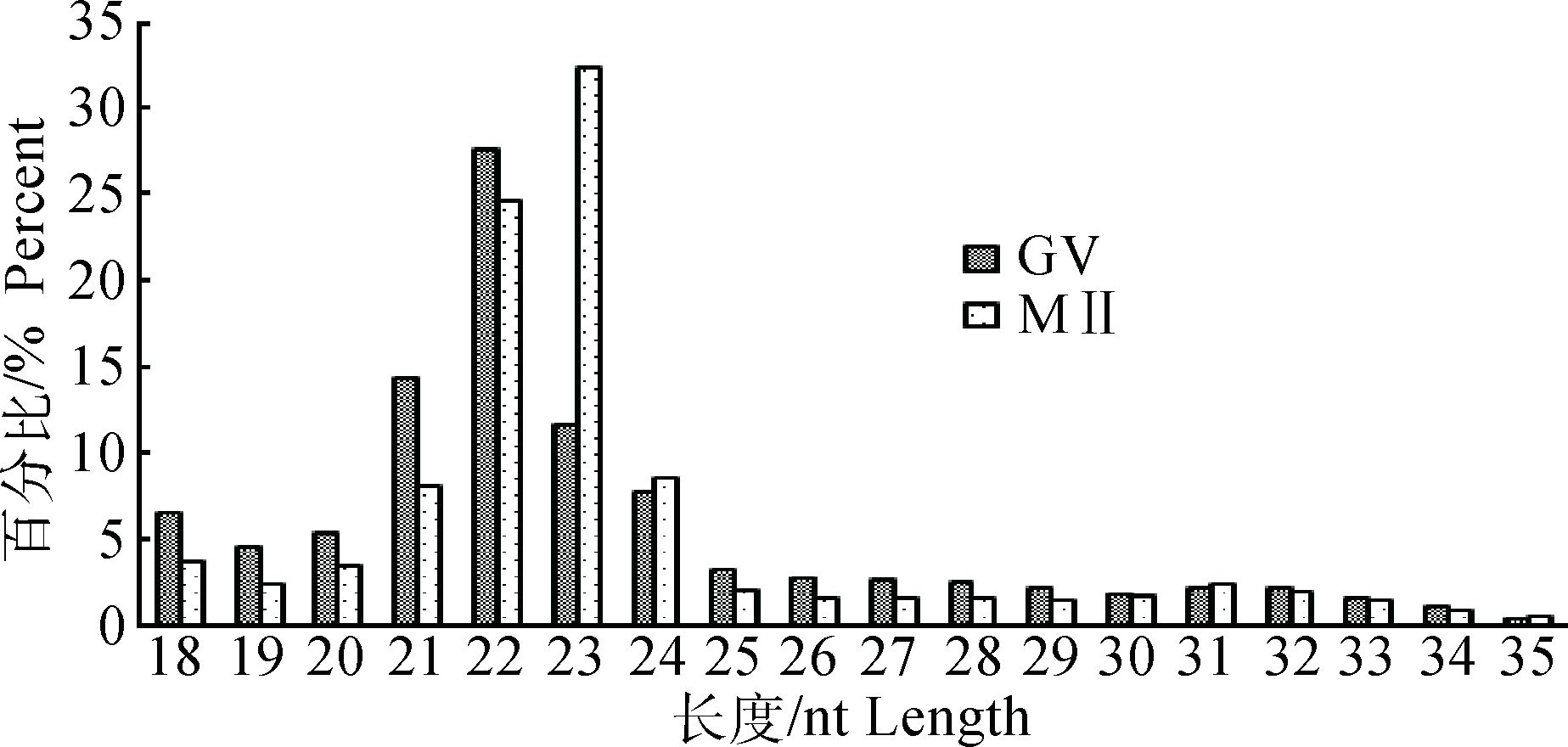
经过Solexa测序和生物信息学分析,去除接头、去除污染及低质量序列后,GV期和MⅡ期CCs两文库分别得到了12 963 256和10 430 776条Clean reads,统计2个sRNA文库中所有sRNA序列的长度分布,大部分序列长度都集中在21~24 nt,其中在GV期CCs sRNA的全部序列中,22 nt长度的比例最高,占27.54%,其次是21 nt长度的比例,占14.34%;MⅡ期CCs sRNA的全部序列中,23 nt长度的比例最高,占32.33%,其次是22 nt长度的比例,占24.60%(图 2)。将长度筛选后的sRNA定位到参考序列上,得到sRNA在参考序列上的分布情况(表 3)。
|
图 2 小RNA长度分布 Figure 2 Length distribution of small RNA sequencing |
|
|
表 3 与参考基因组比对信息统计 Table 3 Comparision of information statistics with reference genome |
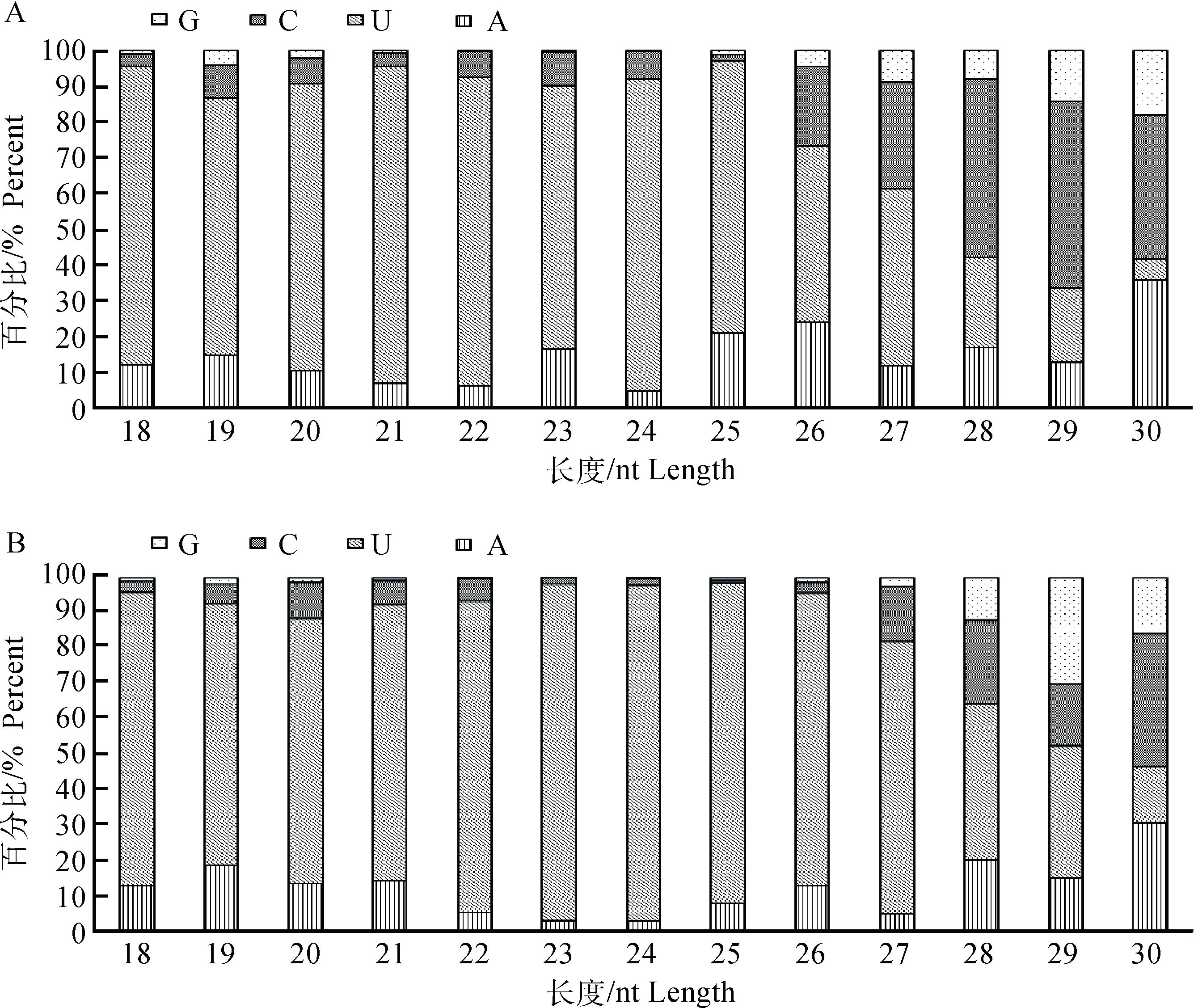
将上述mapped到Bos taurus参考基因组(ftp://ftp.ensembl.org/pub/release-80/gtf/)的reads,与miRBase中指定范围序列进行比对,得到各样品匹配上的sRNA的情况(表 4),包括比对上的已知miRNA的二级结构,各样本中miRNA的序列、长度、出现的次数等信息。对miRNA成熟体序列首位碱基的偏向性进行分析,得到两个时期的miRNAs首位碱基偏好性情况(图 3),发现对U碱基偏好性最强,其次是A碱基,其中U、A碱基在GV期对应的CCs miRNAs文库中分别为84.60%、8.13%,在MⅡ期对应的CCs miRNAs文库中分别为90.66%、5.23%。
|
|
表 4 2个文库中已知miRNAs miRBase比对分析 Table 4 miRBase alignment of conserved miRNAs from 2 libraries |
|
图 3 GV期CCs(A)和MⅡ期CCs(B)文库中miRNAs各长度首位碱基偏好性 Figure 3 Nucleotide bias with different length of miRNAs in GV and MⅡCCs libraries |
通过截取一定长度与miRBaes 21.0中Bos taurus 保守miRNAs没有比对上的sRNA并和参考序列进行比对,利用miREvo[10]和mirdeep2[11]miRNA软件对其二级结构及Dicer酶切位点信息、能量等特征进行分析,预测样品中新miRNA。共预测得到48个miRNA成熟体、13个miRNA *链和52个前体(表 5)。
|
|
表 5 两个样品中新miRNAs的预测 Table 5 Prediction of novel miRNAs in two samples |
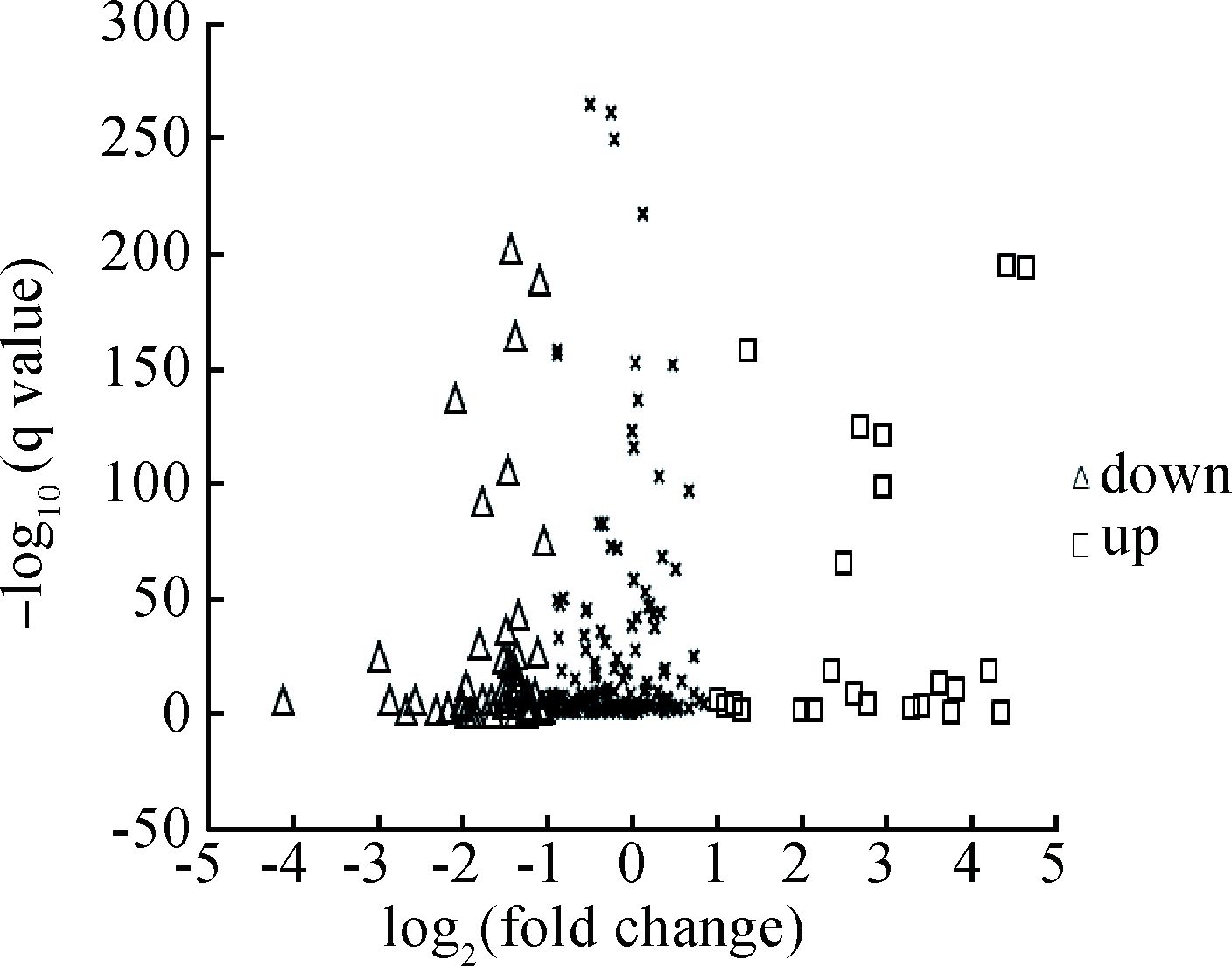
两个时期的卵丘细胞miRNAs表达量用TPM归一化处理后,采用TMM对read count数据进行标准化处理,之后用DEGseq进行差异分析,结合q value<0.01和log2|(差异倍数)|>1两个水平对差异表达情况进行评估,结果如图 4所示:总共得到69个差异表达的miRNAs,相对于GV期CCs,在MⅡ期CCs中有24个miRNAs表达上调,45个表达下调,其中表达上调排在前5位的分别为bta-miR-155、bta-miR-21-5p、bta-miR-132、bta-miR-222、bta-let-7g,其中表达下调排在前5位的分别为bta-miR-106b、bta-miR-16b、bta-miR-195、bta-miR-19b、bta-miR-15a。
|
□、△表示显著差异表达的miRNAs(log2|(差异倍数)|>1,q value<0.01) The square and triangle indicate significant difference in expression of miRNAs(log2|Fold change|>1, q value < 0.01) 图 4 显著差异表达miRNAs分析火山图 Figure 4 The vocano plot of significant differentially expressed miRNAs |
对69个差异表达的miRNA进行靶基因预测共得到7 956个靶基因,并对这些靶基因进行Gene Ontology富集分析,结果如图 5所示,在生物学过程分类中,这些靶基因在代谢过程所占比重最大(58.16%),其次是有机物质代谢过程(49.44%);在细胞组分分类中,靶基因定位到细胞内的占最大比重(66.53%),其次是定位到细胞外的(64.59%);在分子功能分类中有绝大部分的靶基因定位到分子结合功能上(80.85%),其次是蛋白结合功能上(67.35%),再次是催化活性方面(33%)和离子结合功能上(24.49%)。
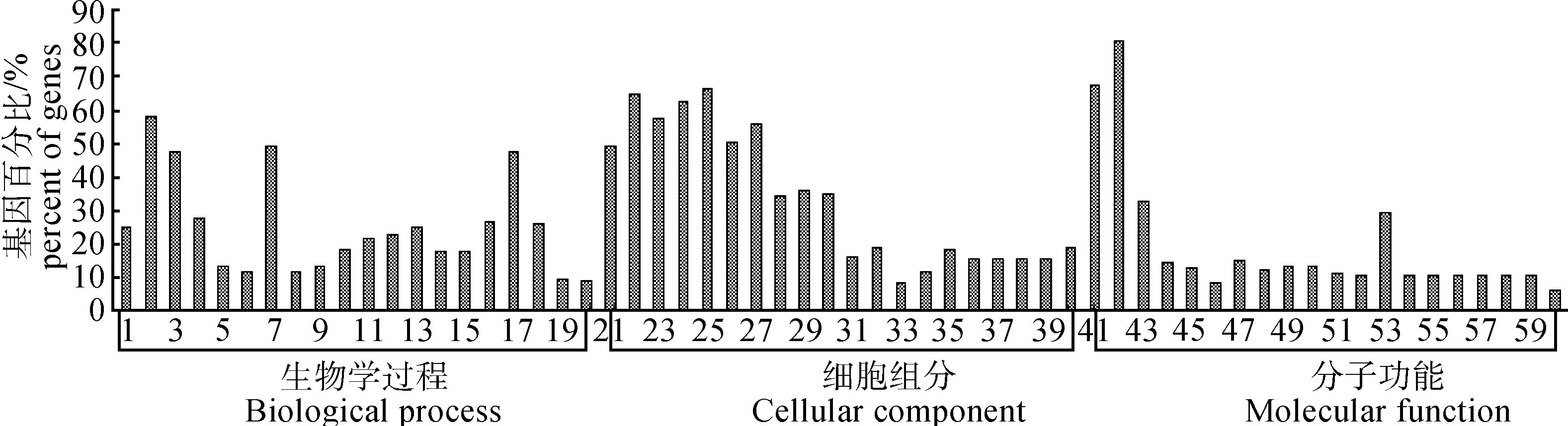
|
1.单一的有机体的代谢;2.代谢过程;3.细胞代谢过程;4.定位;5.大分子定位;6.蛋白定位;7.有机物质代谢过程;8.有机物质的运输;9.细胞定位;10.高分子改性;11.运输;12.定位系统的建立;13.生物过程的正调控;14.蛋白质修饰;15.蛋白质改性;16.细胞组分的组织和生物合成;17.初级代谢过程;18.细胞组分的组织;19.蛋白质定位的建立;20.蛋白质转运;21.细胞质;22.细胞内组分;23.膜结合细胞器;24.细胞器;25.细胞内;26.细胞内有膜细胞器;27.细胞内的细胞器;28.胞浆部分;29.细胞器;30.细胞内细胞器;31.内膜系统;32.囊泡;33.有膜细胞器;34.细胞器膜;35.膜小泡;36.细胞外的细胞器;37.细胞外有膜细胞器;38.胞囊;39.胞外体;40.胞外区部分;41.蛋白结合;42.结合;43.催化活性;44.阴离子配位;45.转移酶活性;46.酶结合;47.小分子的结合;48.碳水化合物衍生物的结合;49.核苷酸结合;50.核苷磷酸结合;51.嘌呤核苷酸结合;52.核苷结合;53.离子结合;54.核糖核苷酸结合;55.嘌呤核糖核苷酸结合;56.核糖核苷结合;57.嘌呤核苷结合;58.嘌呤核糖核苷酸结合;59.嘌呤核糖核苷三磷酸结合;60.相同的蛋白结合 1.Single-organism metabolic; 2.Metabolic process; 3.Cellular metabolic; 4.Localization; 5.Macromolecule localization; 6.Protein localization; 7.Organic substance metabolic; 8.Organic substance transport; 9.Cellular localization; 10.Macromolecule modification; 11.Transport; 12.Establishment of localization; 13.Positive regulation of biological process; 14.Cellular protein modification; 15.Protein modification process; 16.Cellular component organization or biogenesis; 17.Primary metabolic process; 18.Cellular component organization; 19.Establishment of protein localization; 20.Protein transport; 21.Cytoplasm; 22.Intracellular part; 23.Membrane-bounded organelle; 24.Organelle cellular; 25.Intracellular; 26.Intracellular membrane-bounded organelle; 27.Intracellular organelle; 28.Cytoplasmic part; 29.Organelle part; 30.Intracellular organelle part; 31.Endomembrane system; 32.Vesicle cellular; 33.Bounding membrane of organelle; 34.Organelle membrane; 35.Membrane-bounded vesicle; 36.Extracellular organelle; 37.Extracellular membrane-bounded organelle; 38.Extracellular vesicle; 39.Extracellular exosome; 40.Extracellular region part; 41.Protein binding; 42.Binding; 43.Catalytic activity; 44.Anion binding; 45.Transferase activity; 46.Enzyme binding; 47.Small molecule binding; 48.Carbohydrate derivative binding; 49.Nucleotide binding; 50.Nucleoside phosphate binding; 51.Purine nucleotide binding; 52.Nucleoside binding; 53.Ion binding; 54.Ribonucleotide binding; 55.Purine ribonucleotide binding; 56.Ribonucleoside binding; 57.Purine nucleoside binding; 58.Purine ribonucleoside binding; 59.Purine ribonucleoside triphosphate binding; 60.Identical protein binding 图 5 差异表达miRNAs靶基因的GO功能分类 Figure 5 GO annotation plot for target genes of miRNAs differentially expressed |
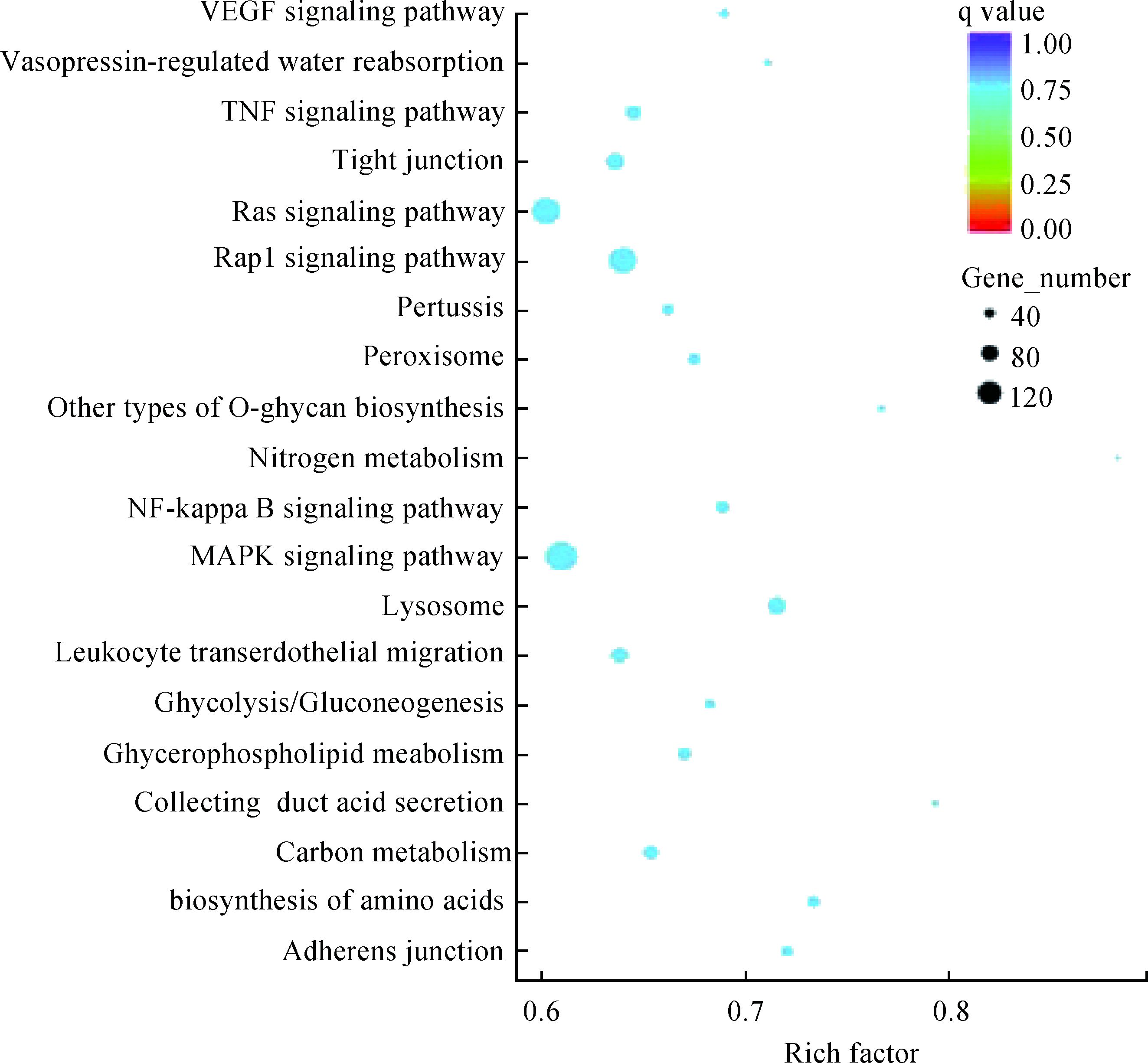
在生物体内,不同基因相互协调行使其生物学功能,通过Pathway显著性富集能确定候选靶基因参与的最主要生化代谢途径和信号转导途径。KEGG富集程度通过Rich factor、qvalue和富集到此通路上的基因个数来衡量,共得到276条显著富集的信号通路,挑选富集前20条的信号通路进行作图(图 6),发现主要包括血管内皮生长因子、Rap1、肿瘤坏死因子、糖酵解/糖异生、碳代谢、氮代谢、紧密连接、黏合连接等20条信号通路,这些信号通路主要集中在细胞增殖、凋亡,物质代谢,细胞连接等方面。推测卵丘细胞通过物质代谢为卵母细胞的成熟提供能量,通过物质转运为卵母细胞提供所需的小分子物质。卵母细胞仅能利用少量的葡萄糖,所需丙酮酸主要通过卵丘细胞糖酵解作用产生并经缝隙连接进入卵母细胞中,供给卵母细胞的糖代谢[12]。卵丘细胞通过缝隙连接运输氨基酸、核苷酸等小分子物质进入到卵母细胞,还可以增加卵母细胞中谷胱甘肽(GSH)的含量,防止卵母细胞因氧化应激而凋亡[13]。同时,卵丘细胞在卵母细胞成熟过程中逐渐扩张, 细胞连接减弱,从而减少卵丘细胞从间隙连接运输到卵母细胞的cGMP,卵母细胞PDE3A活性恢复,卵母细胞中的cAMP含量下降,减数分裂得以恢复[14]。
|
图 6 差异表达miRNAs靶基因KEGG富集散点图 Figure 6 KEGG enriched scatter plot for target genes of miRNAs differentially expressed |
在GV/MⅡ期卵丘细胞差异表达的69个miRNAs中,随机选取4条表达上调的miRNAs(bta-miR-21-5p、bta-miR-155、bta-miR-30d、bta-miR-486) 和4条表达下调的miRNAs(bta-miR-491、bta-miR-1296、bta-miR-106b、novel-214) 进行qRT-PCR验证,结果发现和测序结果表达趋势一致(表 6)。
|
|
表 6 qRT-PCR与RNA-Seq结果比较 Table 6 Comparision of results between qRT-PCR and RNA-Seq |
卵母细胞体外成熟培养技术是动物繁殖领域中不断改进的一个关键技术,卵丘细胞在卵母细胞体外成熟中起重要作用,因此加强对卵丘细胞的研究显得尤为重要。本研究利用高通量测序技术对水牛COCs成熟前后卵丘细胞的miRNAs进行Solex二代测序,旨在构建卵丘细胞两个时期的miRNA文库,并进行生物信息学分析,探索miRNA在卵丘细胞中发挥何种作用,为进一步阐明卵丘细胞在卵母细胞体外成熟过程中发挥的重要作用提供科学依据。
本试验通过对水牛卵母细胞体外成熟前后GV/MⅡ期卵丘细胞miRNAs研究,丰富了水牛miRNAs数据库,分析得出两个时期的卵丘细胞已知miRNAs成熟体有446个,前体有521个;预测的novel miRNAs成熟体有48个,前体有52个;差异表达的miRNAs共有69个,其中上调有24个,全部为已知miRNA,下调有45个,其中有两个新miRNAs,分别为novel-148、novel-214。G.C.Gilchrist等在对牛卵母细胞GV、MⅡ两个时期的miRNA测序发现,bta-miR-155、bta-miR-222、bta-miR-21在GV、MⅡ卵母细胞中表达差异显著(P < 0.05),且Pri-miR-155、Pri-miR-222在GV期没有检测到,而Pri-miR-155在MⅡ期卵母细胞中有表达[15]。而本研究发现,bta-miR-155、bta-miR-222、bta-miR-21-5p在GV、MⅡ期卵母细胞对应的卵丘细胞中表达差异显著,呈现表达上调的趋势,推测miRNA可能由卵丘细胞经过微囊体和外泌体的方式作用于卵母细胞,具体还需要进一步验证。胞外囊泡包括微囊体和外泌体已经在包括人、牛、马等很多哺乳动物的卵泡液中鉴定到[16-19],在癌细胞系中发现胞外囊泡,可以通过运载蛋白质、RNA等来影响其他细胞的基因表达[20]。miR-21-5p、miRNA-155、miRNA-222在闭锁卵泡中表达量高于健康卵泡,且miRNA-155在颗粒细胞中可能靶向于HIF1A(缺氧诱导因子1),miRNA-155、miRNA-222在膜细胞中可能靶向于ETS1基因,miR-155可能靶向于MSH2,这些结果表明,miRNAs可能作用于相应靶基因参与到卵泡闭锁中[21]。K.Sayasith等研究表明,LH通过几条信号通路刺激成熟卵泡排卵,RGS2的受调控被认为参与其中,而ETS1被验证具有调控RGS2表达的作用[22]。P.Feuerstein等对成熟卵母细胞和未成熟卵母细胞对应时期的卵丘细胞miRNA进行微阵列分析,发现在P < 0.000 1的基因中,PLIN2/RGS2/ANG被验证可以作为潜在的标记基因,因为试验因素的影响,比如病人之间和qRT-PCR变化等,采用广义线性混合模型进行计算,发现只有RGS2和卵母细胞发育能力相关[23]。同时发现相对于未怀孕的对应CCs而言,怀孕对应的CCs中,RGS2的表达量显著增加((4.77±1.68) vs (1.75±0.23),P < 0.05)[23]。M.L.Bernhardt等研究表明,MⅡ期卵母细胞中RGS2蛋白的表达量是未成熟卵母细胞中表达量的20倍以上,注射Rgs2-siRNA的卵母细胞成熟到MⅡ期之后,对低pH和乙酰胆碱更加敏感,导致Ca2+的异常释放和未成熟卵母细胞的激活,发现RGS2可以平衡未成熟卵母细胞中Ca2+释放来调控卵母细胞成熟[24]。因此,miRNA-155、miRNA-222可能作用于ETS1基因,进而调控细胞中RGS2的表达,在卵母细胞和卵丘细胞的不同阶段发挥作用,具体还有待于进一步研究。HIF1通过在血管内皮生长因子(VEGFA)启动子中扮演一个单独缺氧效应元件,进而发挥其调节作用,FSH、LH通过缺氧诱导因子(HIF1) 调控颗粒细胞中VEGFA的表达情况,而VEGFA在卵泡发育过程中发挥着重要作用[25]。miRNA-155、miRNA-199a-5p可能作用于HIF1基因进而调控VEGFA的表达情况,在卵泡发育过程中发挥重要作用,具体还有待于进一步研究。
同时本次测序中其它显著差异表达的miRNAs在已有研究中也有提到。miR-132参与到cAMP信号通路中,通过抑制颗粒细胞中Nurr1的表达来促进雌二醇的合成[26]。A.V.Sirotkin等研究发现,在颗粒细胞培养液中,添加miR-15a抑制剂显著增加细胞增殖基因(MAPK/ERK1/2、PCNA)和凋亡相关基因(Caspase3、bax),减少黄体素和睾酮表达量,增加雌二醇的表达量,而培养液中添加pre-Mir15a则结果相反[27]。猪颗粒细胞中let-7g通过靶向于TGFBR1基因诱导颗粒细胞的凋亡[28];let-7g在猪凋亡卵泡中高度富集,颗粒细胞中过表达let-7g促进颗粒细胞的凋亡[29]。
4 结论本研究成功构建了水牛卵母细胞GV/MⅡ期的卵丘细胞miRNA数据库,并筛选出了两个时期差异表达的miRNAs;同时结合靶基因预测及KEGG信号通路富集分析,推测出差异表达的miRNAs主要通过细胞增殖、凋亡,物质代谢,细胞连接等途径在卵丘细胞中发挥作用,进而调控卵母细胞的成熟。这些结果将为后续研究水牛卵丘细胞miRNA在卵母细胞成熟过程中发挥的重要作用提供科学依据。
| [1] | DEY S R, DEB G K, HA A N, et al. Coculturing denuded oocytes during the in vitro maturation of bovine cumulus oocyte complexes exerts a synergistic effect on embryo development[J]. Theriogenology, 2012, 77(6): 1064–1077. DOI: 10.1016/j.theriogenology.2011.10.009 |
| [2] | NISHI Y, TAKESHITA T, SATO K, et al. Change of the mitochondrial distribution in mouse ooplasm during in vitro maturation[J]. J Nippon Med Sch, 2003, 70(5): 408–415. DOI: 10.1272/jnms.70.408 |
| [3] | ZHANG L, JIANG S E, WOZNIAK P J, et al. Cumulus cell function during bovine oocyte maturation, fertilization, and embryo development in vitro[J]. Mol Reprod Dev, 1995, 40(3): 338–344. DOI: 10.1002/(ISSN)1098-2795 |
| [4] | KIM Y J, KU S Y, KIM Y Y, et al. microRNAs transfected into granulosa cells may regulate oocyte meiotic competence during in vitro maturation of mouse follicles[J]. Hum Reprod, 2013, 28(11): 3050–3061. DOI: 10.1093/humrep/det338 |
| [5] | HUANG X, LIU C, HAO C F, et al. Identification of altered microRNAs and mRNAs in the cumulus cells of PCOS patients: miRNA-509-3p promotes oestradiol secretion by targeting MAP3K8[J]. Reproduction, 2016, 151(6): 643–655. DOI: 10.1530/REP-16-0071 |
| [6] | LI C J, CHEN C, CHEN L, et al. BDNF-induced expansion of cumulus-oocyte complexes in pigs was mediated by microRNA-205[J]. Theriogenology, 2016, 85(8): 1476–1482. DOI: 10.1016/j.theriogenology.2016.01.004 |
| [7] | PAN B, TOMS D, SHEN W, et al. microRNA-378 regulates oocyte maturation via the suppression of aromatase in porcine cumulus cells[J]. Am J Physiol Endocrinol Metab, 2015, 308(6): E525–E534. DOI: 10.1152/ajpendo.00480.2014 |
| [8] | YAO G D, LIANG M, LI J H, et al. microRNA-224 is involved in the regulation of mouse cumulus expansion by targeting Ptx3[J]. Mol Cell Endocrinol, 2014, 382(1): 244–253. DOI: 10.1016/j.mce.2013.10.014 |
| [9] | ZHOU L, CHEN J H, LI Z Z, et al. Integrated profiling of microRNAs and mRNAs: microRNAs located on Xq27. 3 associate with clear cell renal cell carcinoma[J]. PLoS One, 2010, 5(12): e15224. DOI: 10.1371/journal.pone.0015224 |
| [10] | WEN M, SHEN Y, SHI S H, et al. miREvo: an integrative microRNA evolutionary analysis platform for next-generation sequencing experiments[J]. BMC Bioinform, 2011, 13(1): 140. |
| [11] | FRIEDLÄNDER M R, MACKOWIAK S D, LI N, et al. miR Deep2 accurately identifies known and hundreds of novel microRNA genes in seven animal clades[J]. Nucleic Acids Res, 2012, 40(1): 37–52. DOI: 10.1093/nar/gkr688 |
| [12] | WANG Q, CHI M M, SCHEDL T, et al. An intercellular pathway for glucose transport into mouse oocytes[J]. Am J Physiol Endocrinol Metab, 2012, 302(12): E1511–E1518. DOI: 10.1152/ajpendo.00016.2012 |
| [13] | TATEMOTO H, SAKURAI N, MUTO N. Protection of porcine oocytes against apoptotic cell death caused by oxidative stress during in vitro maturation: role of cumulus cells[J]. Biol Reprod, 2000, 63(3): 805–810. DOI: 10.1095/biolreprod63.3.805 |
| [14] | ZHANG M J, XIA G L. Hormonal control of mammalian oocyte meiosis at diplotene stage[J]. Cell Mol Life Sci, 2012, 69(8): 1279–1288. DOI: 10.1007/s00018-011-0867-3 |
| [15] | GILCHRIST G C, TSCHERNER A, NALPATHAMKALAM T, et al. microRNA expression during bovine oocyte maturation and fertilization[J]. Int J Mol Sci, 2016, 17(3): 396. DOI: 10.3390/ijms17030396 |
| [16] | SOHEL M M, HOELKER M, NOFERESTI S S, et al. Exosomal and non-exosomal transport of extra-cellular microRNAs in follicular fluid: implications for bovine oocyte developmental competence[J]. PLoS One, 2013, 8(11): e78505. DOI: 10.1371/journal.pone.0078505 |
| [17] | DA SILVEIRA J C, VEERAMACHANENI D N R, WINGER Q A, et al. Cell-secreted vesicles in equine ovarian follicular fluid contain miRNAs and proteins: a possible new form of cell communication within the ovarian follicle[J]. Biol Reprod, 2001, 86(3): 71. |
| [18] | SANG Q, YAO Z Y, WANG H, et al. Identification of microRNAs in human follicular fluid: characterization of microRNAs that govern steroidogenesis in vitro and are associated with polycystic ovary syndrome in vivo[J]. J Clin Endocrinol Metab, 2013, 98(7): 3068–3079. DOI: 10.1210/jc.2013-1715 |
| [19] | DIEZ-FRAILE A, LAMMENS T, TILLEMAN K, et al. Age-associated differential microRNA levels in human follicular fluid reveal pathways potentially determining fertility and success of in vitro fertilization[J]. Hum Fertil, 2014, 17(2): 90–98. DOI: 10.3109/14647273.2014.897006 |
| [20] | COCUCCI E, MELDOLESI J. Ectosomes and exosomes: shedding the confusion between extracellular vesicles[J]. Trends Cell Biol, 2015, 25(6): 364–372. DOI: 10.1016/j.tcb.2015.01.004 |
| [21] | DONADEU F X, MOHAMMED BT, IOANNIDIS J. A miRNA target network putatively involved in follicular atresia[J]. Domest Anim Endocrinol, 2017, 58: 76–83. DOI: 10.1016/j.domaniend.2016.08.002 |
| [22] | SAYASITH K, SIROIS J, LUSSIER J G. Expression and regulation of regulator of G-protein signaling protein-2 (RGS2) in equine and bovine follicles prior to ovulation: molecular characterization of RGS2 transactivation in bovine granulosa cells[J]. Biol Reprod, 2014, 91(6): 139. |
| [23] | FEUERSTEIN P, PUARD V, CHEVALIER C, et al. Genomic assessment of human cumulus cell marker genes as predictors of oocyte developmental competence: impact of various experimental factors[J]. PLoS One, 2012, 7(7): e40449. DOI: 10.1371/journal.pone.0040449 |
| [24] | BERNHARDT M L, LOWTHER K M, PADILLABANKS E, et al. Regulator of G-protein signaling 2 (RGS2) suppresses premature calcium release in mouse eggs[J]. Development, 2015, 142(15): 2633–2640. DOI: 10.1242/dev.121707 |
| [25] | RICO C, DODELET-DEVILLERS A, PAQUET M, et al. HIF1 activity in granulosa cells is required for FSH-regulated Vegfa expression and follicle survival in mice[J]. Biol Reprod, 2014, 90(6): 135. |
| [26] | WU S G, SUN H X, ZHANG Q, et al. microRNA-132 promotes estradiol synthesis in ovarian granulosa cells via translational repression of Nurr1[J]. Reprod Biol Endocrinol, 2015, 13(1): 94. DOI: 10.1186/s12958-015-0095-z |
| [27] | SIROTKIN A V, KISOVÁ G, BRENAUT P, et al. Involvement of microRNA Mir15a in control of human ovarian granulosa cell proliferation, apoptosis, steroidogenesis, and response to FSH[J]. microRNA, 2014, 3(1): 29–36. DOI: 10.2174/2211536603666140227232824 |
| [28] | ZHOU J L, LIU J Y, PAN Z X, et al. The let-7g microRNA promotes follicular granulosa cell apoptosis by targeting transforming growth factor-β type 1 receptor[J]. Mol Cell Endocrinol, 2015, 409: 103–112. DOI: 10.1016/j.mce.2015.03.012 |
| [29] | CAO R, WU W J, ZHOU X L, et al. Expression and preliminary functional profiling of the let-7 family during porcine ovary follicle atresia[J]. Mol Cells, 2015, 38(4): 304–311. DOI: 10.14348/molcells.2015.2122 |



