2. 张家港市畜禽有限公司, 苏州 215623
2. Zhangjiagang Livestock and Poultry Co., Ltd, Suzhou 215623, China
我国现有地方鸡品种资源107个,在长期的进化选择过程中形成了鲜明的特征特性,保护好这些地方鸡种资源丰富的遗传多样性对于家禽产业可持续发展具有重要意义。鹿苑鸡(Luyuan chicken),属肉蛋兼用型品种,因产于江苏省张家港市鹿苑镇而得名。鹿苑鸡饲养历史悠久,1989年被《中国家禽品种志》收录,2004年被列入中国畜禽遗传资源名录,2014年列入国家级重点保护品种名录[1]。目前,鹿苑鸡采用保种场和基因库两种方式保护。张家港市畜禽有限公司承担原产地保种任务,国家级地方鸡种基因库于1996年从原产地引种(抢救性保护,原始群体数量不足200只)进行异地保存。经历近20个世代后,两个保种群的遗传多样性现状如何,群体间是否存在遗传分化,针对这些问题一直未开展相关研究。
保种效果监测是保种实施过程中的一项重要内容,依靠科学、可靠的保种效果监测与评价方法,能够及时发现保种实践中出现的各种问题,进而制定更合理的保种技术方案。目前家禽遗传资源保种效果监测仍主要依赖于常规表型性状记录、系谱记录和低密度DNA分子标记信息,由于对群体遗传多样性度量效力不高,导致对保种群的状态难以实现全面评估。新一代测序技术NGS的发展为更全面精确地监测保种效果提供了新的方法,其中简化基因组测序技术能鉴定出合适数量、广泛覆盖基因组范围的变异位点,被广泛应用于资源群体的遗传多样性研究,包括三刺鱼群体差异与选择[2]、瓶草蚊群体系统进化[3]、虹鳟鱼SNPs发掘[4]以及大麦[5]、黑麦草[6]、小菜蛾[7]构建连锁图谱等。
本研究利用RAD-seq简化基因组测序比较分析基因库与保种场两个鹿苑鸡群体的遗传多样性差异,评价保种效果,为进一步优化保种技术方案提供依据,也为其他地方鸡品种资源的保种效果监测提供借鉴。
1 材料与方法 1.1 试验材料两个鹿苑鸡群体分别来自于国家级地方鸡种基因库和张家港鹿苑鸡保种场,藏鸡、茶花鸡、仙居鸡、固始鸡、白耳黄鸡、大骨鸡、北京油鸡、隐性白羽肉鸡来自于国家级地方鸡种基因库。每个群体按照家系选择30个个体(10公、20母),个体间无亲缘关系。翅静脉采血1~1.5 mL,柠檬酸钠(ACD)抗凝,-20 ℃保存备用。
1.2 试验方法 1.2.1 RAD-seq简化基因组测序常规酚-氯仿法提取基因组DNA。采用ddRAD建库的方式构建长度为300~500 bp的pair-end文库,进行RAD-seq双酶切简化基因组测序。
1.2.2 SNP质控SNP的检测主要使用GATK[8]和samtools[9]程序。质控条件分为两步,第一步为Q20(正确率99%的碱基数目比例)>95%;ddRAD depth(双酶切基因组测序的覆盖深度)>60%,SNP Call rate (在鸡群中单核苷酸多态性检出率)>70%,MAF>0.05;第二步在第一步质控的基础上,SNP Call rate >90%(在单个鸡种中的检出率)。
1.2.3 统计分析连锁不平衡(LD)分析应用Haploview软件。平均杂合度(Ho)、近交系数(Fis)和群体分化指数(Fst)计算应用PopGen32[10]软件。群体聚类分析应用admixture软件,以其他地方鸡种(藏鸡、茶花鸡、仙居鸡、固始鸡、白耳黄鸡、大骨鸡、北京油鸡、隐性白羽肉鸡)作为外群,每个群体30个个体(10公、20母)。选择信号检测采用PLINK软件,以100 kb为窗口,10 kb为step进行滑动的θπ值和Fst分布计算。
2 结果 2.1 基因组SNP鉴定经过两步数据质控,在两个鹿苑鸡群体中分别鉴定出SNP数目为395 021和428 314个。其中共有SNPs位点为369 354个,共有SNPs位点在染色体上的分布见图 1,SNP分布数量与染色体长度总体上呈正相关。
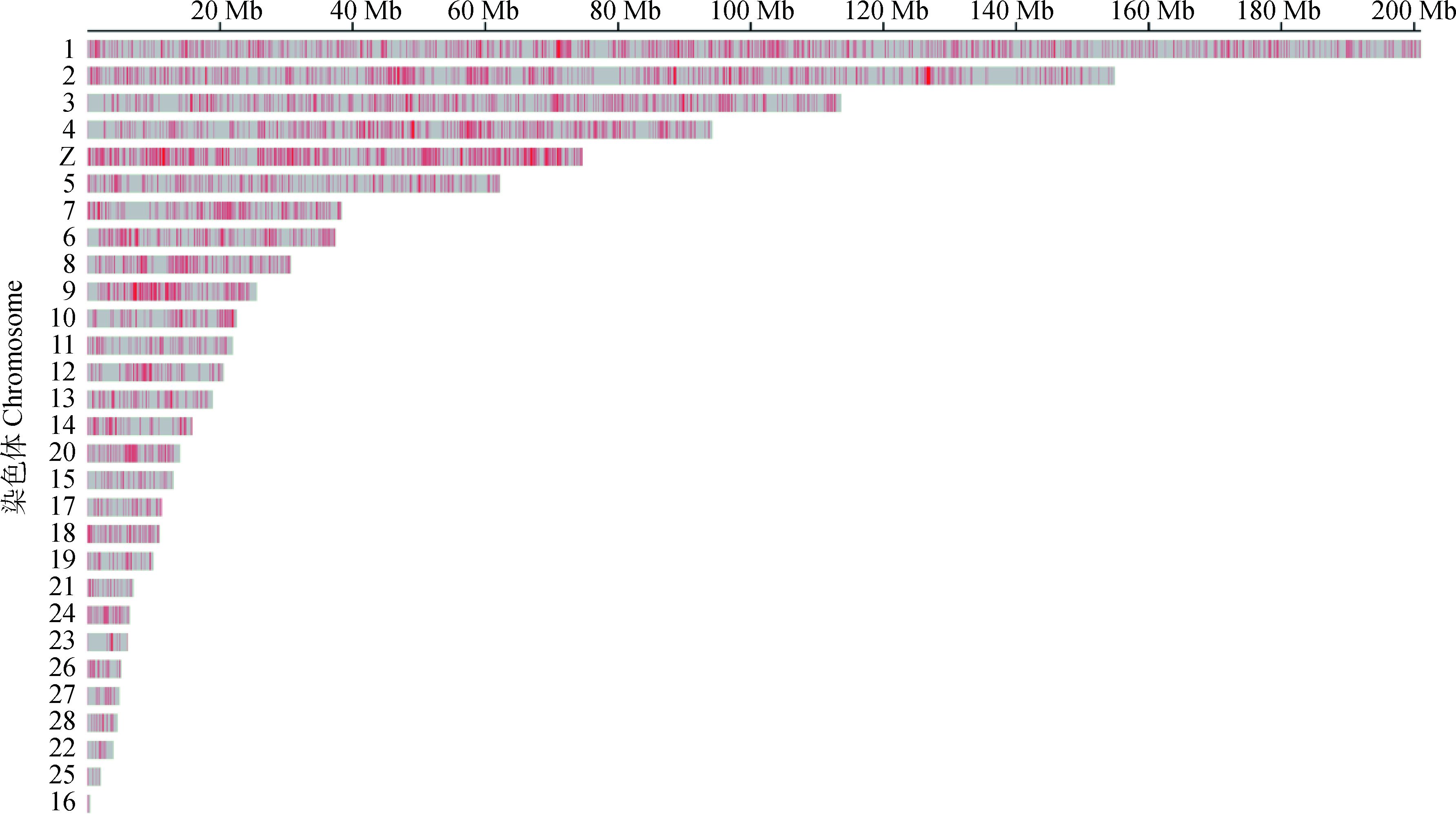
|
图 1 SNPs在染色体上的分布 Figure 1 Distribution of SNPs on chromosomes |
基因库与张家港两个鹿苑鸡群体的平均杂合度分别为0.211 1和0.206 8,差异不显著(P>0.05);近交系数分别为0.178 8和0.193 5,近交水平相对较高(表 1)。
|
|
表 1 两个鹿苑鸡保种群杂合度与近交系数统计 Table 1 The heterozygosity and inbreeding coefficient of two Luyuan chicken populations |
连锁不平衡模式见图 2,结果表明,LD随距离增长逐渐下滑至平缓,SNP距离为0~200 kb范围内的LD水平比在200~1 000 kb范围内高,但未超过r2=0.33的临界水平,显示在总体范围内,各等位基因不存在强烈的连锁不平衡水平。
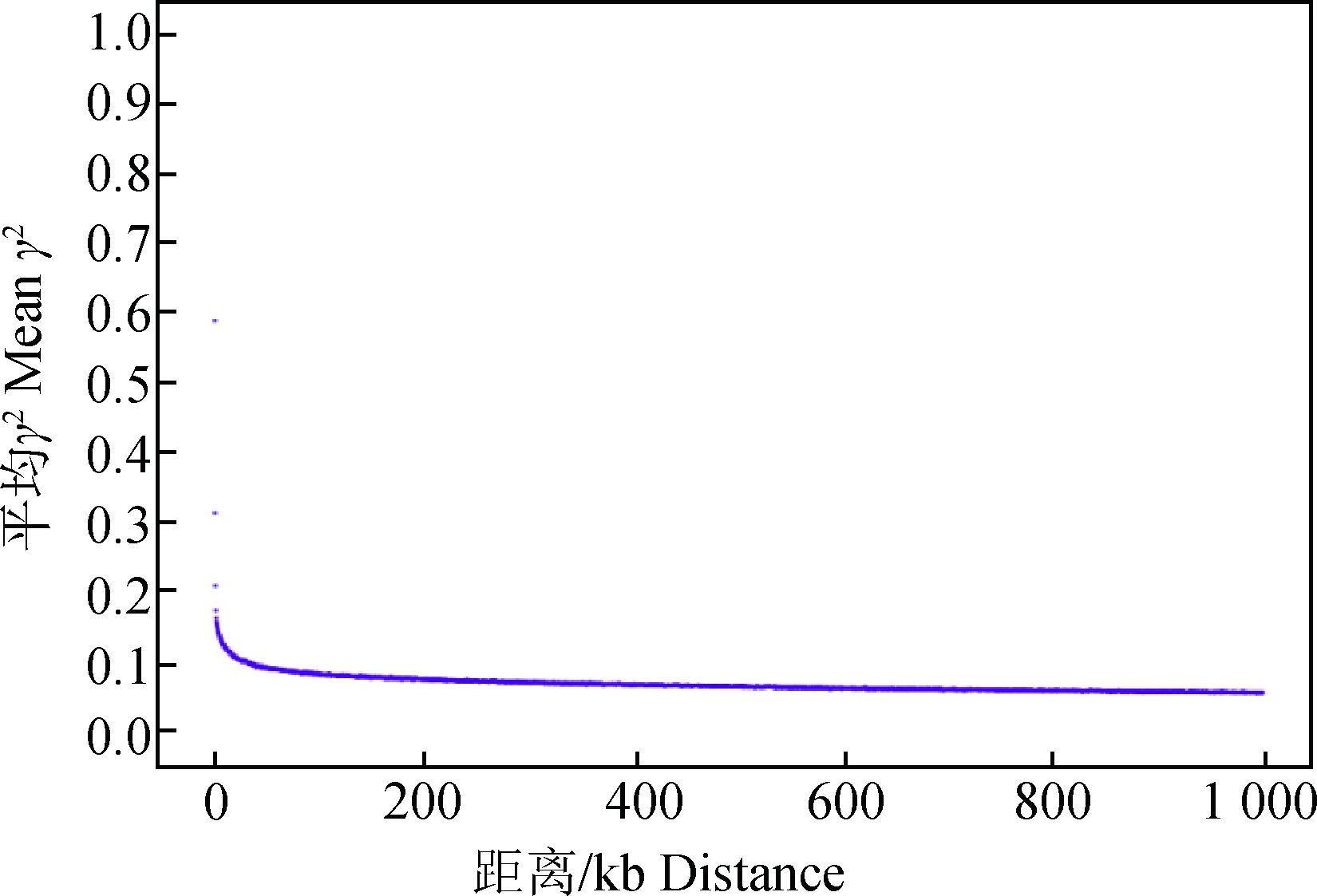
|
图 2 LD随距离增长的衰减图 Figure 2 LD decay with distance |
通过对全部SNPs位点的遗传分化系数Fst值求平均值,得到两个鹿苑鸡群体平均分化指数为0.005 6。以藏鸡(TIB)、茶花鸡(CHA)、仙居鸡(XJ)、固始鸡(GS)、白耳黄鸡(BE)、大骨鸡(DG)、北京油鸡(BY)和隐性白羽肉鸡(REC)为外群的Structure模型聚类分析结果(图 3)也表明,两个鹿苑鸡群体始终聚为一类,没有个体分离出来。
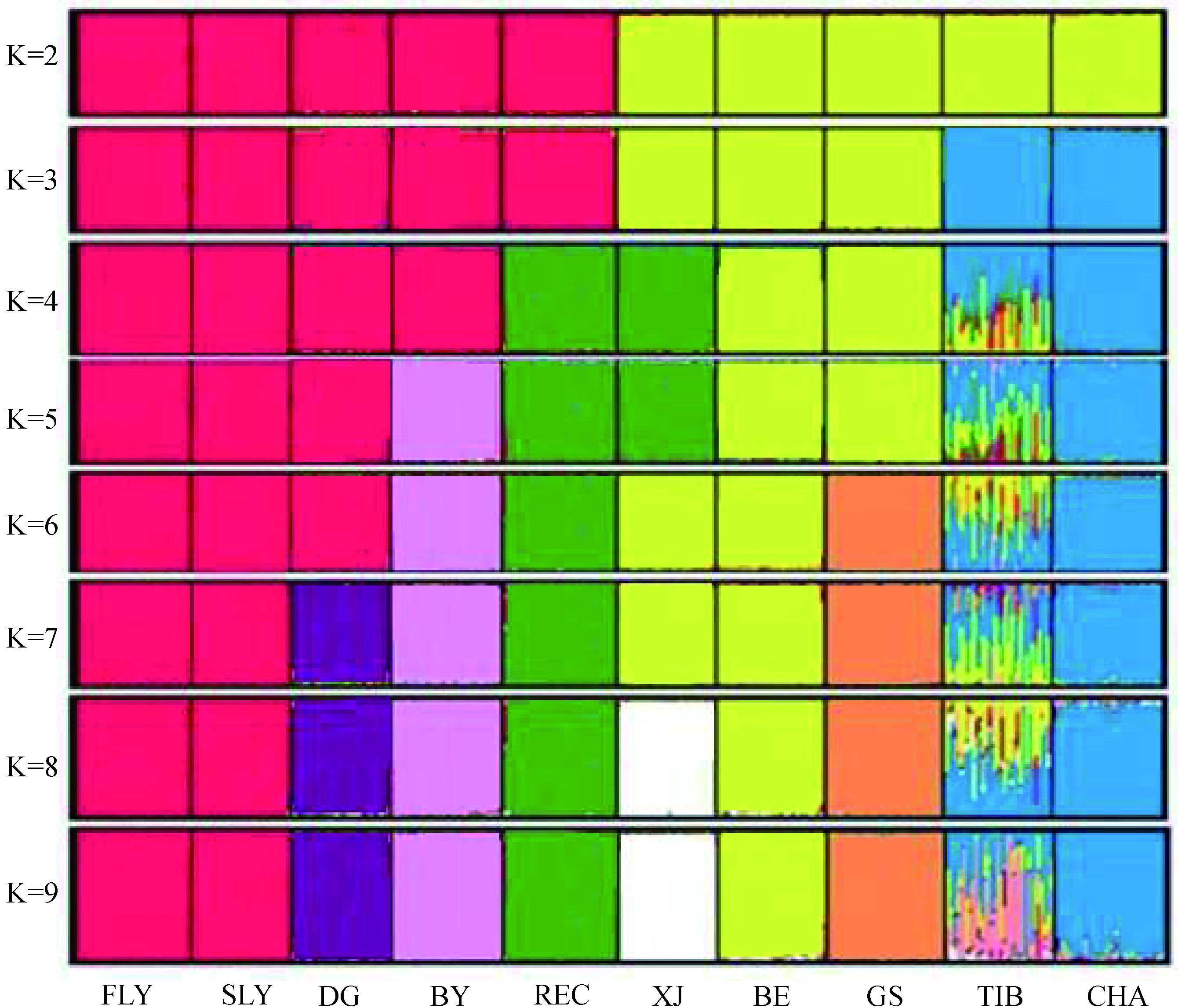
|
图中每种颜色代表一个群;图中展示了10个鸡种330个样品分群值2~9的聚类情况 Each color represents a population; Figure shows that 2-9 clustering value of 330 samples from 10 chicken breeds 图 3 群体结构聚类图 Figure 3 The picture of group structure clustering |
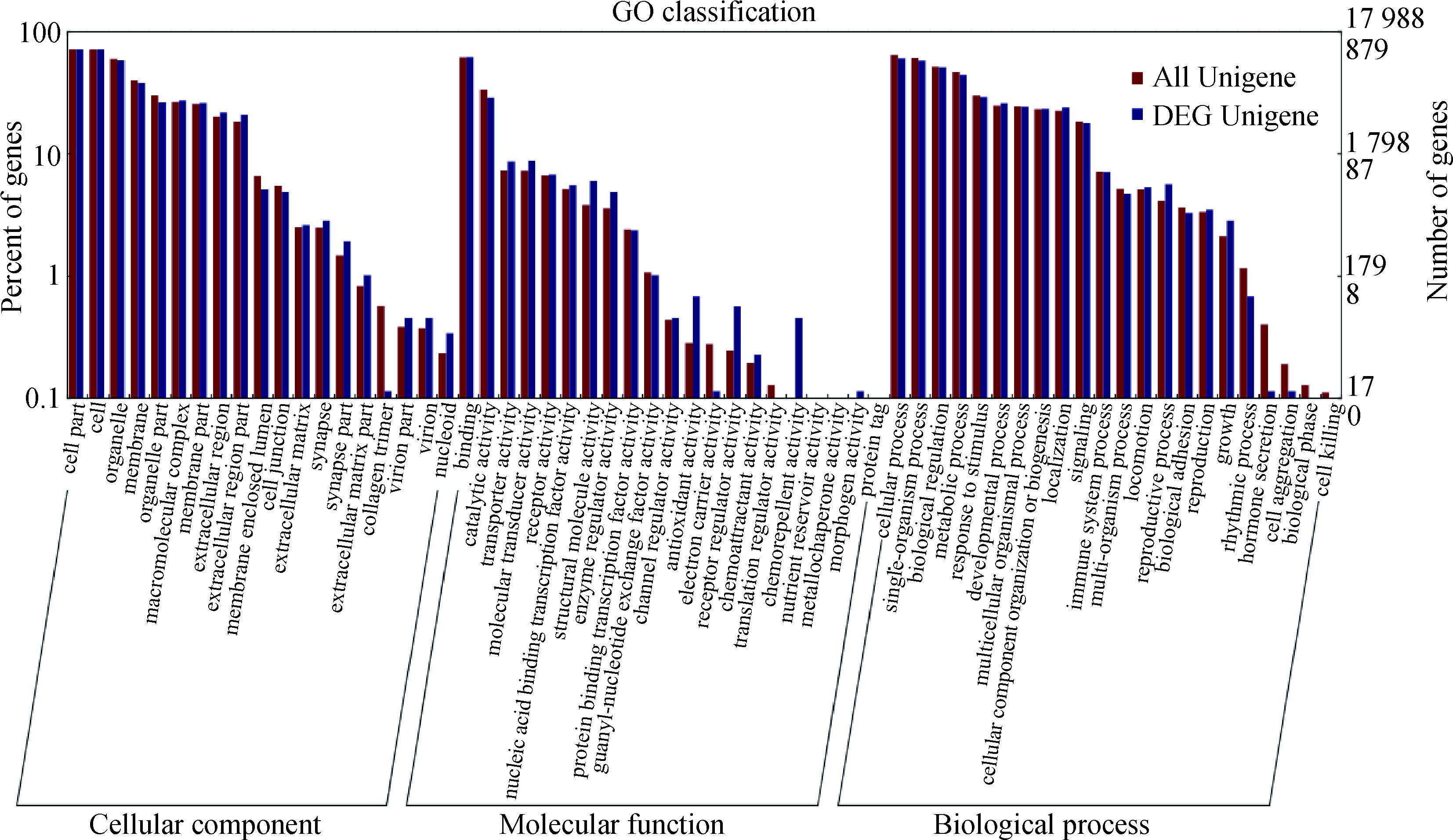
结合核苷酸多样度θπ比率和遗传分化系数Fst的选择信号分析见图 4,受选择的基因组区域阈值为Fst>0.301,θπ≥1.18/θπ≤0.37,共有58个区域受到选择,这些受选择区域主要集中在1、2、4、5、Z等染色体上,筛选到受选择候选基因96个。GO分析结果表明,受选择候选基因主要富集在能量代谢、生长发育、信号传递、应激免疫反应等生物学通路(图 5)。KEGG分析结果表明,受选择基因主要集中在神经传递、氨基酸代谢、肌肉发育(心肌、血管平滑肌)、免疫反应等信号通路上(表 2)。
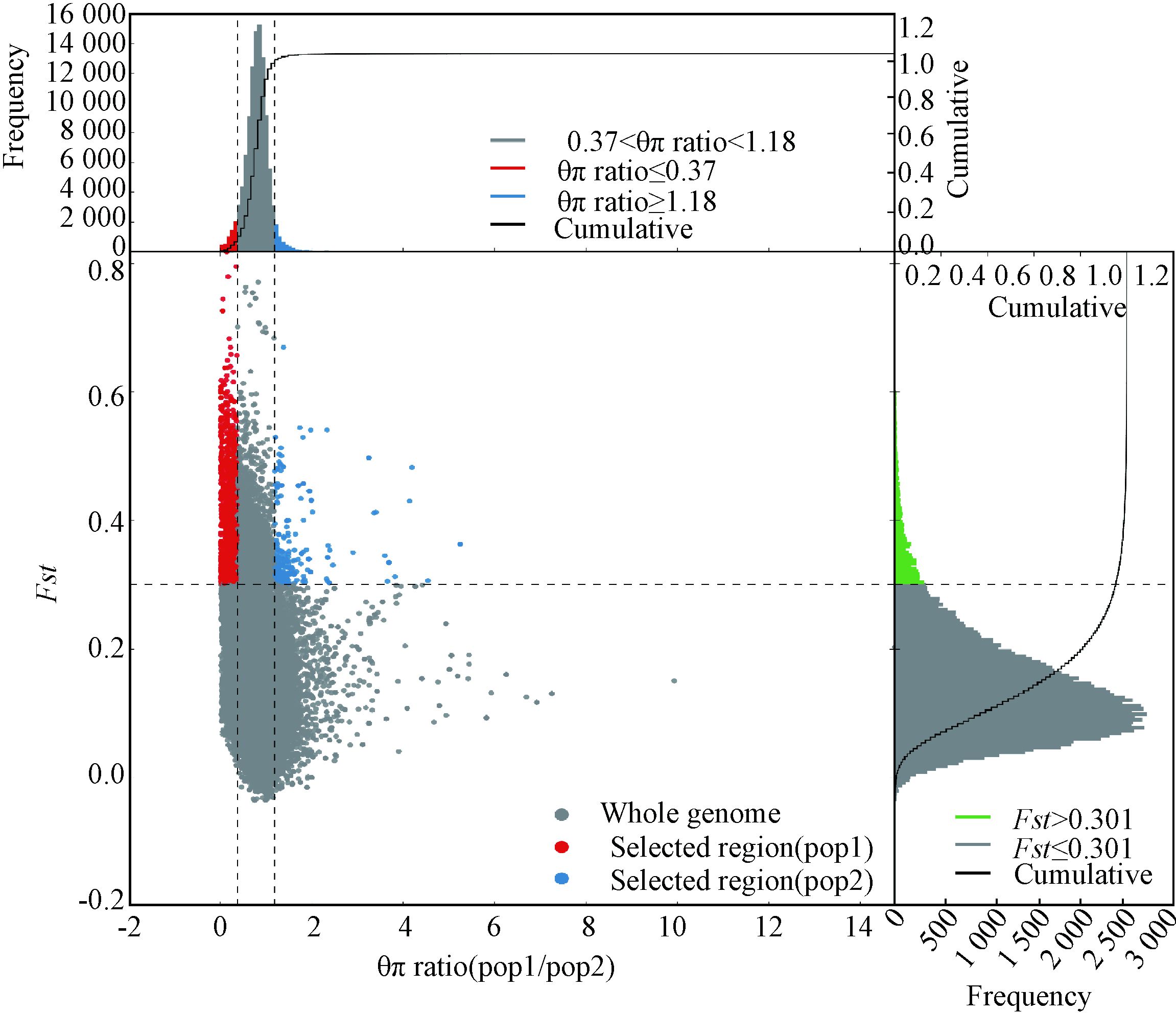
|
图中pop1表示基因库鹿苑鸡群体,pop2表示张家港鹿苑鸡群体 Pop1 represents Luyuan chicken population from national chicken genetic resources, Pop2 represents Luyuan chicken population from Zhangjiagang conservation farm 图 4 种群分化选择区域分布图 Figure 4 The regional distribution of population differentiation |
|
横坐标为GO分类;纵坐标左边为基因数目所占百分比,右边为基因数目 The horizontal coordinate is GO classification; the left side of the vertical coordinate is the percent of genes, and the right is the number of genes 图 5 区域内基因GO注释分类统计图 Figure 5 GO annotation and classification of genes in region |
|
|
表 2 受选择基因的KEGG通路注释 Table 2 KEGG pathway annotation of differential expression genes |
许多研究者开展了针对地方禽种保种效果监测与评价的研究,李慧芳等[11]采用13对微卫星标记分析了7个国家级重点保护鸡种原产地保种群与基因库异地保种群间的保种效果;赵振华等[12]利用5个微分子标记和5个血液蛋白位点标记比较了不同保种方法的保种效果;曲鲁江等[13]采用28对微卫星引物分析了大骨鸡和北京油鸡在不同保种场的保种效果;段修军等[14]用12个微卫星标记对3个世代金定鸭保种群进行了遗传检测,分析了小群保种效果。上述研究由于采用的分子标记较少,无法做到在全基因组水平上比较遗传差异、评价保种效果。
随着高通量测序技术的应用和推广,简化基因组测序技术逐渐得到大家的认可,其在人类医学、模式生物以及动植物基因定位、遗传分析等方面发挥重要作用[15-19]。RAD-seq简化基因组测序技术主要是对限制性酶切位点两端的段区域进行高通量测序[20], 此方法可大幅度降低基因组的复杂度,操作简便,同时还可不受基因组的限制,可以快速鉴定出高密度的SNPs位点,能够帮助实现遗传进化分析及重要性状候选基因的预测[21]。本研究利用RAD-seq测序在两个鹿苑鸡群体中鉴定的SNPs数量为400K左右,远高于利用微卫星等方法获得的分子标记,进一步证实RAD-seq技术可以进行高通量SNP标记开发。SNP标记数量的增加显著提升了群体遗传学统计量的度量准确性。两个鹿苑鸡群体共享SNPs位点在1、2、5、Z染色体上分布最多,这些区段是遗传多样性最为丰富的区域,根据此区域内丰富的SNP位点,可以对保种群实行遗传监测,并在世代之间进行比较分析,从而可以更全面准确的监测保种效果。
两个鹿苑鸡群体的平均杂合度Ho为0.206 8~0.211 1,低于微卫星标记所测得的鹿苑鸡群体平均杂合度(0.593~0.737)[22-24],这是由于微卫星标记位点多态性高且分布相对集中,因此平均杂合度较高;RAD-seq简化基因组测序覆盖度广,虽然群体杂合度较低但可以较全面准确的反映整个基因组多态位点情况,这也是简化基因组测序与微卫星标记分析群体遗传多样性的不同之处。两个鹿苑鸡群体近交系数水平相对较高,原因可能是对鹿苑鸡实施抢救性保护时(1996年),起始群体规模较小所导致的。较高的近交系数提示在保种过程中要确保群体规模,做好系谱记录,有效控制近交。
根据S.Wright[25]的理论,如果群体Fst值为0~0.05,说明各亚群间不存在分化;Fst值为0.05~0.15,说明各亚群间为中度分化;Fst值为0.15~0.25,则说明各亚群体间已经高度分化。本研究中两个鹿苑鸡群体的平均遗传分化指数Fst为0.0 056,同时聚类分析结果显示两个鹿苑鸡群体始终聚在一起,没有个体分离出来,表明两个群体间不存在明显遗传分化。两个鹿苑鸡群体的平均杂合度差异也不显著(P>0.05),进一步证实了两个保种单位保种现状较好。
为鉴别出两个鹿苑鸡群体间在一些染色体特定区域(位点)上存在的分化,通过Fst和θπ检验进行选择信号分析,在1、2、5、Z染色体上鉴定出58个受选择区域,筛选到96个受选择候选基因。GO和KEGG分析表明,这些差异基因主要富集在能量代谢、信号传递、应激免疫反应等调控通路。这些差异信息为针对性地开展基因库与保种场间血源补充,丰富保种群遗传多样性提供了科学依据。引起这些差异的原因有待进一步的研究证实。
4 结论基因库与保种场两个鹿苑鸡保种群没有出现明显的遗传分化现象,表明两个单位保种现状较好,可以利用此结果在世代之间进行比较,进一步分析保种效果。利用全基因组SNP标记信息可以更全面地评价保种现状、分析保种效果,研究结果为进一步优化鹿苑鸡保种技术方案提供了依据。
| [1] |
陈国宏, 王克华, 王金玉, 等.
中国禽类遗传资源[M]. 上海: 上海科学技术出版社, 2004.
CHEN G H, WANG K H, WANG J Y, et al. Poultry genetic resources in China[M]. Shanghai: Shanghai Science and Technology Press, 2004. (in Chinese) |
| [2] | HOHENLOHE P A, BASSHAM S, ETTER P D, et al. Population genomics of parallel adaptation in threespine stickleback using sequenced RAD tags[J]. PLoS Genet, 2010, 6(2): e1000862. DOI: 10.1371/journal.pgen.1000862 |
| [3] | EMERSON K J, MERZ C R, CATCHEN J M, et al. Resolving postglacial phylogeography using high-throughput sequencing[J]. Proc Natl Acad Sci U S A, 2010, 107(37): 16196–16200. DOI: 10.1073/pnas.1006538107 |
| [4] | HOHENLOHE P A, AMISH S J, CATCHEN J M, et al. Next-generation RAD sequencing identifies thousands of SNPs for assessing hybridization between rainbow and westslope cutthroat trout[J]. Mol Ecol Resour, 2011, 11(S1): 117–122. |
| [5] | CHUTIMANITSAKUN Y, NIPPER R W, CUESTA-MARCOS A, et al. Construction and application for QTL analysis of a restriction site associated DNA (RAD) linkage map in barley[J]. BMC Genomics, 2011, 12: 4. DOI: 10.1186/1471-2164-12-4 |
| [6] | PFENDER W F, SAHA M C, JOHNSON E A, et al. Mapping with RAD (restriction-site associated DNA) markers to rapidly identify QTL for stem rust resistance in Lolium perenne[J]. Theor Appl Genet, 2011, 122(8): 1467–1480. DOI: 10.1007/s00122-011-1546-3 |
| [7] | BAXTER S W, DAVEY J W, JOHNSTON J S, et al. Linkage mapping and comparative genomics using next-generation RAD sequencing of a non-model organism[J]. PLoS One, 2011, 6(4): e19315. DOI: 10.1371/journal.pone.0019315 |
| [8] | MCKENNA A, HANNA M, BANKS E, et al. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Res, 2010, 20(9): 1297–1303. DOI: 10.1101/gr.107524.110 |
| [9] | LI H, HANDSAKER B, WYSOKER A, et al. The Sequence alignment/map (SAM) format and SAM tools[J]. Transplant Proc, 1987, 19(1): 1653–1654. |
| [10] | YEH F C, YANG R C, BOYLE J, et al. POPGENE, the user-friendly shareware for population genetic analysis[R]. Alberta, Canada: Molecular Biology and Biotechnology Centre, University of Alberta, 1999: 10. |
| [11] |
李慧芳, 高玉时, 苏一军, 等. 中国地方鸡种资源不同保种方法的分子检测[J]. 云南农业大学学报, 2006, 21(2): 228–230, 245.
LI H F, GAO Y S, SU Y J, et al. Molecular evaluation of different conservation measures of Chinese indigenous chicken breeds[J]. Journal of Yunnan Agricultural University, 2006, 21(2): 228–230, 245. (in Chinese) |
| [12] |
赵振华, 黎寿丰, 吴兆林, 等. 不同保种方法对地方鸡种的保种效果分析[J]. 中国家禽, 2009, 31(14): 23–25.
ZHAO Z H, LI S F, WU Z L, et al. Effect of different conservation measures in domestic chicken[J]. China Poultry, 2009, 31(14): 23–25. (in Chinese) |
| [13] |
曲鲁江, 吴桂琴, 李显耀, 等. 采用微卫星DNA标记分析部分地方鸡种保种场的保种效果[J]. 遗传学报, 2004, 31(6): 591–595.
QU L J, WU G Q, LI X Y, et al. Conservation efficiency of local chicken breeds in different farms as revealed by microsatellite markers[J]. Acta Genetica Sinica, 2004, 31(6): 591–595. (in Chinese) |
| [14] |
段修军, 王丽华, 龚道清, 等. 利用微卫星标记检测金定鸭小群保种效果[J]. 畜牧兽医学报, 2008, 39(9): 1159–1164.
DUAN X J, WANG L H, GONG D Q, et al. Analysis on conserved effect of small population in Jinding duck using microsatellite markers[J]. Acta Veterinaria et Zootechnica Sinica, 2008, 39(9): 1159–1164. (in Chinese) |
| [15] | SIMÓN-SÁNCHEZ J, SCHULTE C, BRAS J M, et al. Genome-wide association study reveals genetic risk underlying Parkinson's disease[J]. Nat Genet, 2009, 41(12): 1308–1312. DOI: 10.1038/ng.487 |
| [16] | RIOUX J D, XAVIER R J, TAYLOR K D, et al. Genome-wide association study identifies new susceptibility loci for Crohn disease and implicates autophagy in disease pathogenesis[J]. Nat Genet, 2007, 39(5): 596–604. DOI: 10.1038/ng2032 |
| [17] | VAN HEEL D A, FRANKE L, HUNT K A, et al. A genome-wide association study for celiac disease identifies risk variants in the region harboring IL2 and IL21[J]. Nat Genet, 2007, 39(7): 827–829. DOI: 10.1038/ng2058 |
| [18] | HILL-BURNS E M, CLARK A G. Functional regulatory divergence of the innate immune system in interspecific Drosophila hybrids[J]. Mol Biol Evol, 2010, 27(11): 2596–2605. DOI: 10.1093/molbev/msq146 |
| [19] | HUANG J, ZHANG J H, LI W Z, et al. Genome-wide association analysis of ten chilling tolerance indices at the germination and seedling stages in maize[J]. J Integr Plant Biol, 2013, 55(8): 735–744. DOI: 10.1111/jipb.v55.8 |
| [20] | DAVEY J W, BLAXTER M L. RADSeq: next-generation population genetics[J]. Brief Funct Genomics, 2010, 9(5-6): 416–423. DOI: 10.1093/bfgp/elq031 |
| [21] |
汪文强, 赵生国, 马利青, 等. 动物基因组学重测序的应用研究进展[J]. 畜牧兽医学报, 2016, 47(10): 1947–1953.
WANG W Q, ZHAO S G, MA L Q, et al. The research progress and application of resequencing based on animal genomics[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(10): 1947–1953. (in Chinese) |
| [22] |
王德前. 运用微卫星标记分析地方鸡亲缘关系[D]. 扬州: 扬州大学, 2003.
WANG D Q. The genetic relationship analysis among Chinese native chicken breeds by means of microsatellite[D]. Yangzhou: Yangzhou University, 2003. (in Chinese) (in Chinese) |
| [23] |
沈见成, 陈宽维, 王金玉, 等. 利用微卫星标记分析江苏三个地方鸡品种的遗传多样性[J]. 中国家禽, 2004, 26(6): 14–16.
SHEN J C, CHEN K W, WANG J Y, et al. Analysis of genetic diversity of three Jiangsu native chickens with microsatellite markers[J]. China Poultry, 2004, 26(6): 14–16. (in Chinese) |
| [24] |
吴兆林, 高玉时, 童海兵. 鹿苑鸡微卫星和AFLP指纹分析[J]. 中国家禽, 2005, 9(1): 83–85.
WU Z L, GAO Y S, TONG H B. Analysis of genetic variation of Luyuan chicken using microsatellite and AFLP fingerprintings[J]. China Poultry, 2005, 9(1): 83–85. (in Chinese) |
| [25] | WRIGHT S. Evolution and the genetics of populations, Volume 4: variability within and among natural population[M]. Chicago: University of Chicago Press, 1978: 439-459. |



