2. 华中农业大学动物科学技术学院/动物医学院, 武汉 430070
2. College of Animal Science and Technology/College of Animal Veterinary Medicine, Huazhong Agricultural University, Wuhan 430070, China
猪产仔数受排卵率和胚胎存活率等诸多遗传和环境因素的影响。产仔数的遗传力低,传统杂交改良周期长,寻找控制猪产仔数的主效基因及其遗传标记,使用标记辅助选择加快选育进程,这对提高母猪产仔数具有重要意义。早期研究发现,ESR基因[1]与产仔数存在显著关联,随着全基因组关联(GWAS)研究的发展,一些新的突变位点如VRTN[2]、ENOX1[3]和SPESP1[4]也被发现与产仔数存在关联。同时,仍然有较多报道从基因功能来推测基因可能影响产仔数,通过扩增多态位点进行关联分析来证明SNP与产仔数的关系,如KCNC2[5]和RBP4[6]。这些研究使得产仔数可利用的标记不断增加。
ZP4(Zona pellucida sperm-binding protein 4) 是透明带蛋白基因家族的重要成员。ZP基因家族主要功能是介导受精过程中的顶体反应,即精卵结合前诱导精子穿过透明带蛋白释放顶体囊中内容物,精卵结合后释放物质阻止其他精子再与卵子结合[7]。一些研究表明,在猪、牛中ZP3/ZP4形成的结合蛋白是精卵结合的第一受体,而ZP4主要引导了ZP3/ZP4结合蛋白的形成[8-11],人ZP4基因也能介导顶体反应[12-14],由此可见,ZP4基因在精卵结合中发挥重要作用。已有报道ZP3中SNP与猪产仔数存在关联[15-16],但ZP4是否与猪产仔数存在相关还未见报道。
虽然许多基因与产仔数存在关联,是产仔数的重要候选基因,但是存在关联的位点或基因是否为因果基因仍要结合多方面证据来判断。结合群体遗传学研究,我们假设对于育种中对产仔数这一性状的持续选择压,如果基因位点突变影响到产仔数,那么选择压可能会传递到突变位点上来,即SNP可能受到选择压作用。基于这种假设,笔者利用一些群体遗传学的分析方法来检测ZP4基因多态性位点的选择压,以此来证明ZP4与产仔数的相关性。
1 材料与方法 1.1 试验群体及PCR扩增试验样本来自3个猪群体共328头母猪,其中165头大白母猪群体采自唐人神种猪育种公司,长白母猪群体116头和长大二元猪群体47头采自湖北盛龙农业科技开发有限公司。对群体采取耳静脉抽血,酚-氯仿法抽提血液全基因组DNA。记录上述母猪各胎次的总产仔数和产活仔数。
依据猪ZP4基因序列(GI:347618780, 位于57 496 103~57 503 521 bp)设计8对引物(表 1)。这8对引物的扩增产物包含了ZP4基因的12个外显子和部分内含子序列,扩增产物序列长度共4 965 bp,占ZP4基因总长度的66.9%。
|
|
表 1 扩增ZP4基因所用引物序列 Table 1 The primers designed from 12 exons of swine ZP4 gene |
扩增ZP4基因PCR程序:94 ℃预变性4 min;94 ℃变性30 s,退火温度退火30 s,72 ℃延伸30 s,34个循环;72 ℃延伸2 min。扩增ZP4的引物及退火温度见表 1。
PCR反应产物直接送生工生物工程有限公司测序。SNP检测和基因型识别利用Sequencer软件完成(http://www.genecodes.com)。
1.2 构建连锁图谱SNP位点间连锁分析利用Haploview[17]完成。
1.3 基因效应分析采用SAS9.1中的GLM程序对ZP4基因SNP与总产仔数和产活仔数进行关联分析,不同基因型对应的产仔数用“最小二乘均值±标准误(LSM±SE)”表示。模型:
Yij=μ+HYSi+Gj+eij(模型1)
Yijk=μ+HYSi+Gj+Pk+eijk(模型2)
式中,Y为性状观察值,分别对应本研究中的总产仔数和产活仔数,μ为群体均值,HYS是场年季效应,P是胎次效应,G是基因型效应,e是随机效应残差。在分析中第1胎次产仔数使用模型1,第2至最后一个胎次的产仔数使用模型2。
1.4 正选择检验方法利用lositan软件来分析SNP的选择压。中性进化条件下,虽然群体基因流会使大多数突变位点形成较为平均的遗传分化程度,但是受到约束性选择的位点会偏离群体均值形成离群现象。可以通过在基于孤岛模型下的近交系数和群体杂合度的期望分布来判断SNP位点是否受到选择作用。
多个突变位点之间可能存在连锁从而形成一些连锁区。利用Tajima′s D检验和Fu and Li′s D检验来检测每个连锁区是否受到选择[18-19]。Tajima′s D检验的主要原理是检验群体中观察到的遗传变异量与中性模型下期望的遗传变异量之间的差值。D值为负说明群体中存在纯化选择,D值为正说明群体中存在平衡选择并将突变率维持在居中水平。
2 结果 2.1 SNP等位基因及频率通过对ZP4基因序列的重测序发现,在大白猪群体、长大二元杂交群体和长白猪群体中有27个碱基位置发生突变,其中20个突变发生在内含子区域,7个位点突变发生在编码区。有3个编码区突变为非同义突变:G728A、C2183T和G5723A,分别导致所编码的氨基酸由精氨酸、脯氨酸和丙氨酸,突变为组氨酸、丝氨酸和苏氨酸,核苷酸位置以ZP4第一个外显子的第一个密码子核苷酸位置为参考值1。等位基因频率和基因型频率见表 2。
|
|
表 2 ZP4基因27个SNPs等位基因及基因型频率 Table 2 Allele and genotype frequency of 27 SNPs in ZP4 |
关联分析发现,ZP4基因的27个SNPs在大白猪群体、长白猪群体和长大二元杂交群体中与第1胎次的产活仔数和总产仔数之间关联都不显著(P>0.05)。合并第2到第6胎次的总产仔数和产活仔数进行关联分析,发现A268T在3个猪群体中均与总产仔数显著相关(P < 0.05),A268T在长白猪和大白猪群体中与产活仔数显著关联(P < 0.05),该SNP在长大二元猪群体中与产活仔数关联不显著(P>0.05,表 3)。此外C706A和G728A在长大二元群体中与产仔数存在显著关联,C-113T和G3264A在长白群体中也与产仔数存在关联,但是这些关联的显著性在其他两个群体中都没有检测到。
|
|
表 3 ZP4基因SNP与产仔数的关联分析(合并第2至6胎产仔数) Table 3 The association between ZP4 and TNB, NBA in pigs (litter size from the 2nd to 6th parities) |
对3个群体突变位点使用lositan软件模拟了中性状态下Fst和杂合性He的联合分布,突变位点离群分析的结果为Fst=0.54,显著的大于Fst模拟值(P>0.99)。这说明T2950C位点处于正选择下(图 1),推测T2950C可能有利于增强群体的适应性。
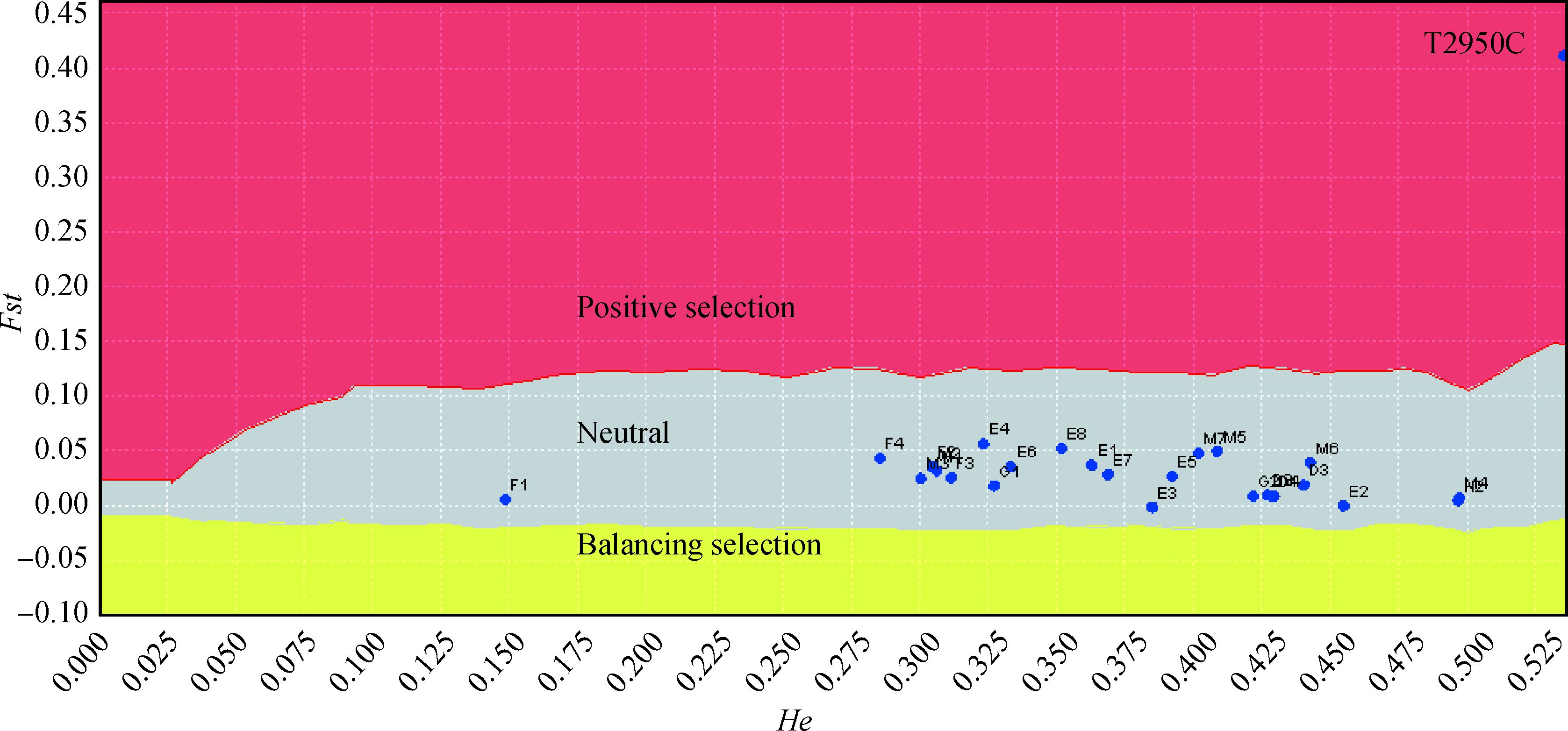
|
·Markers 图 1 突变位点离群分析 Figure 1 Outlier loci detection for selection |
SNP位点的选择压分析结果说明虽然T2950C与产仔数并不存在显著关联。由于位点间连锁现象普遍存在,检测T2950C是否与其他突变存在连锁很有必要。大白猪、长大二元猪和长白猪群体中ZP4基因27个SNPs形成了4个连锁区,每个区所包含的SNP数目不等(图 2)。第1~12个SNPs在大白猪群体中紧密连锁在一起,此区域在其他两个群体中连锁并不紧密,只有10~12 3个SNPs连锁在一起。第13~15个SNPs在3个猪群体中都连锁在一起。第16~18个SNPs在大白猪群体中独自形成一个连锁区,在长大二元猪群体中这3个SNPs和第19~23个SNPs连锁在一起,而在大白猪群体中第19~27个SNPs形成了该群体中的第4个连锁区块。第26~27个SNPs在长大二元猪群体中形成了该群体中的第4个连锁区,而在长白猪群体中第25~27个SNPs形成一个连锁区。
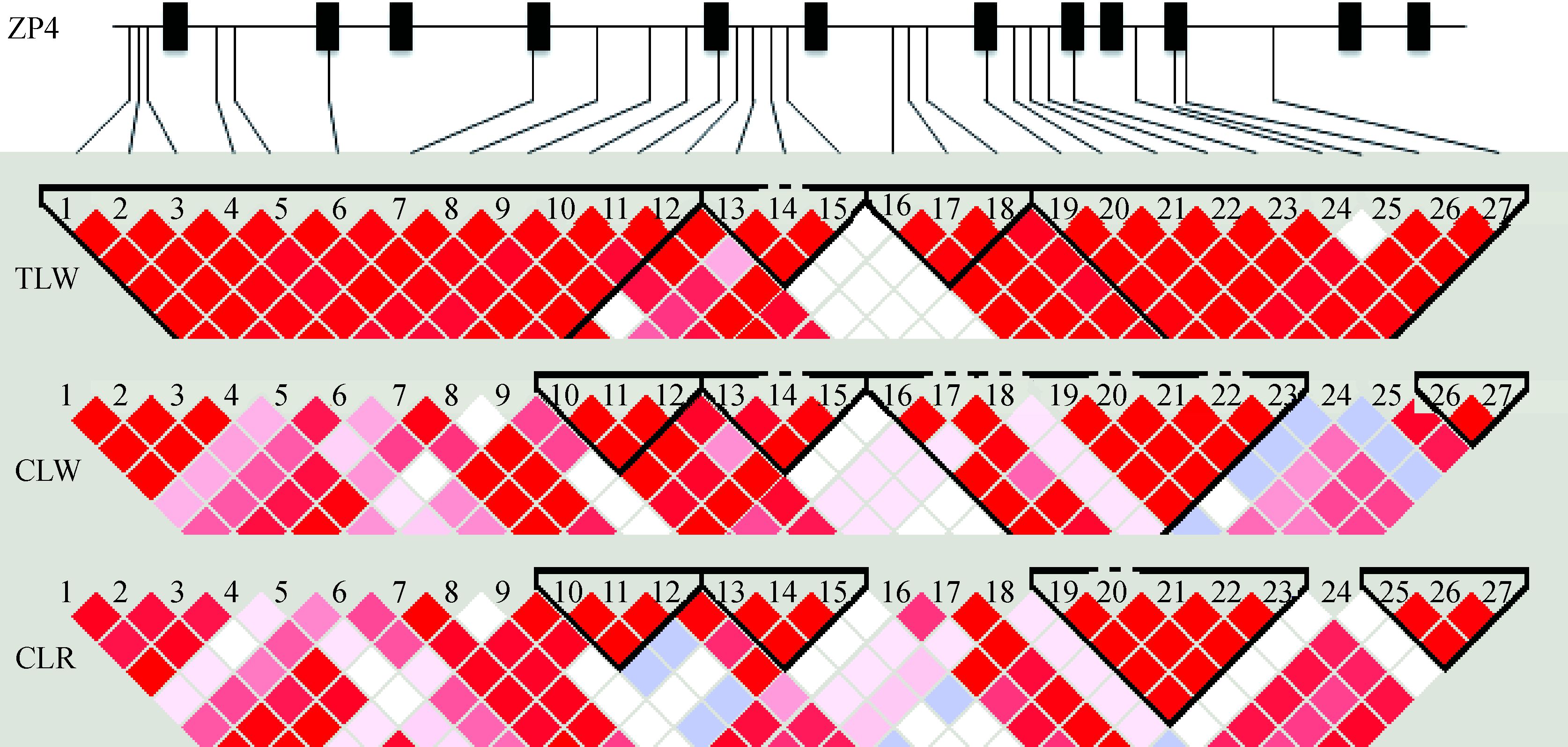
|
最上端为ZP4基因结构示意图,黑色长方形代表外显子 The top panel is gene structure of ZP4, the blacks blocks represent exon regions 图 2 ZP4基因27个SNPs在大白猪群体(TLW)、长大二元猪群体(CLW)、长白猪群体(CLR)中的连锁图谱 Figure 2 ZP4 gene structure and linkage disequilibrium (LD) structures in the 3 populations |
对图 2中每个连锁区域在3个群体的等位基因频谱进行了群体遗传学分析,发现在每个群体中的第二个连锁区域B2的Tajima′D值为正,并且检验结果显著(P>0.95)。在大白猪群体(TLW)中B1区域Tajima′D值为正,并且检验结果显著(P>0.95)。Tajima′D正值说明B2区域在群体进化过程中受到了平衡选择作用,但也不能排除群体正处于分化之中。除长大二元猪群体中B4区域外,其余区域虽然Fu和Li检验D值在3个群体中均为正值,但是均没有达到显著水平(表 4)。
|
|
表 4 ZP4多态位点Tajima′D检验和Fu and Li′s D检验 Table 4 Tajima′D testing and Fu and Li′s D testing for ZP4 in 3 populations |
B1区域的Fu and Li’s F*值只有在大白猪群体中显著大于0(P < 0.05),据此可以判断这个该区域受到了正选择。Fu′s Fs值在3个连锁区显著(P < 0.05),其中在长白猪群体和长大二元猪群体中的B2区域为正值说明这2个区域受正选择。另外,在长大二元猪群体中还检测出B3区域受到正选择而在大白猪群体中B1、B2区域均未给出显著的检测结果,这说明Fs参数可能受到群体历史等因素的干扰(表 4)。
3 讨论猪透明带蛋白基因家族有ZP2、ZP3和ZP4 3个单体基因[20],这些基因主要作为精卵结合的受体或者辅助受体在精卵结合中发挥调控作用[21]。一般ZP3作为顶体反应的第一受体,然而猪和牛的ZP3与ZP4易形成ZP3/ZP4结合体来介导精卵结合,并且主要由ZP4来引导ZP3/ZP4结合蛋白的形成[9],目前,尚无ZP4 SNP与繁殖性状关联的报道,可能与先前该基因被认为与鼠中ZP1同源而被命名为ZP1有关[10],研究发现,人ZP4侧翼序列1个SNP与青光眼(POAG)有关联[22],然而在其他群体中则关联不显著[23],ZP4的作用机制尚不明确。一些试验表明,定点突变产生人源化猪ZP4B细胞表位是可行的[24-25],在犬科动物卵母细胞胞质中使用共焦显微镜能观察到ZP4的表达高于ZP3[26],多囊卵巢综合症患者ZP4的表达量较高[27],这些证据说明了ZP4在生殖活动中的重要性。由于ZP4在精卵结合过程中的重要调控作用,一些卵透明带疫苗已经应用于抑制哺乳动物的生育力[28],猪中已报道ZP3与产仔数存在关联[15-16],本研究发现,ZP4基因SNP变异位点多达27个,多态性比ZP3更丰富,特别是A268T可以作为产仔数的候选标记。
功能涉及生殖调控的基因可能会影响物种或群体的适应性而更容易受到正选择,分子进化研究发现ZP3基因受正选择驱动[29-30],由于ZP4与ZP3形成ZP3/ZP4结合蛋白参与精卵结合过程,因此ZP4很有可能搭遗传“顺风车”效应[31-32]受到正选择。本研究中虽然ZP4基因的T2950C受到正选择作用,但是并没有发现T2950C与产仔数显著关联,而中性突变的A268T与产仔数存在显著关联。这种看似矛盾的结果在大白群体中可能是由于T2950C与A268T紧密连锁造成的(图 2),这也得到了Tajima′ D检验和Fu and Li′D检验结果的支持(表 4)。对于长白猪和长大二元猪2个群体,在前一个群体中虽然T2950C位于A268T与G3264A之间,但是在该群体中这3个位点连锁不紧密。在后一个群体中与产仔数显著关联的C706A和G728A与T2950C连锁也不紧密(图 1),因此很难推测T2950C在这2个群体中受正选择的原因。
随着高密度芯片和基因分型技术的发展,开展全基因组关联分析研究成为现实。利用GWAS发现了许多与产仔数有关的QTLs和候选基因[33],然而GWAS对SNP的选择仍然是基于连锁不平衡理论的。虽然GWAS可以发现与性状的相关区域或位点,但是检测真正的因果变异仍然很困难[34],并且GWAS研究结果重复性面临挑战,因此基于经济性和重复性考虑,开展基因SNP与性状关联研究仍然具有意义。此外,进行关联分析时结合进化压力分析对于寻找因果变异具有指导意义。
致谢: 感谢唐人神种猪育种公司和湖北盛龙农业科技开发有限公司提供试验群体。| [1] | ROTHSCHILD M F, JACOBSON C, VASKE D A, et al. The estrogen receptor locus is associated with a major gene influencing litter size in pigs[J]. Proc Natl Acad Sci USA, 1996, 93: 201–205. DOI: 10.1073/pnas.93.1.201 |
| [2] | BERGFELDER-DRüING S, GROSSE-BRINKHAUS C, LIND B, et al. A genome-wide association study in Large White and Landrace pig populations for number piglets born alive[J]. PLoS One, 2015, 10(3): e0117468. DOI: 10.1371/journal.pone.0117468 |
| [3] | SELL-KUBIAK E, DUIJVESTEIJN N, LOPES M S, et al. Genome-wide association study reveals novel loci for litter size and its variability in a Large White pig population[J]. BMC Genomics, 2015, 16: 1049. DOI: 10.1186/s12864-015-2273-y |
| [4] | GUO X, SU G, CHRISTENSEN O F, et al. Genome-wide association analyses using a Bayesian approach for litter size and piglet mortality in Danish Landrace and Yorkshire pigs[J]. BMC Genomics, 2016, 17: 468. DOI: 10.1186/s12864-016-2806-z |
| [5] | SATO S, KIKUCHI T, UEMOTO Y, et al. Effect of candidate gene polymorphisms on reproductive traits in a Large White pig population[J]. Anim Sci J, 2016, 87(12): 1455–1463. DOI: 10.1111/asj.2016.87.issue-12 |
| [6] | MARANTIDIS A, LALIOTIS G P, AVDI M. Association of RBP4 genotype with phenotypic reproductive traits of sows[J]. Genet Res Int, 2016, 2016: 4940532. |
| [7] | OKABE M. Sperm Acrosome Biogenesis and Function During Fertilization[M]. Cham: Springer International Publishing, 2016: 1-13. |
| [8] | KANAI S, KITAYAMA T, YONEZAWA N, et al. Disulfide linkage patterns of pig zona pellucida glycoproteins ZP3 and ZP4[J]. Mol Reprod Dev, 2008, 75(5): 847–856. DOI: 10.1002/(ISSN)1098-2795 |
| [9] | YONEZAWA N, KANAI-KITAYAMA S, KITAYAMA T, et al. Porcine zona pellucida glycoprotein ZP4 is responsible for the sperm-binding activity of the ZP3/ZP4 complex[J]. Zygote, 2012, 20: 389–397. DOI: 10.1017/S0967199411000608 |
| [10] | CONNER S J, LEFIÈVRE L, HUGHES D C, et al. Cracking the egg: increased complexity in the zona pellucida[J]. Human Reprod, 2005, 20(5): 1148–1152. DOI: 10.1093/humrep/deh835 |
| [11] | SUZUKI K, TATEBE N, KOJIMA S, et al. The hinge region of bovine zona pellucida glycoprotein ZP3 is involved in the formation of the sperm-binding active ZP3/ZP4 complex[J]. Biomolecules, 2015, 5(4): 3339–3353. DOI: 10.3390/biom5043339 |
| [12] | MONNÉ M, JOVINE L. A structural view of egg coat architecture and function in fertilization[J]. Biol Reprod, 2011, 85(4): 661–669. DOI: 10.1095/biolreprod.111.092098 |
| [13] | GUPTA S K, BHANDARI B, SHRESTHA A, et al. Mammalian zona pellucida glycoproteins: structure and function during fertilization[J]. Cell Tissue Res, 2012, 349: 665–678. DOI: 10.1007/s00441-011-1319-y |
| [14] | PRASAD SV S S, CARINO C, WANG N, et al. Structure and function of the proteins of the mammalian Zona pellucida[J]. Cells Tissues Organs, 2000, 166: 148. DOI: 10.1159/000016730 |
| [15] | YUAN J F, JAFER O, AFFARA N A, et al. Association of four new single-nucleotide polymorphisms in follicle-stimulating hormone receptor and zona pellucida glycoprotein with reproductive traits in pigs[J]. Animal, 2007: 1249–1253. |
| [16] | YUAN J F, MOAEEN-UD-DIN M, GONG Y Z, et al. Identification of mutations of zona pellucida glycoprotein (ZP3) and its association with pig reproductive traits[J]. J Anim Breed Genet, 2007, 124(3): 144–149. DOI: 10.1111/jbg.2007.124.issue-3 |
| [17] | BARRETT J C, FRY B, MALLER J, et al. Haploview: analysis and visualization of LD and haplotype maps[J]. Bioinformatics, 2005, 21(2): 263–265. DOI: 10.1093/bioinformatics/bth457 |
| [18] | TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585–595. |
| [19] | FU Y X, LI W H. Statistical tests of neutrality of mutations[J]. Genetics, 1993, 133(3): 693–709. |
| [20] | GOUDET G, MUGNIER S, CALLEBAUT I, et al. Phylogenetic analysis and identification of pseudogenes reveal a progressive loss of zona pellucida genes during evolution of vertebrates[J]. Biol Reprod, 2008, 78(5): 796–806. DOI: 10.1095/biolreprod.107.064568 |
| [21] | GUPTA S K, BHANDARI B. Acrosome reaction: relevance of zona pellucida glycoproteins[J]. Asian J Androl, 2011, 13(1): 97–105. DOI: 10.1038/aja.2010.72 |
| [22] | NAKANO M, IKEDA Y, TANIGUCHI T, et al. Three susceptible loci associated with primary open-angle glaucoma identified by genome-wide association study in a Japanese population[J]. Proc Natl Acad Sci U S A, 2009, 106(31): 12838–12842. DOI: 10.1073/pnas.0906397106 |
| [23] | KONDKAR A A, EDWARD N B, KALANTAN H, et al. Lack of association between polymorphism rs540782 and primary open angle glaucoma in Saudi patients[J]. J Neg Res BioMed, 2017, 16(1): 3. DOI: 10.1186/s12952-017-0068-1 |
| [24] |
袁敏敏, 李大金, 周雷, 等. 猪ZP4B细胞表位人源化定点突变[J]. 中国免疫学杂志, 2004, 20: 184–186.
YUAN M M, LI D J, ZHOU L, et al. Porcine ZP4B cell epitope humanized site -directed mutagenesis[J]. Chinese Journal of Immunology, 2004, 20: 184–186. DOI: 10.3321/j.issn:1000-484X.2004.03.010 (in Chinese) |
| [25] |
袁敏敏, 李大金, 周雷, 等. B细胞表位人源化的猪ZP4在大肠杆菌中的表达与鉴定[J]. 中国免疫学杂志, 2005, 21: 826–827.
YUAN M M, LI D J, ZHOU L, et al. Expression and identification of human ZP4 in B cell epitopes in Escherichia[J]. Chinese Journal of Immunology, 2005, 21: 826–827. DOI: 10.3321/j.issn:1000-484X.2005.11.006 (in Chinese) |
| [26] | BUKOWSKA D, KEMPISTY B, ZIÓKOWSKA A, et al. Expression and distribution of zona pellucida proteins 3 and 4 in morphologically abnormal canine oocytes: a confocal microscopic observation-based study[J]. Medycyna Weterynaryjna, 2016, 72: 68–74. |
| [27] | MECZEKALSKI B, NAWROT R, NOWAK W, et al. Study on the zona pellucida 4 (ZP4) gene sequence and its expression in the ovaries of patients with polycystic ovary syndrome[J]. J Endocrinol Invest, 2015, 38(7): 791–797. DOI: 10.1007/s40618-015-0260-4 |
| [28] |
陈芳芳, 张玲. 女性免疫避孕的研究进展[J]. 生殖与避孕, 2016, 36(7): 585–588.
CHEN F F, ZHANG L. Advance of female contraceptive vaccine[J]. Reproduction & Contraception, 2016, 36(7): 585–588. DOI: 10.7669/j.issn.0253-357X.2016.07.0585 (in Chinese) |
| [29] | AMARAL A R, MOLLER L M, BEHEREGARAY L B, et al. Evolution of 2 reproductive proteins, ZP3 and PKDREJ, in cetaceans[J]. J Hered, 2011, 102(3): 275–282. DOI: 10.1093/jhered/esq131 |
| [30] | CHEN S, COSTA V, BEJA-PEREIRA A. Evolutionary patterns of two major reproduction candidate genes (Zp2 and Zp3) reveal no contribution to reproductive isolation between bovine species[J]. BMC Evol Biol, 2011, 11: 24. DOI: 10.1186/1471-2148-11-24 |
| [31] | KIM Y, STEPHAN W. Joint effects of genetic hitchhiking and background selection on neutral variation[J]. Genetics, 2000, 155(3): 1415–1427. |
| [32] | RENZETTE N, KOWALIK T F, JENSEN J D. On the relative roles of background selection and genetic hitchhiking in shaping human cytomegalovirus genetic diversity[J]. Mol Ecol, 2016, 25(1): 403–413. DOI: 10.1111/mec.13331 |
| [33] |
赵谦, 浦亚斌, 关伟军, 等. 猪重要性状全基因组关联分析的研究进展[J]. 畜牧兽医学报, 2015, 46(6): 873–881.
ZHAO Q, PU Y B, GUAN W J, et al. Research progress of genome-wide association studies for important traits in pig[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(6): 873–881. (in Chinese) |
| [34] |
郝兴杰, 胡林, 张淑君. 全基因组关联分析方法的研究进展[J]. 畜牧兽医学报, 2016, 47(2): 213–217.
HAO X J, HU L, ZHANG S J. Progresses in research of genome-wide association study methods[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(2): 213–217. (in Chinese) |



