生物机体是一个复杂的体系,研究组织的发育特性或应激反应可能需要同时考虑上千个基因,并在基因调控系统背景下,考察基因与发育之间的联系。参与生命活动的物质相互联系,形成一个复杂的网络系统。分子网络种类和层次较多,如基因网络(相关或共表达网络)、蛋白质相互作用网络、代谢网络和信号传导网络等。这些网络内的互作关系均可在基因共表达网络中体现。
基于基因表达相似性的网络称为基因共表达网络。机体内具有相同功能或参与相同调控通路的基因,往往呈现类似的转录或表达谱。如以图来描述这种关系,可用图中的节点代表基因,以连接节点的边表示两个基因之间的共表达相互作用关系[1-2]。共表达的基因常在网络中形成致密的连通子图,一般对应于具有相似功能的基因集团或信号通路,具有特定的生物学功能,构成网络的局部亚结构-模块[3-6],可从系统水平揭示基因间的相互作用关系,有助于研究者深入理解基因间的相互作用机制,找到共表达基因的调控中心;可发现与性状高相关的模块,有利于快速发现与目标性状相关的关键基因,尤其是控制发育与繁殖的关键基因[1, 7]。
基因共表达网络的构建主要分为两步,第一是对所有候选基因进行相似性评估,第二是通过阈值的选择确定共表达网络的边。研究者已开发出多种构建基因共表达网络的方法[8-13],如权重基因共表达网络分析技术(WGCNA, weighted gene co-expression network analysis)。
猪是重要的经济动物,将共表达网络技术应用于猪的遗传育种领域,有利于挖掘关键基因,探讨调控机理,促进遗传改良。本研究以猪的RNA-seq数据为基础,利用WGCNA技术构建基因共表达网络模块,对模块进行功能、组织定位和可视化分析,旨在为探究猪特有的基因资源信息提供新思路和新途径。
1 材料与方法 1.1 试验材料从NCBI的SRA(Sequencing Read Achieve)数据库,下载试验ERP002055(SRA accession)的RNA-seq数据(https://www.ncbi.nlm.nih.gov/sra/)[14],共含100个样本,来自2头不相关的1岁雄性长白猪(Landrace boars),涉及10种组织(心、肝、脾、肺、肾、大脑额叶皮层、大脑枕叶皮层、背最长肌、下丘脑和小脑),每个组织10个重复,测序reads总量为2.23亿个碱基,基因组覆盖度达到80%。基于这100个RNA-Seq数据建立猪的多组织基因共表达网络。
1.2 试验方法 1.2.1 转录本的组装和量化首先,利用fastq-dump将NCBI下载的SRA文件转换成fastq文件格式[15]。然后,以默认参数,使用IlluQC.pl对测序reads进行筛选,要求平均reads质量大于20[16],再通过TopHat 2.1.0将筛选得到的RNA-seq reads比对到猪的基因组(Sus Sscrofa 10.2)[17],然后将比对结果提交到Cufflinks 2.0.2进行转录本的组装[18]。以FPKM(Fragments per kilobase of exon per million fragments mapped)值标准化基因的表达水平[19]。
1.2.2 基因和样本的筛选计算每个基因在样本中表达水平的标准差,并根据其分布选取标准差大于5的基因,作为构建共表达网络的候选基因。对样本进行聚类,删除离群样本。另外,为降低缺失值对后续计算的影响,转录本在同一组织中必须在3个以上样本中表达。本研究的统计分析均基于R语言程序包。
1.2.3 基因共表达网络模块构建基于R语言中WGCNA程序包构建猪的基因共表达网络,步骤:(1)为了使网络符合无尺度拓扑特性,使用pickSoftThreshold()函数来分析网络拓扑并帮助选择合适的β值;(2)基于无尺度拓扑标准(Scale-free topology criterion),选取合适的β值作为构建网络的软阈值(Soft-thresholding power),并评估模块中所有基因的平均连通性(Mean connectivity);(3)在此β值下,通过计算所有基因之间的皮尔逊相关性(Pearson’s correlations)创建邻接矩阵(Adjacency matrix);(4)利用邻接矩阵,计算TOM(Topological overlap measure)值,其代表基因之间的相似度;(5)计算拓扑差异值dissTOM=1-TOM,并用层次聚类法创建基因的聚类树状图;(6)模块构建,利用动态剪枝法(Dynamic tree cut)检测具有高关联性的基因团,并将其定义为模块,模块的最小基因数为30,将模块以颜色标记,便于模块的识别和可视化分析;(7)使用函数moduleEigengenes()将上一步定义的模块进行聚类分析,选取0.25的距离值,即0.75的相关性来整合相似的模块,使用mergeCloseModules()函数合并相似模块。详细过程参见文献[20]。
1.2.4 模块的功能富集和组织定位对模块利用DAVID 6.8进行基因本体论的生物学过程富集和组织定位分析[21-22],利用KOBAS 2.0进行KEGG通路和疾病的富集分析[23],设置FDR(Fasle discovery rate)P < 0.01作为显著性比例。以猪的基因组水平为富集统计分析的背景(Background)。利用Cytoscape 3.3.0可视化网络[24]。
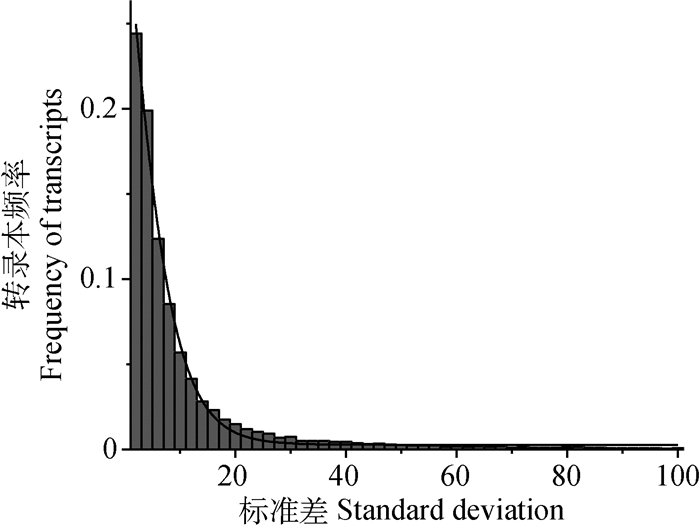
2 结果 2.1 猪的基因表达变异度分析转录本表达水平的标准差分布分析显示,所有转录本表达的变异度呈现幂率分布(Power-law)(图 1),以标准差大于5的13 433个转录本为构建网络的候选基因。
|
图 1 转录本表达水平变异分布 Figure 1 The distribution of variation of transcription expression level |
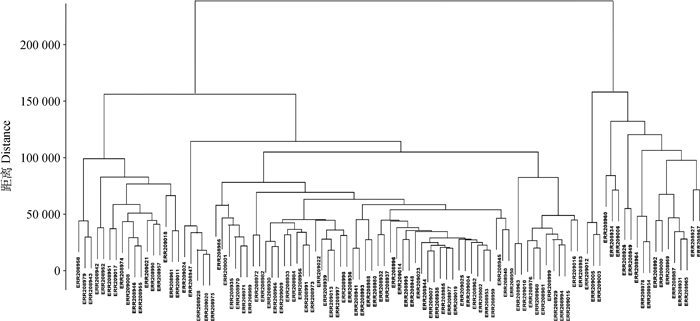
样本的聚类分析显示(图 2),样本明显分为2个分支,较大的分支有81个样本,较小的分支有19个样本,样本的聚集度较好,表明样本间遗传背景相近。过滤缺失值较多的转录本后,生成了在100个样本中12 165个转录本的表达数据集。
|
图 2 样本聚类分析 Figure 2 The analysis of samples clustering |
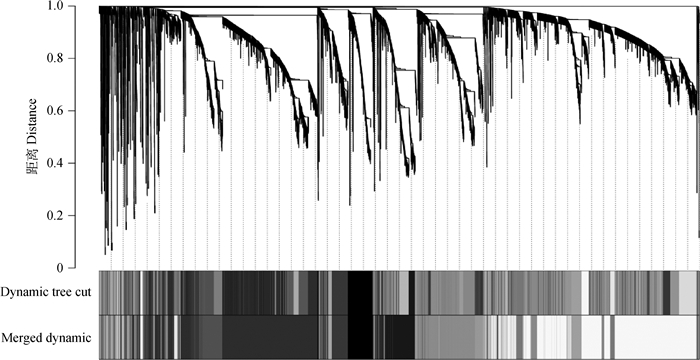
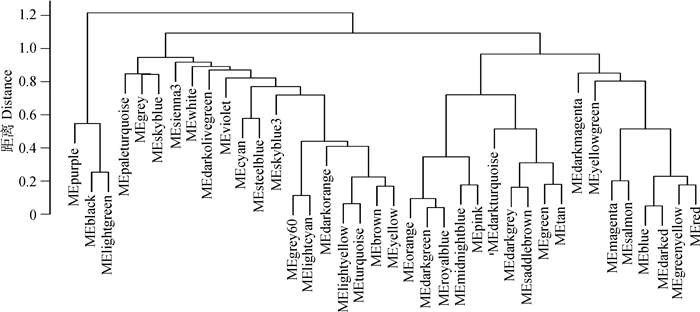
拓扑分析表明,当β=6时,无尺度拓扑拟合指数(R2)接近0.9,显示网络接近为无尺度网络。从而,以β=6作为构建网络的软阈值。在该软阈值下,网络节点度的平均值为15。基于聚类和动态剪枝法,将高相关性基因聚集为模块,12 165个基因被划分为38个模块(图 3 Dynamic tree cut部分)。图 3以不同颜色可视化了这些模块的形成;再对这38个模块进行聚类,显示有许多模块之间的距离很近,即有些模块之间具有较高的相关性(图 4),进而选取0.25的距离值,即0.75的相关性标准,整合相关性较高的模块。最后,共整合成24个模块(图 3 Merged dynamic部分),基本信息见表 1。
|
Dynamic tree cut.初步构建的模块;Merged dynamic.整合的模块 Dynamic tree cut. Primary constructed modules; Merged dynamic. Merged modules 图 3 转录本的聚类与模块构建 Figure 3 The cluster of transcriptions and construction of modules |
|
图 4 模块的聚类分析 Figure 4 The clustering analysis of modules |
|
|
表 1 猪基因共表达网络模块信息 Table 1 The information of gene co-expression network modules in pig |
模块所包含基因数目差别较大,最小的skyblue 3模块包含31个转录本,而最大的lightyellow模块则包含3 794个转录本(表 1)。
2.4 模块功能的特异性对24个模块分别进行生物学过程、KEGG通路和疾病的富集分析,显示不同的模块所富集的功能存在明显的差异,例如不同模块对应不同器官的发育阶段、不同的代谢通路等(图 5)。
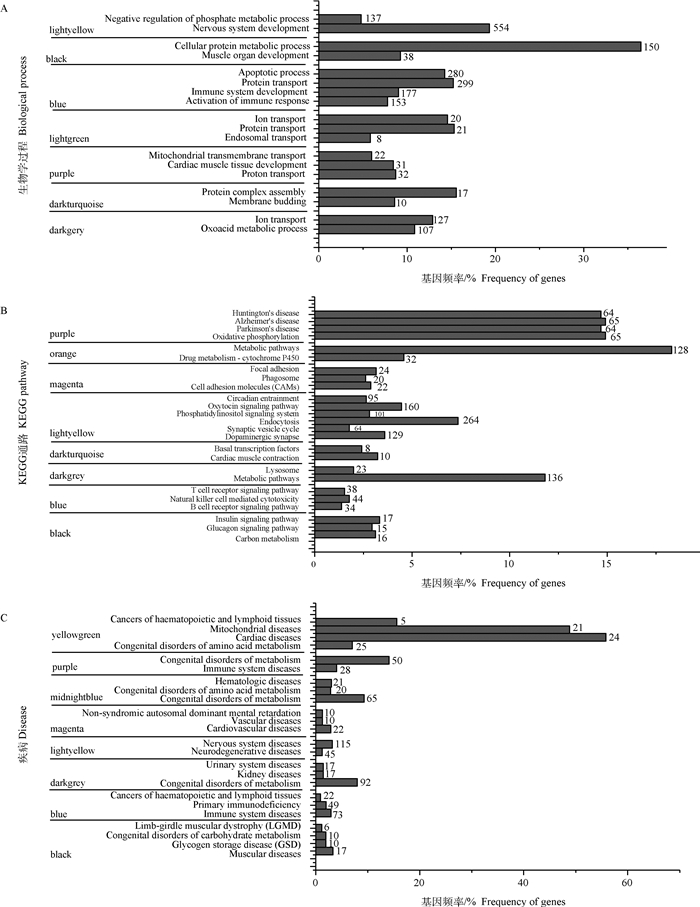
|
每个柱子上的数字表示模块内基因富集的数目 The digits in columns indicate number of genes enriched in modules 图 5 模块的功能富集分析 Figure 5 The analysis of functional enrichment in modules |
生物学过程的功能富集显示(图 5A),大多数模块能显著富集一个或几个生物学过程(PFDR < 0.01)。例如,在blue模块中,基因富集于细胞免疫反应的相关生物学过程,而lightyellow则主要和神经系统的发育相关。基于生物学过程富集分析,将模块分为了4种类型:(1)基础代谢相关的模块;(2)物质运输相关的模块;(3)机体免疫相关的模块;(4)组织和器官发育相关的模块。不同模块富集的功能可能相互重叠,但大多数模块具有特定的生物学功能,表明模块在功能上具有一定的特异性。
2.4.2 KEGG通路和疾病富集分析KEGG通路富集分析表明,大多数模块显著与一些代谢通路相关(PFDR < 0.01),表现出模块代谢通路的特异性。例如,darkgrey模块显著与氨基酸的代谢通路相关,而black模块则富集到糖代谢通路(图 5B)。
疾病富集分析表明,模块疾病的特异性表现为同一模块通常富集同一类型或相同组织的疾病。例如,black显著与肌肉和糖代谢疾病相关,因为肌肉运动消耗大量的能量,糖代谢受阻则无法提供充足的能量,从而导致肌肉相关疾病的发生。其他模块也具有相似的特征,如神经系统疾病与肌肉萎缩(lightyellow模块)、免疫缺陷和先天性代谢疾病(midnightblue模块)(图 5C)。
大多数模块虽然富集多个生物学功能,但仍在生物学过程、代谢通路和疾病分析中表现一定的功能特异性。
2.5 模块功能的协同性 2.5.1 模块间功能的协同性在4种类型的模块中,基础代谢相关的模块为细胞提供生命活动所必须的能量和物质[25],如grey模块富集的磷酸化作用,为lightyellow模块富集的神经发育和信号传递提供能量;而物质运输的相关模块,主要负责细胞将能量和物质分配给不同的生理过程,以便完成细胞的发育、免疫应答等生物学过程,如darkgery模块富集的物质转运和white模块富集的细胞通讯(Cell communication),为blue模块的细胞免疫运输必要的免疫球蛋白;而darkturquoise模块的蛋白质的膜结合、翻译和组装功能为lightgreen模块执行蛋白质运输提供了物质基础。
2.5.2 模块内功能的协同性图 5A显示,在blue模块富集了不同免疫应答途径,以及purple模块中富集的各种神经退行性疾病。这表明在基因表达过程中,同一模块内的基因不仅在转录水平上具有显著地相关性,也直观地表现为功能上的协同性。KEGG通路富集结果(图 5B)显示,blue模块含执行免疫应答相关的信号通路。这些信号通路之间,存在密切的联系,具有相互调节、相互配合的特点。例如,blue模块中大量基因在免疫细胞受体信号通路中起作用,无论是体液免疫(B细胞受体信号通路),还是细胞免疫(T细胞受体信号通路),以及其他免疫过程,都要经过calcium、PI3K-Akt、MAPK和Ubiquitin mediated信号通路等,才能起到免疫效果。如在细胞免疫反应中,MAPK信号通路中的Ras蛋白与Raf相结合活化,进而激活蛋白激酶MEK并被转入细胞核,激活涉及增殖反应的转录因子,促进免疫细胞在短期内大量增殖[26]。图 5C显示,在purple模块中,该模块显著与氧化磷酸化(Oxidative phosphorylation)相关。除此之外,该模块还显著与帕金森病(Parkinson’s disease)、阿尔茨海默症(Alzheimer’s disease)以及亨廷顿病(Huntington’s每个柱子上的数字表示模块内基因富集的数目The digits in columns indicate number of genes enriched in modules disease)有关。从KEGG代谢通路图可以看出,导致这3种疾病发生的主要原因是神经细胞的退化与死亡,在这些过程中氧化磷酸化起到重要的作用[27]。神经信号的传递需要耗费大量的能量,而随着神经细胞的退化,细胞内磷酸化水平也将逐渐降低,进而不能为神经细胞间的信号传递提供能量,最终导致神经细胞的死亡[28]。
图 6A显示了blue模块中5个Ras蛋白家族成员的共表达网络图,Ras蛋白处于该网络的中心,都具有较高的连通性,它们之间分享大量相同的共表达基因(网络内圈部分,下同),说明它们可能共同调控相同基因的表达,网络中的基因都与Ras蛋白存在紧密的共表达关系,表明Ras蛋白在免疫应答反应中的重要性。这说明在模块中,基因功能之间存在显著的协同性,某些信号通路的开启或关闭将会影响其上游或下游的功能,尤其是可能导致某些疾病的发生。在blue模块中,NDUFV1、NDUFV2和NDUFV3都是组成NADH脱氢酶(NADH dehydrogenase)的核心亚基之一,这3个基因的关闭或失调都有可能直接影响氧化磷酸化过程中的电子传递,从而导致能量代谢受阻,神经细胞信号传递不畅,进而引起如阿尔茨海默症类型的神经退行性疾病[29]。图 6B显示,purple模块中3个NADH脱氢酶核心亚基的共表达网络,3个基因也处于网络的中心,它们除了分享一些共表达基因外,还有大量特异性的共表达基因(图 6B外圈部分,下同),表明NADH脱氢酶的3个亚基可能具有功能的特异性,在不同的细胞或环境发挥质子传递作用[30]。图 6C显示了SIX1和PRKAG3基因的共表达网络。这2个基因已经有了较为深入的研究,但还有许多与产肉性状相关的基因还未被充分挖掘。图 6D显示了在lightyellow模块中催产素(OXT)和催产素受体(OXTR)的共表达网络,表明催产素和其受体受到许多基因的作用,催产素途径受到许多基因的调节。因此,在遗传育种中要兼顾考虑这些基因间的相互作用。
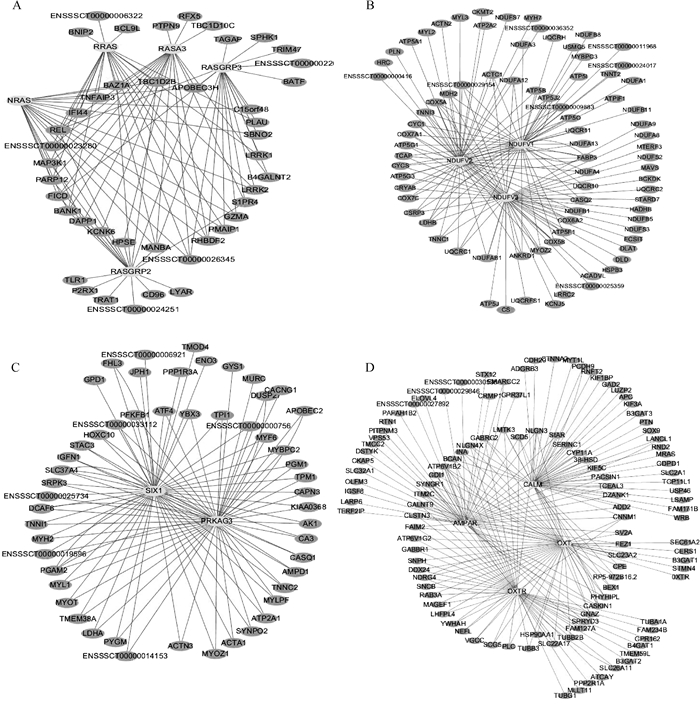
|
A.Ras蛋白家族的共表达网络;B.NADH脱氢酶核心亚基的共表达网络;C.肌肉发育相关基因的共表达网络;D.生殖相关基因的共表达网络。网络中只列出权重较高的共表达基因 A. The co-expression network of Ras superfamily; B. The co-expression network of NADH dehydrogenase subunits; C. The co-expression network of genes related to muscle development; D. The co-expression network of genes related to reproduction. Co-expression genes with higher weight were selected in network 图 6 基因共表达网络 Figure 6 The gene co-expression networks |
对模块内基因的组织定位分析表明,模块具有一定的组织特异性,即模块内基因主要富集到一个或几个组织中。虽然有些模块内基因可在多个组织中表达,但模块整体却显示具有组织特异性。这些模块主要定位于肌肉、脑和生殖等组织中(表 2)。
|
|
表 2 模块组织定位 Table 2 The tissue location of modules |
基因共表达网络分析是基于系统生物学方法,将具有协同表达特征的基因进行聚类,以网络模块的形式表现基因的共调控特性[31]。模块间具有功能和组织特异性,但模块内基因通常执行相似的或具有协同性的功能,同一模块中不同功能或信号通路之间具有显著的上、下游调控关系。网络模块可为猪遗传育种等研究提供新思路。
为了了解猪的基因共表达网络模块的功能特征,本研究利用猪的RNA-seq数据,通过WGCNA技术建立猪的权重基因共表达网络模块,将猪的基因转录组分为24个模块。模块中所包含的基因数目存在较大的差异,可能与不同功能所需的基因数量不同有关。通过对每个模块执行功能富集分析,将模块分为4种类型,并发现模块间具有功能的特异性,表现为不同模块与不同的生命活动相关;而模块内功能具有协同性,表现为模块内富集的不同代谢通路需要相同基因参与。模块在功能上具有特异性和协同性的特征,与共表达网络的拓扑原理相一致[32],这一特点可以辅助挖掘功能相关的一系列基因,为基因的调控网络研究提供一个较为快捷的思路。
本研究中,有些模块主要定位于肌肉组织中,如black模块,这和功能富集的结果一致,表明该模块主要作用于肌肉组织的发育,且该模块内的基因主要在肌肉组织中发挥作用。例如,black模块中SIX1基因主要与胚胎成肌细胞分化和细胞增殖相关[33];酸肉基因PRKAG3则是影响猪肉品质的主效应基因之一[34-35]。以模块作为基因分类和功能识别的框架,利用功能关联原则(Guilt by association)和模块内研究较透彻的基因,借助其与其他基因之间的网络关系,推测其他基因的功能,有利于快速鉴定新目标基因。另外,purple模块也定位于肌肉组织,但其功能分析显示,其主要与神经退行性疾病相关,这可能是由于这些疾病最终都会导致肌肉萎缩,和运动神经受损有关,例如帕金森病通常表现为肌强直等肌肉损伤症状[36-37]。
一些模块定位到子宫和睾丸等组织,与猪的生殖相关(表 2)。虽然lightyellow定位于脑组织,但一些下丘脑分泌的激素对猪的生殖和哺乳具有重要作用。例如,lightyellow模块富集的催产素信号途径(Oxytocin signaling pathway)和昼夜节律(Circadian entrainment)都与猪的生殖相关。催产素信号途径产生的催产素在分娩时引发子宫收缩,刺激乳汁排出[38-39]。模块的组织定位分析发现,模块具有组织特异性。模块的组织定位结合功能分析有利于较快的了解模块内基因的基本表达情况,辅助育种过程中筛选目标基因。如在lightyellow模块中,昼夜节律对动物的发情周期、卵巢卵泡发育与排卵、睾丸精子发生、受精和胚胎着床调节非常关键。图 6D表示了昼夜节律的相关基因CALM和AMPAR的共表达网络,这2个基因的异常导致细胞内外的基因表达和物质运输发生紊乱,如在卵巢的周期性变化过程中,会引起类固醇合成相关基因StAR、P450(CYP11A)、3β-HSD在发情期表达量显著升高[40]。关键基因的异常表达会导致共表达的基因异常,模块的这些特征可为猪的生产和生殖相关研究提供获选基因资源。
样本的选择是模块功能富集的一个重要的因素。本研究样本涉及脑部组织,包括下丘脑、额叶皮层、枕叶皮层和小脑。功能分析显示,最大的lightyellow模块主要涉及神经系统发育和调节相关的功能,并且有多个模块被定位到脑部相关组织中,这可能和样本中包含较多的脑组织样本有关。在一些较小的模块中,模块往往富集具有特异性的功能,例如cyan和grey60模块主要与神经系统有关;而在较大的模块中,除了富集模块特有的功能外,还通常和基础代谢相关,如lightyellow模块富集神经系统的发育和死亡,以及磷酸化和物质运输等功能相互协调。
4 结论本研究构建了24个猪多组织基因共表达网络模块,模块间表现功能和组织特异性,模块内表现功能协同性,富集了许多与功能相关的候选基因,可为探究中国地方猪品种特有的基因资源信息、丰富分子育种理论奠定基础。
| [1] | TALUKDAR H A, ASL F H, JAIN R K, et al. Cross-tissue regulatory gene networks in coronary artery disease[J]. Cell Syst, 2016, 2(3): 196–208. DOI: 10.1016/j.cels.2016.02.002 |
| [2] | ALBERT R. Scale-free networks in cell biology[J]. J Cell Sci, 2005, 118(Pt 21): 4947–4957. |
| [3] | WEIRAUCH M T. Gene coexpression networks for the analysis of DNA microarray data[M]//DEHMER M, EMMERT-STREIB F, GRABER A, et al. Applied Statistics for Network Biology: Methods in Systems Biology. Weinhiem: Wiley-VCH Verlag GmbH & Co. KGaA, 2011: 215-250. |
| [4] | BARABÁSI A L, OLTVAI Z N. Network biology: Understanding the cell's functional organization[J]. Nat Rev Genet, 2004, 5(2): 101–113. DOI: 10.1038/nrg1272 |
| [5] | HARTWELL L H, HOPFIELD J J, LEIBLER S, et al. From molecular to modular cell biology[J]. Nature, 1999, 402(6761 Suppl): C47–C52. |
| [6] | STUART J M, SEGAL E, KOLLER D, et al. A gene-coexpression network for global discovery of conserved genetic modules[J]. Science, 2003, 302(5643): 249–255. DOI: 10.1126/science.1087447 |
| [7] | KOGELMAN L J A, CIRERA S, ZHERNAKOVA D V, et al. Identification of co-expression gene networks, regulatory genes and pathways for obesity based on adipose tissue RNA Sequencing in a porcine model[J]. BMC Med Genomics, 2014, 7(1): 57–73. DOI: 10.1186/1755-8794-7-57 |
| [8] | JOSHI A, DE SMET R, MARCHAL K, et al. Module networks revisited: Computational assessment and prioritization of model predictions[J]. Bioinformatics, 2009, 25(4): 490–496. DOI: 10.1093/bioinformatics/btn658 |
| [9] | KADARMIDEEN H N, WATSON-HAIGH N S. Building gene co-expression networks using transcriptomics data for systems biology investigations: Comparison of methods using microarray data[J]. Bioinformation, 2012, 8(18): 855–861. DOI: 10.6026/bioinformation |
| [10] | ZHAO W, LANGFELDER P, FULLER T, et al. Weighted gene coexpression network analysis: State of the art[J]. J Biopharm Stat, 2010, 20(2): 281–300. DOI: 10.1080/10543400903572753 |
| [11] | LI B S, TSOI L C, SWINDELL W R, et al. Transcriptome analysis of psoriasis in a large case-control sample: RNA-Seq provides insights into disease mechanisms[J]. J Invest Dermatol, 2014, 134(7): 1828–1838. DOI: 10.1038/jid.2014.28 |
| [12] | LEE J H, GAO C, PENG G D, et al. Analysis of transcriptome complexity through RNA sequencing in normal and failing murine hearts[J]. Circ Res, 2011, 109(12): 1332–1341. DOI: 10.1161/CIRCRESAHA.111.249433 |
| [13] | DARLINGTON T M, EHRINGER M A, LARSON C, et al. Transcriptome analysis of inbred long sleep and inbred short sleep mice[J]. Genes Brain Behav, 2013, 12(2): 263–274. DOI: 10.1111/gbb.2013.12.issue-2 |
| [14] | FARAJZADEH L, HORNSHøJ H, MOMENI J, et al. Pairwise comparisons of ten porcine tissues identify differential transcriptional regulation at the gene, isoform, promoter and transcription start site level[J]. Biochem Biophys Res Commun, 2013, 438(2): 346–352. DOI: 10.1016/j.bbrc.2013.07.074 |
| [15] | KODAMA Y, SHUMWAY M, LEINONEN R. The sequence read archive: Explosive growth of sequencing data[J]. Nucleic Acids Res, 2012, 40(D1): D54–D56. DOI: 10.1093/nar/gkr854 |
| [16] | PATEL R K, JAIN M. NGS QC toolkit: A toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7(2): e30619. DOI: 10.1371/journal.pone.0030619 |
| [17] | TRAPNELL C, PACHTER L, SALZBERG S L. TopHat: Discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25(9): 1105–1111. DOI: 10.1093/bioinformatics/btp120 |
| [18] | TRAPNELL C, WILLIAMS B A, PERTEA G. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nat Biotechnol, 2010, 28(5): 511–515. DOI: 10.1038/nbt.1621 |
| [19] | MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nat Methods, 2008, 5(7): 621–628. DOI: 10.1038/nmeth.1226 |
| [20] | LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9(1): 559. DOI: 10.1186/1471-2105-9-559 |
| [21] | HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44–57. |
| [22] | HUANG D W, SHERMAN B T, LEMPICKI R A. Bioinformatics enrichment tools: Paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Res, 2009, 37(1): 1–13. DOI: 10.1093/nar/gkn923 |
| [23] | XIE C, MAO X Z, HUANG J J, et al. KOBAS 2.0: A web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Res, 2011, 39(Suppl. 2): W316–W322. |
| [24] | SHANNON P, MARKIEL A, OZIER O, et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks[J]. Genome Res, 2003, 13(11): 2498–2504. DOI: 10.1101/gr.1239303 |
| [25] | NELSON D L, COX M M. Lehninger Principles of Biochemistry[M]. 5th ed. New York: W. H. Freeman and Company, 2008. |
| [26] | BOILLY B, VERCOUTTER-EDOUART A S, HONDERMARCK H, et al. FGF signals for cell proliferation and migration through different pathways[J]. Cytokine Growth Factor Rev, 2000, 11(4): 295–302. DOI: 10.1016/S1359-6101(00)00014-9 |
| [27] | MATTSON M P. Neurodegenerative Disease[M]//MOOREN F C. Encyclopedia of Exercise Medicine in Health and Disease. Berlin Heidelberg: Springer, 2012. |
| [28] | PIZZA V, AGRESTA A, D′ACUNTO C W, et al. Neuroinflamm-aging and neurodegenerative diseases: An overview[J]. CNS Neurol Disord Drug Targets, 2011, 10(5): 621–634. DOI: 10.2174/187152711796235014 |
| [29] | KIRBY D M, SALEMI R, SUGIANA C, et al. NDUFS6 mutations are a novel cause of lethal neonatal mitochondrial complex I deficiency[J]. J Clin Invest, 2004, 114(6): 837–845. DOI: 10.1172/JCI20683 |
| [30] | BRANDT U. Energy converting NADH: Quinone oxidoreductase (complex I)[J]. Annu Rev Biochem, 2006, 75: 69–92. DOI: 10.1146/annurev.biochem.75.103004.142539 |
| [31] | D'HAESELEER P, LIANG S D, SOMOGYI R. Genetic network inference: From co-expression clustering to reverse engineering[J]. Bioinformatics, 2000, 16(8): 707–726. DOI: 10.1093/bioinformatics/16.8.707 |
| [32] | DAS S, MEHER P K, RAI A, et al. Statistical approaches for gene selection, hub gene identification and module interaction in gene co-expression network analysis: An application to aluminum stress in soybean (Glycine max L.)[J]. PLoS One, 2017, 12(1): e0169605. DOI: 10.1371/journal.pone.0169605 |
| [33] | WU W J, HUANG R H, WU Q H, et al. The role of Six1 in the genesis of muscle cell and skeletal muscle development[J]. Int J Biol Sci, 2014, 10(9): 983–989. DOI: 10.7150/ijbs.9442 |
| [34] |
陶新, 邓波, 门小明, 等. 不同猪种肉质相关基因Hal、RN和FTO的多态性研究[J]. 畜牧兽医学报, 2012, 43(5): 676–683.
TAO X, DENG B, MEN X M, et al. Research on polymorphism of Hal, RN and FTO genes related to meat quality in different pig breeds[J]. Acta Veterinaria et Zootechnica Sinica, 2012, 43(5): 676–683. (in Chinese) |
| [35] | MILAN D, JEON J T, LOOFT C, et al. A mutation in PRKAG3 associated with excess glycogen content in pig skeletal muscle[J]. Science, 2000, 288(5469): 1248–1251. DOI: 10.1126/science.288.5469.1248 |
| [36] | KALIA L V, LANG A E. Parkinson's disease[J]. Lancet, 2015, 386(9996): 896–912. DOI: 10.1016/S0140-6736(14)61393-3 |
| [37] | POEWE W. Psychosis in Parkinson's disease[J]. Mov Disord, 2003, 18(S6): 80–87. DOI: 10.1002/(ISSN)1531-8257 |
| [38] | GRINEVICH V, KNOBLOCH-BOLLMANN H S, ELIAVA M, et al. Assembling the puzzle: Pathways of oxytocin signaling in the brain[J]. Biol Psychiatry, 2015, 79(3): 155–164. |
| [39] | CHATTERJEE O, PATIL K, SAHU A, et al. An overview of the oxytocin-oxytocin receptor signaling network[J]. J Cell Commun Signal, 2016, 10(4): 355–360. DOI: 10.1007/s12079-016-0353-7 |
| [40] | SAKAI T, ISHIDA N. Circadian rhythms of female mating activity governed by clock genes in Drosophila[J]. Proc Natl Acad Sci U S A, 2001, 98(16): 9221–9225. DOI: 10.1073/pnas.151443298 |



