2. 生态环境部南京环境科学研究所, 江苏 南京 210042
2. Nanjing Institute of Environmental Sciences, Ministry of Ecology and Environment, Nanjing 210042, China
腈是一类含氰基基团(C—R≡N)的有机化合物,种类繁多且在自然界分布广泛,例如苯甲腈天然存在于焙烤过的花生、榛子、可可和奶制品等。苯甲腈类化合物也是重要的有机合成原料和中间体,在医药、农药、染料、香料、缓蚀剂及液晶材料方面有着广泛的应用。苯甲腈类具有毒性,能引起动物组织的痉挛、神经麻痹等[1];另外,它对环境也有一定的危害,可造成水体和土壤的污染。已报道的消除苯甲腈污染的方法中以化学降解法居多,如LIU等[2]和ZHANG等[3]用化学吸附的方式降低苯甲腈的污染;孔令仁等[4]利用光化学法降解水体中的苯甲腈类污染物。相比于化学降解法,微生物酶解法降解腈类污染物具有高效性、高选择性、反应条件温和、环境污染小等优点[5],因此利用微生物酶系降解腈类污染物越来越受到青睐。
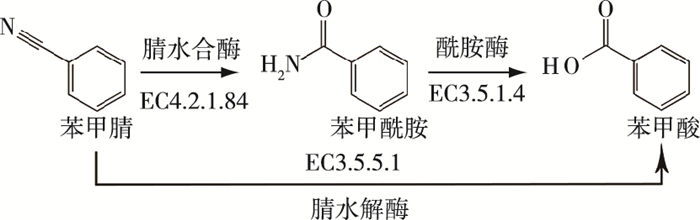
微生物降解腈类由腈转化酶(nitrile-converting enzyme)催化(图 1)[6]。根据酶的不同,将腈类降解分为2条途径,一是在腈水解酶(nitrilase,EC3.5.5.1)催化下由腈类直接转化为羧酸,这种酶在微生物中普遍存在,例如,周冬杰等[7]利用高通量筛选法从土壤中分离的金黄杆菌属(Chryseobacterium)、芽孢杆菌属(Bacillus)、葡萄球菌属(Staphylococcus)和寡养单胞菌属(Stenotrophomonas)可利用该酶降解对羟基苯甲腈。Paraburkholderia graminis腈水解酶具有广泛的底物选择性,可降解脂肪族丙烯腈、杂环的3-氰基吡啶及芳香族苯乙腈和苯甲腈[8]。DENNETT等[9]研究表明Pyrococcus sp. M24D13腈水解酶对苯甲腈和丁腈具有很高的降解活性。另一条腈类降解途径是由腈水合酶(nitrile hydratase,EC4.2.1.84)和酰胺酶(amidase,EC3.5.1.4)联合作用,将腈类先降解为相应酰胺,然后由酰胺酶催化产生羧酸和氨气,如Corynebacterium sp. C5利用这一途径降解苯甲腈[10];VESELA等[11]报道的Rhodococcus rhodochrous PA-34和R. erythropolis A4腈水合酶-酰胺酶可降解苯甲腈及苯甲腈类除草剂。相对于腈水解酶途径,利用腈水合酶-酰胺酶途径降解苯甲腈的报道较少,特别是对这一途径中苯甲酰胺酶的研究鲜见报道[12]。
|
图 1 苯甲腈的降解途径 Fig. 1 The pathways for degradation of benzonitrile |
笔者发现草木樨箭菌(以前称为草木樨中华根瘤菌)Ensifer meliloti CGMCC 7333可降解含氰基的新烟碱类杀虫剂啶虫脒和噻虫啉,并证明其腈水合酶将这2种农药代谢为酰胺类产物[13]。采用全基因组已测序的En. meliloti 1021代谢氰基化合物,发现En. meliloti 1021可降解苯甲腈。En. meliloti 1021是一种根瘤菌,不仅可以与草木樨属(Melilotus)的植物结瘤,还可与苜蓿属(Medicago)和葫芦巴属(Trigonella)内的某些种共生固氮[14],因此,En. meliloti 1021既可作为有机污染物降解菌株用于消除环境污染,又可固氮用作生物氮肥。为此,笔者开展了En. meliloti 1021降解苯甲腈的代谢途径和代谢酶基因的克隆表达研究,以期为消除环境中苯甲腈污染物提供理论依据和参考价值。
1 材料与方法 1.1 试验材料 1.1.1 试剂苯甲腈、苯甲酰胺、苯甲酸、3-吲哚乙腈、3-吲哚乙酰胺、3-氰基吡啶、烟酰胺、敌草腈、氟虫腈、溴虫腈、丙烯酰胺、乙酰胺和苯乙酰胺购于美国Sigma-Aldrich公司(试剂纯度w在98%以上),色谱级乙腈购于美国Tedia公司。
1.1.2 试验菌株和质粒En. meliloti 1021由实验室尚广东老师提供,Escherichia coli Rosetta (DE3)和质粒pET28a (+)由实验室保存。
1.1.3 培养基改良LB培养基(g·L-1):蛋白胨10.0 g,酵母粉5.0 g,NaCl 10.0 g,pH值7.5,加入终浓度为0.1 mmol·L-1CoCl2。固体培养基加入琼脂粉20.0 g。
磷酸盐缓冲液(PBS,50 mmol·L-1,pH=7.5):NaH2PO4·2H2O 1.25 g,Na2HPO4·12H2O 15.04 g。
氯霉素母液(Chl,34 mg·mL-1):称1 g氯霉素溶于29.4 mL无水乙醇,过滤除菌。
卡那霉素母液(Kan,30 mg·mL-1):称0.75 g卡那霉素溶于25 mL无菌水,过滤除菌。
诱导剂异丙基硫代半乳糖苷母液(IPTG,100 mmol·L-1):称2.38 g IPTG溶于100 mL无菌水中,过滤除菌,分装于1.5 mL离心管中,-20 ℃条件下储存备用。
1.2 试验方法 1.2.1 En. meliloti 1021静息细胞降解苯甲腈和苯甲酰胺用无菌接种环挑取-80 ℃条件下保藏的En. meliloti 1021,在LB固体平板上4区划线,于恒温培养箱中30 ℃倒置培养至长出单菌落。挑取单菌落接种到含3 mL液体LB培养基的试管中,30 ℃、200 r·min-1培养24 h。按φ=1%的接种量接种到200 mL LB培养基中,30 ℃、200 r·min-1摇床培养12 h。用50 mL离心管收集菌液(OD600=5.0),4 ℃,以8 000 r·min-1离心5 min(离心半径为10.8 cm),弃去上清液,用PBS清洗菌体2次,向清洗好的菌体中加入15 mL PBS,涡旋振荡混匀,平均分到3个50 mL离心管中,然后加入终浓度为9.7 mmol·L-1苯甲腈;同样的方式收集菌体,然后加入1.6 mmol·L-1苯甲酰胺转化液;设置底物对照(底物+转化液)和菌体对照(菌体+转化液),保鲜膜封口并留1个小孔,在每次取样前需补足转化过程中蒸发掉的水分。取样1 mL,以12 000 r·min-1离心10 min(离心半径为7.7 cm),上清液用乙腈(终止反应)稀释10倍。经0.22 μm孔径的滤膜过滤后用高效液相色谱仪(HPLC)分析。
1.2.2 生物信息学分析在美国国家生物技术信息中心网站(NCBI)上检索En. meliloti 1021的全基因组(NC_003047.1),分别搜索腈水合酶(nitrile hydratase)、酰胺酶(amidase)和腈水解酶(nitrilase),下载相关序列。搜索苯甲酰胺水解酶(benzamide amidase或benzamide amidohydrolase)蛋白序列并下载,用MEGA 6.0软件、Neighbor-joining法构建苯甲酰胺水解酶的系统发育树并进行聚类分析。
1.2.3 En. meliloti 1021腈水合酶和酰胺酶的克隆表达PCR扩增所用引物由上海生工生物工程有限公司合成,序列见表 1,引物上含EcoRI和XhoI限制性酶切位点。PCR体系为10×LA缓冲液2 μL,25 mmol·L-1 MgCl2 2 μL,dNTP 2 μL,En. meliloti 1021基因组DNA 2 μL,LA taq DNA聚合酶0.2 μL,ddH2O 11 μL,上下游引物分别0.4 μL;扩增程序如下:95 ℃预变性5 min,95 ℃变性5 s,退火温度58 ℃反应40 s,72 ℃延伸2 min,共30个循环,最后72 ℃延伸10 min。克隆的目的基因片段用EcoRI和XhoI双酶切,琼脂糖凝胶电泳验证,然后用ClonExpressII一步克隆试剂盒(南京诺唯赞生物科技有限公司)将目的片段连入含相同酶切位点的pET28a质粒。E. coli Rosetta (DE3)用氯化钙处理成感受态[15]。根据文献[16]报道的方法在E.coli Rosetta (DE3)中进行En. meliloti 1021腈水合酶和酰胺酶的质粒构建。挑取阳性克隆,送南京斯普金生物有限公司测序。
|
|
表 1 文中所用引物 Table 1 PCR primers used in this study |
E.coli Rosetta(DE3)重组菌株用Kan和Chl抗性的LB培养至OD600=0.6~0.7,然后加入终浓度为0.2 mmol·L-1的IPTG,30 ℃诱导6 h(OD600=3)。以8 000 r·min-1离心5 min(离心半径为10.8 cm),收集菌体,PBS清洗后分别加入97 mmol·L-1苯甲腈和1.6 mmol·L-1苯甲酰胺底物,设置底物对照和菌体对照。取样1 mL,以12 000 r·min-1离心10 min(离心半径为7.7 cm),苯甲腈样品稀释100倍,苯甲酰胺样品稀释10倍,经0.22 μm孔径滤膜过滤后进行HPLC分析。另外,取5 mL菌液收集菌体,超声破碎后进行聚丙烯酰胺凝胶电泳(SDS-PAGE)检测蛋白表达情况。
1.2.4 重组的过表达菌株对不同底物的转化腈水合酶重组菌株转化杂环的3-氰基吡啶和3-吲哚乙腈,除草剂敌草腈、氟虫腈和溴虫腈,浓度均为9.7 mmol·L-1;酰胺酶重组菌株转化杂环的烟酰胺和3-吲哚乙酰胺,芳香族的苯乙酰胺,脂肪族的丙烯酰胺和乙酰胺,浓度均为1.6 mmol·L-1。用50 mL离心管收集5 mL诱导至OD600=3的重组菌株,PBS清洗2次,然后加入5 mL相应的底物,30 ℃,200 r·min-1进行转化。腈类底物密封离心管口转化15 min,每隔5 min取样;酰胺类底物转化5 h,每隔1 h取样;样品以12 000 r·min-1离心10 min(离心半径为7.7 cm),上清液稀释适当倍数,经0.22 μm孔径的微孔滤膜过滤后HPLC分析。腈水合酶酶活定义为每分钟生成1 μmol酰胺产物所需的细胞液的量为1个NHase活力单位(U)。
1.3 HPLC分析采用安捷伦1200型高效液相色谱仪进行分析。HPLC条件:Agilent HC-C18柱(4.6 mm×250 mm,5 μm);进样体积为20 μL,柱温为30 ℃,检测器为Agilent G1314A紫外检测器;流动相A为经0.22 μm孔径滤膜过滤的双蒸水,然后加入φ=0.1‰色谱级乙酸,流动相B为色谱级乙腈;A和B的比例见表 2,流速为1 mL·min-1,数据由Agilent Chem-station工作站自动采集。
|
|
表 2 底物的HPLC分析条件 Table 2 HPLC conditions for the substrates used in this study |
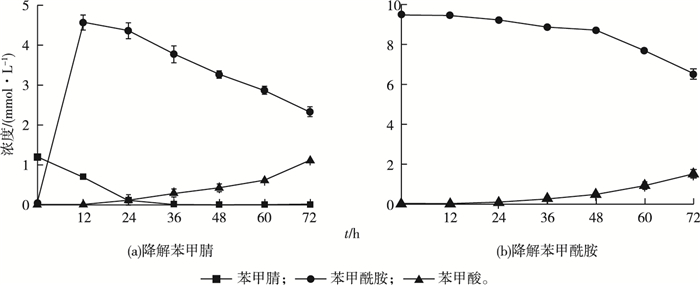
由图 2可知,以苯甲腈为底物时,24 h之前培养液中检测到苯甲腈和苯甲酰胺,在12 h苯甲酰胺的产量达到最高为4.35 mmol·L-1,24 h之后苯甲酰胺含量降低,并且检测到苯甲酸的产生,72 h时苯甲酸的产量达1.11 mmol·L-1。以苯甲酰胺为底物时,En. meliloti 1021可将苯甲酰胺代谢为苯甲酸,转化72 h,苯甲酸产量为1.51 mmol·L-1且有增加的趋势(图 2)。
|
图 2 Ensifer meliloti 1021静息细胞对苯甲腈和苯甲酰胺的降解过程 Fig. 2 Time course of benzonitrile and benzamide degradation by Ensifer meliloti 1021 |
En. meliloti 1021以苯甲腈为底物可产生苯甲酰胺和苯甲酸,以苯甲酰胺为底物可产生苯甲酸;查找En. meliloti 1021的全基因组(NC_003047.1)发现没有腈水解酶(nitrilase)基因,因此确定苯甲腈的降解途径是由腈水合酶和酰胺酶完成。微生物体内的腈水合酶和酰胺酶通常情况下共表达完成生物转化[17],很多研究已报道这2种腈转化酶对腈类的代谢,例如,对脂肪族乙腈[18]及丁腈[10, 19]等的降解,对芳香族腈类的转化中以3-氰基吡啶居多[20-21];对降解芳香族苯甲腈的报道很少[22],BRANDÁO等[23]曾报道过放线菌属(Actinomycetes)腈水合酶对苯甲腈的转化,但转化率比其他腈类低。
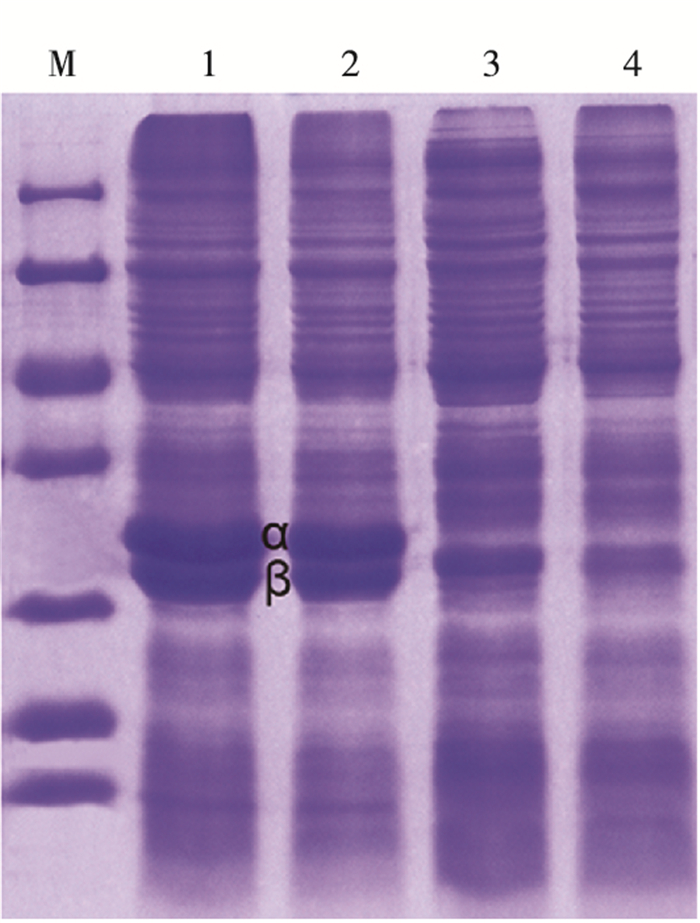
2.2 腈水合酶的克隆表达及其表达菌株降解苯甲腈用引物Nha-F和Nha-R扩增获得基因长度1 671 bp的En. meliloti 1021腈水合酶基因序列,包括3个基因:α亚基基因(642 bp)、β亚基基因(660 bp)和激活蛋白基因(387 bp),其中α亚基和β亚基有4 bp的ATGA重叠序列,β亚基和激活蛋白有14 bp的TTGAACACGCTTAG重叠序列。3个亚基编码的蛋白分子量分别为21、23和14 kDa,等电点为5.45。表达蛋白中激活蛋白没有明显的条带(图 3),这可能是由于激活蛋白位于α亚基和β亚基下游,与T7启动子序列相隔较远,虽然激活蛋白的表达量低,但是对腈水合酶的活性有极大的促进作用[24]。En. meliloti 1021腈水合酶异源表达菌株在5 min之内将97 mmol·L-1的苯甲腈转化为58.6 mmol·L-1苯甲酰胺,降解率达90%,10 min苯甲腈基本检测不到,生成66.9 mmol·L-1苯甲酰胺,降解效果比一些未经改造的微生物酶解和化学法消除苯甲腈更佳[2, 25-26]。
|
M—116,66.2,45,35,25,18.4,14.4;1—E. coli Rosetta(DE3)- pET28a-腈水合酶全蛋白;2—E. coli Rosetta(DE3)-pET28a-腈水合酶可溶性蛋白;3—E. coli Rosetta(DE3)-pET28a全蛋白;4—E. coli Rosetta(DE3)-pET28a可溶性蛋白。 图 3 SDS-PAGE分析Ensifer meliloti 1021腈水合酶 Fig. 3 SDS-PAGE of Ensifer meliloti 1021 NHase gene cluster over-expressed in Escheriachia coli Rosetta(DE3) |
将En. meliloti 1021基因组中的酰胺酶与Genbank中检索到的苯甲酰胺水解酶蛋白序列构建系统发育树(图 4)。

|
图 4 Ensifer meliloti 1021酰胺酶的系统进化树 Fig. 4 Neighbor-joining phylogenetic tree of Ensifer meliloti 1021 amidase |
选择与3个苯甲酰胺水解酶(BAJ23981.1、BAJ23976.1和BAJ23974.1)亲缘关系近的登录号为AAK65414.1、CAC47672.1、CAC41593.1和AAK65936.1的酰胺酶进行基因克隆和表达,用Ami1引物克隆的CAC47672.1酰胺酶诱导表达后具有苯甲酰胺酶活性,该酰胺酶有434个氨基酸组成,分子量约47 kDa,等电点为5.37。
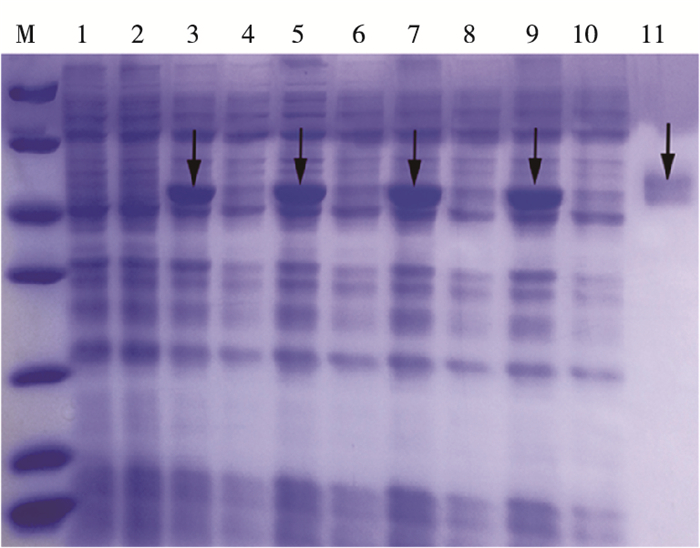
异源表达的酰胺酶转化1.6 mmol·L-1苯甲酰胺时,4 h产生的苯甲酸为13.3 nmol·L-1,而对照组含空载质粒的E. coli Rosetta未能检测到苯甲酸的生成。SDS-PAGE显示苯甲酰胺酶的可溶性低,通过降低诱导温度(由30降至20 ℃)和诱导剂浓度(由0.2降至0.1 mmol·L-1),以及将pET28a更换为pET32a质粒,其可溶性并未得到改善,进一步采用变性和复性的方式纯化包涵体,蛋白可溶性有所提高(图 5),但是纯化蛋白未能检测到活性。目前,Rhodococcus sp. R312和R. erythropolis AJ270中的酰胺酶异源表达菌株对苯甲酰胺有活性,其中E. coli BL21(DE3)表达的R. erythropolis AJ270酰胺酶在LB培养基中培养,重组菌株最高酶活为2.45 U·mL[12]。有关苯甲酰胺酶的表达仍需进一步优化,以提高蛋白的可溶性和酶活性。
|
M—116,66.2,45,35,25,18.4,14.4;1—E. coli Rosetta(DE3)-pET28a全蛋白;2—E. coli Rosetta(DE3)-pET28a可溶性蛋白;3—E. coli Rosetta(DE3)-pET28a-酰胺酶全蛋白;4—E. coli Rosetta(DE3)-pET28a-酰胺酶可溶性蛋白;5—20 ℃诱导E. coli Rosetta(DE3)-pET28a-酰胺酶全蛋白;6—20 ℃诱导E. coli Rosetta(DE3)-pET28a-酰胺酶可溶性蛋白;7—0.1 mmol·L-1IPTG诱导E. coli Rosetta(DE3)-pET28a-酰胺酶全蛋白;8—0.1 mmol·L-1IPTG诱导E. coli Rosetta(DE3)-pET28a-酰胺酶可溶性蛋白;9—E. coli Rosetta(DE3)-pET32a-酰胺酶全蛋白;10—E. coli Rosetta(DE3)-pET32a-酰胺酶可溶性蛋白;11—镍柱纯化的包涵体内酰胺酶。箭头处为目的蛋白。 图 5 SDS-PAGE分析Ensifer meliloti 1021酰胺酶 Fig. 5 SDS-PAGE of Ensifer meliloti 1021 amidase gene over-expressed in Escheriachia coli Rosetta(DE3) |
HPLC分析E. coli Rosetta(DE3)腈水合酶重组菌株除降解苯甲腈外,还可转化3-吲哚乙腈和3-氰基吡啶,但对敌草腈、溴虫腈和氟虫腈没有降解活性。表达的腈水合酶对3-吲哚乙腈的相对酶活性为14.5%(苯甲腈的腈水合酶酶活定义为100%),对3-氰基吡啶相对酶活为318%。含酰胺酶的E. coli Rosetta(DE3)重组菌株可代谢苯甲酰胺,对以上提到的其他几种含酰胺基团的化合物均无活性,这与R. erythropolis AJ270酰胺酶的异源表达菌株具有宽泛的底物选择性不同[12]。
3 结论(1) En. meliloti 1021可降解苯甲腈生成苯甲酰胺和苯甲酸,其降解途径中的酶系为腈水合酶-酰胺酶。
(2) 腈水合酶过表达的E. coli可在5 min内降解90%的浓度为97 mmol·L-1的苯甲腈,反应10 min后基本上检测不到苯甲腈的存在,产生66.9 mmol·L-1苯甲酰胺。
(3) 通过系统进化树分析筛选并克隆和表达了En. meliloti 1021代谢苯甲酰胺的酰胺酶,该酶具有严格的苯甲酰胺底物特异性。
| [1] |
LI T G, LIU J X, BAI R B, et al. Biodegradation of Organonitriles by Adapted Activated Sludge Consortium With Acetonitrile-Degrading Microorganisms[J]. Water Research, 2007, 41(15): 3465-3473. DOI:10.1016/j.watres.2007.04.033 (  0) 0) |
| [2] |
LIU A A, ZHI H, DENG G H, et al. Adsorption of Benzonitrile at the Air/Water Interface Studied by Sum Frequency Generation Spectroscopy[J]. Chinese Science Bulletin, 2013, 58(13): 1529-1535. DOI:10.1007/s11434-013-5763-9 (  0) 0) |
| [3] |
ZHANG P, SHENG G Y, FENG Y C, et al. Predominance of Char Sorption Over Substrate Concentration and Soil pH in Influencing Biodegradation of Benzonitrile[J]. Biodegradation, 2006, 17(1): 1-8. DOI:10.1007/s10532-005-1919-x (  0) 0) |
| [4] |
孔令仁, 石正方, 王晓书, 等. 苯甲腈类有机污染物在水中光化学降解的研究[J]. 南京大学学报(自然科学), 1995, 31(3): 404-410. [ KONG Ling-ren, SHI Zheng-fang, WANG Xiao-shu, et al. A Funcitional Study on Photodecomposition of Benzonitrilc compounds in Aqueous Solution[J]. Journal of Nanjing University(Natural Sciences Edition), 1995, 31(3): 404-410.] (  0) 0) |
| [5] |
BANERJEE A, SHARMA R, BANERJEE U. Retraction Noe to:The Nitrile-Degrading Enzymes:Current Status and Future Prospect[J]. Applied Microbiology and Biotechnology, 2002, 60(1/2): 33-44. (  0) 0) |
| [6] |
PACE H C, BRENNER C. The Nitrilase Superfamily:Classification, Structure and Function[J]. Genome Biology, 2001, 2(1): 1-9. (  0) 0) |
| [7] |
周冬杰, 欧阳立明, 许建和, 等. 土壤中腈水解酶产生菌的快速筛选[J]. 华东理工大学学报(自然科学版), 2009, 35(4): 545-548. [ ZHOU Dong-jie, OUYANG Li-ming, XU Jian-he, et al. Rapid Screening of Nitrilase Producing Strains From Soil[J]. Journal of East China University of Science and Technology (Natural Science Edition), 2009, 35(4): 545-548. DOI:10.3969/j.issn.1006-3080.2009.04.010] (  0) 0) |
| [8] |
FAN H Y, CHEN L F, SUN H H, et al. Characterization of a Novel Nitrilase, BGC4, From Paraburkholderia graminis Showing Wide-Spectrum Substrate Specificity, a Potential Versatile Biocatalyst for the Degradation of Nitriles[J]. Biotechnology Letters, 2017, 39(11): 1725-1731. DOI:10.1007/s10529-017-2410-6 (  0) 0) |
| [9] |
DENNETT G V, BLAMEY J M. A New Thermophilic Nitrilase From an Antarctic Hyperthermophilic Microorganism[J]. Frontiers in Bioengineering and Biotechnology, 2016, 4(5): 1-9. (  0) 0) |
| [10] |
TANI Y, KURIHARA M, NISHISE H. Characterization of Nitrile Hydratase and Amidase, Which are Responsible for the Conversion of Dinitriles to Mononitriles, From Corynebacterium sp.[J]. Agricultural and Biological Chemistry, 1989, 53(12): 3151-3158. DOI:10.1080/00021369.1989.10869858 (  0) 0) |
| [11] |
VESELA B A, PELANTOVÁ H, ŠULC M, et al. Biotransformation of Benzonitrile Herbicides via the Nitrile Hydratase-Amidase Pathway in Rhodococci[J]. Journal of Industrial Microbiology and Biotechnology, 2012, 39(12): 1811-1819. DOI:10.1007/s10295-012-1184-z (  0) 0) |
| [12] |
XUE Z Q, CHAO Y P, WANG D X, et al. Overexpression of a Recombinant Amidase in a Complex Auto-Inducing Culture:Purification, Biochemical Characterization, and Regio-and Stereoselectivity[J]. Journal of Industrial Microbiology & Biotechnology, 2011, 38(12): 1931-1938. (  0) 0) |
| [13] |
SUN S L, LU T Q, YANG W L, et al. Characterization of a Versatile Nitrile Hydratase of the Neonicotinoid Thiacloprid-Degrading Bacterium Ensifer meliloti CGMCC 7333[J]. RSC Advances, 2016, 6(19): 15501-15508. DOI:10.1039/C5RA27966F (  0) 0) |
| [14] |
LAJUDIED P. Polyphasic Taxonomy of Rhizobia:Emendation of the Genus Sinorhizobium and Description of Sinorhizobium meliloti comb.nov., Sinorhizobium sahei sp.nov., and Sinorhizobium teranga sp.nov[J]. International Journal of Systematic Bacteriology, 1994, 44(4): 715-733. DOI:10.1099/00207713-44-4-715 (  0) 0) |
| [15] |
GE F, ZHOU L Y, WANG Y, et al. Hydrolysis of the Neonicotinoid Insecticide Thiacloprid by the N2-Fixing Bacterium Ensifer meliloti CGMCC 7333[J]. International Biodeterioration & Biodegradation, 2014, 93: 10-17. (  0) 0) |
| [16] |
DAI Y J, JI W W, CHEN T, et al. Metabolism of the Neonicotinoid Insecticides Acetamiprid and Thiacloprid by the Yeast Rhodotorula mucilaginosa Strain IM-2[J]. Journal of Agricultural and Food Chemistry, 2010, 58(4): 2419-2425. DOI:10.1021/jf903787s (  0) 0) |
| [17] |
SHARMA M, SHARMA N N, BHALLA T C. Amidases:Versatile Enzymes in Nature[J]. Reviews in Environmental Science and Bio/Technology, 2009, 8(4): 343-366. DOI:10.1007/s11157-009-9175-x (  0) 0) |
| [18] |
SOROKIN D Y, PELT S V, TOUROVA T P, et al. Acetonitrile Degradation Under Haloalkaline Conditions by Natronocella acetinitrilica gen.nov., sp.nov.[J]. Microbiology, 2007, 153(4): 1157-1164. DOI:10.1099/mic.0.2006/004150-0 (  0) 0) |
| [19] |
MUKRAM I, RAMESH M, MONISHA T R, et al. Biodegradation of Butyronitrile and Demonstration of Its Mineralization by Rhodococcus sp.MTB5[J]. Biotechnology, 2016, 6(2): 141. (  0) 0) |
| [20] |
CANTARELLA M, CANTARELLA L, GALLIFUOCO A, et al. Nicotinic Acid Bio-Production by Microbacterium imperiale CBS 489-74:Effect of 3-Cyanopyridine and Temperature on Amidase Activity[J]. Process Biochemistry, 2012, 47(7): 1192-1196. DOI:10.1016/j.procbio.2012.03.010 (  0) 0) |
| [21] |
CANTARELLA L, GALLIFUOCO A, MALANDRA A, et al. High-Yield Continuous Production of Nicotinic Acid via Nitrile Hydratase-Amidase Cascade Reactions Using Cascade CSMRs[J]. Enzyme and Microbial Technology, 2011, 48(4/5): 345-350. (  0) 0) |
| [22] |
MUKRAM I, NAYAK A S, KIRANKUMAR B, et al. Isolation and Identification of a Nitrile Hydrolyzing Bacterium and Simultaneous Utilization of Aromatic and Aliphatic Nitriles[J]. International Biodeterioration & Biodegradation, 2015, 100: 165-171. (  0) 0) |
| [23] |
BRANDÃO P F B, BULL A T. Nitrile Hydrolysing Activities of Deep-Sea and Terrestrial Mycolate Actinomycetes[J]. Antonie Van Leeuwenhoek, 2003, 84(2): 89-98. DOI:10.1023/A:1025409818275 (  0) 0) |
| [24] |
KATAOKA S, ARAKAWA T, HORI S, et al. Functional Expression of Thiocyanate Hydrolase is Promoted by Its Activator Protein, P15K[J]. FEBS Letters, 2006, 580(19): 4667-4672. DOI:10.1016/j.febslet.2006.07.051 (  0) 0) |
| [25] |
NAWAZ M S, HEINZE T M, CERNIGLIA C E. Metabolism of Benzonitrile and Butyronitrile by Klebsiella pneumoniae[J]. Applied and Environmental Microbiology, 1992, 58(1): 27-31. (  0) 0) |
| [26] |
CANTARELLA M, CANTARELLA L, SPERA A, et al. Use of a UF-Membrane Reactor for Controlling Selectively the Nitrile Hydratase-Amidase System in Microbacterium imperiale CBS 498-74 Resting Cells Case Study:Benzonitrile Conversion[J]. Enzyme and Microbial Technology, 2006, 38(1): 126-134. (  0) 0) |




