噬藻体(Cyanophage)是一类能够特异性感染蓝藻的双链DNA病毒类群,广泛存在于海洋、淡水等自然环境中,是水生微生物群落的重要组成部分,能在不同时间和空间范围内感染蓝藻,具有丰富的遗传多样性[1]。作为水生态系统中的重要活跃因子,噬藻体与宿主蓝藻的新陈代谢和生命循环密切相关,具有重要的生态地位;在调控蓝藻种群结构及多样性、水体初级生产力、生物地球化学循环以及介导微生物之间水平基因转移等方面起到了重要的作用[2-4]。
噬藻体是高度差异性的病毒类群,尚未发现其基因中存在类似原核生物16SrDNA或真核生物18SrDNA的共有保守序列,因而缺乏广泛通用的遗传标记基因。基于噬藻体的分离纯化及全基因组测序研究发现部分种的噬藻体中存在个别共有保守基因,如 g20 基因[5-6]、psbA光合基因[7]和pol基因[8]等。g20 基因编码肌尾噬藻体衣壳组装蛋白主要存在于肌尾科噬藻体中。pol基因编码DNA聚合酶主要存在于短尾科噬藻体。psbA基因是编码光合系统Ⅱ反应中心D1蛋白的基因,在肌尾科和短尾科噬藻体均有发现[9]。许多噬藻体中都包含编码光合系统Ⅱ中D1蛋白的psbA基因,88%的感染海洋聚球藻和原绿球藻的噬藻体都含有psbA基因[10],全球海洋表层水中60%的psbA基因来源于病毒[11]。SULLIVAN等[12]根据光合基因的保守序列设计引物,对噬藻体的遗传多样性进行了研究,发现噬藻体的核酸序列具有相当丰富的多样性。作为噬藻体的一种新型靶标基因,光合作用基因psbA不仅可用于揭示噬藻体的遗传多样性,还可利用它探索噬藻体与其宿主蓝藻之间的关系[2]。
目前,国内对噬藻体遗传多样性的研究主要集中在中国近海海域[13]、东北稻田水体[14]、武汉东湖[15]等,有关淡水湖泊噬藻体多样性的相关报道仍十分有限。该研究主要是运用以PCR技术为核心的分子生物学方法对云南高原富营养化湖泊异龙湖春季和秋季水样噬藻体psbA基因的多样性进行研究,分析异龙湖水样中噬藻体的遗传多样性,为噬藻体基因多样性研究提供依据,并丰富富营养化湖泊微型生态系统的理论体系。
1 材料与方法 1.1 水样的采集及预处理异龙湖水样采集于2015年4月(春季)与2015年9月(秋季)。参照HJ 495—2009《水质采样方案设计技术规定》,使用采水器采集表层水样(距水体表面约0.5 m处)和底层水样(距水体表面约2 m处)各1.5 L,现场测定水温、pH值和透明度。采集水样后立即带回实验室,将水样混合后经0.75 μm孔径滤纸、0.45和0.22 μm孔径微孔滤膜〔上海市新亚净化器件厂微孔滤膜(沪Q/YY8-1-88)〕过滤,将过滤后的水样用6万分子量的中空纤维膜组件进行超浓缩,浓缩比例为200:1,浓缩液分装后于-20 ℃冰箱避光保存待用。
1.2 水样中噬藻体基因组提取及PCR扩增 1.2.1 水样中游离蓝藻DNA的去除为了去除浓缩水样中游离的蓝藻DNA片段,采用DNaseI处理浓缩水样,并进一步用蓝藻16S-23SrDNA ITS基因[16-18]进行PCR扩增,若为阴性结果,则去除完全,阳性则要使用DNaseI进行再处理。
ITS基因由引物[19]ITS-F(5′-TGTACACACCGCCCGTCACACCA-3′)和ITS-R(5′-GTGGATAC-CTAGGCACACAGAG-3′)进行PCR扩增。PCR扩增体系:2×Premix Taq (TAKARA)10 μL,F-Primer(10 μM) 1 μL,R-Primer (10 μM) 1 μL,模板DNA 2 μL,ddH2O 6 μL,总体积20 μL。阴性对照无菌ddH2O为模板。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s;55 ℃退火30 s;72 ℃延伸90 s;35个循环;72 ℃延伸7 min。
1.2.2 水样中病毒基因组提取及psbA基因的PCR扩增取经处理的水样,使用病毒基因组提取试剂盒Viral DNA Kit(OMEGA)提取噬藻体DNA。从GenBank中下载噬藻体psbA基因序列进行BALST比对,采用Primer 5.0和Oligo 6分析评价,针对psbA基因设计巢式引物:psbA-F1(5′-GARTGGYTNTAYAAYGGNG-3′)和psbA-R1(5′-CCRTTNAGGTTRAANGCCA-3′),psbA-F2(5′-CAYTTCTAYCCNATYTGG-3′)和psbA-R2(5′-TCYN GACTGGTTGAAGTTGA-3′)进行PCR扩增。PCR扩增体系:2×Premix Taq (TaKaRa) 10 μL,F-Primer(10 μM) 1 μL,R-Primer (10 μM) 1 μL,模板DNA 2 μL,ddH2O 6 μL,总体积20 μL。阴性对照无菌ddH2O为模板。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s;退火30 s(psbA 1 47 ℃,psbA2 50 ℃);72 ℃延伸90 s;35个循环;72 ℃延伸7 min。
1.3 胶回收、TA克隆及序列测定从w=2.0%的凝胶上切取580 bp左右的PCR产物,用普通琼脂糖凝胶DNA回收试剂盒(TIANGEN)进行胶纯化,目的DNA片段连接至pEASY-T1 Cloning Vector,转化大肠杆菌JM109感受态细胞。无菌条件下挑取Amp+的LB平板上50个样品克隆,再用通用引物M13F、M13R对菌液进行阳性重组子的菌落PCR快速鉴定,退火温度55 ℃,循环数30。选阳性克隆的培养过夜菌液,送至测序公司(昆明硕擎生物科技)进行一代测序。
1.4 psbA基因的系统进化分析待测序完成后,对异龙湖水域水样中psbA环境序列的单克隆序列分别用Chromas、BioEdit(Ver 7.0.0)进行序列校正、编辑。使用BLAST将得到的序列与数据库中收录的噬藻体psbA基因进行初步的比对分型,用MEGA 6.0软件绘制系统进化树,根据系统进化树来分析异龙湖中噬藻体的遗传多样性。噬藻体序列标注为“YL年月S+第几条序列”。该研究获得的psbA序列已提交至NCBI网站,其序列号为MH822090-MH822104。
2 结果与分析 2.1 异龙湖水样噬藻体psbA基因的PCR扩增及克隆测序将采集的异龙湖水样经0.75 μm孔径滤纸,0.45和0.22 μm微孔滤膜过滤和超浓缩后,DNase处理去除浓缩水样中蓝藻DNA,并用蓝藻16S-23SrDNA ITS基因进行PCR扩增,从结果为阴性的样品中提取噬藻体DNA;PCR扩增psbA基因于580 bp位置有明显亮带(图 1),经胶回收获取目的基因,样品经克隆筛选,将阳性克隆送测序公司测序,共获得15条不同的psbA基因,其中4月(春季)水样中获得3条,9月(秋季)水样中获得12条psbA环境序列。
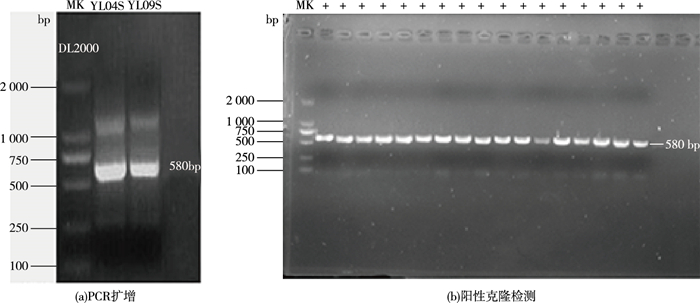
|
图 1 异龙湖水样中噬藻体psbA基因的PCR扩增及阳性克隆检测 Fig. 1 PCR amplification and positive clone of psbA gene from cyanophage in Yilong Lake |
异龙湖水样中扩增出的psbA基因长度为580 bp(引物除外),可翻译成186个氨基酸残基。对获得的psbA基因序列进行BLAST比对后,剔除部分非噬藻体源的psbA基因序列。该研究从异龙湖水样中获得15条psbA基因环境序列,经NCBI网站上基于氨基酸水平进行BLAST比对发现(表 1):有4条psbA基因序列(YL04S-1~3和YL09S-6)与哈萨克斯坦淡水中噬藻体psbA序列(KZ5-52073/3)相似性最高,相似率为99%;有10条psbA基因序列(YL09S-1~5,YL09S-7~8,YL09S-10~12)与中国东北稻田水体克隆(PW-DA-II-21,PW-MDJ-II-4,PW-DA-II-35)具有最高的相似性,相似率在98%~99%之间;仅有1条psbA基因序列(YL09S-9)与日本稻田克隆(NoF6)具有最高的相似性,相似率为98%。
|
|
表 1 异龙湖水体psbA基因与NCBI数据库在氨基酸水平的同源性比较 Table 1 Comparison of homology at amino acid level between psbA gene and NCBI database in Yilong Lake |
psbA基因存在光合作用生物中,因此区分psbA基因是否为病毒来源至关重要。SHARON等[20]通过统计分析发现,psbA基因编码D1蛋白的氨基酸序列模型R/KETTXXXSQ/H中可变的三肽元件XXX的变化可以反映出不同噬藻体所处的分类地位,其中EQE、ENE、EEV、EEE、EDV、EVE、EQV和EDE型等几种三肽元件更易来自于病毒D1亚基,而在宿主蓝藻中极少发现。SULLIVAN等[12]发现,聚球藻噬藻体的基因组序列中鸟嘌呤和胞嘧啶所占的比例(GC含量)比聚球藻的GC含量少10%左右,即噬藻体GC含量低于宿主藻的GC含量。
从异龙湖浓缩水样中获得的15条psbA基因环境系列,构建进化树(图 2),可将psbA基因序列划分成3簇,分别命名为ClusterⅠ~Cluster Ⅲ,对psbA基因序列的GC含量和编码D1蛋白的氨基酸序列中三肽元件R/KETTXXXSQ/H的变化进行分析发现,ClusterⅠ中GC含量为47.9%~50.0%,序列中三肽元件全部为EEV型,ClusterⅠ中包括的10条序列为病毒来源,即来源于噬藻体;Cluster Ⅱ中只含1条序列,GC含量为48.8%,序列中三肽元件为ETE型;Cluster Ⅲ中GC含量为44.5%~44.6%,4条序列中三肽元件全部为ETE型。研究发现,在聚球藻菌株及感染聚球藻的肌尾和短尾科噬藻体中均存在三肽元件ETE型[8],结合水样处理方法、GC含量及序列的系统进化位置及氨基酸同源性比较,推测ClusterⅡ和Cluster Ⅲ的psbA基因来自于噬藻体。因此,该研究获得的15条psbA基因序列均来自于噬藻体。
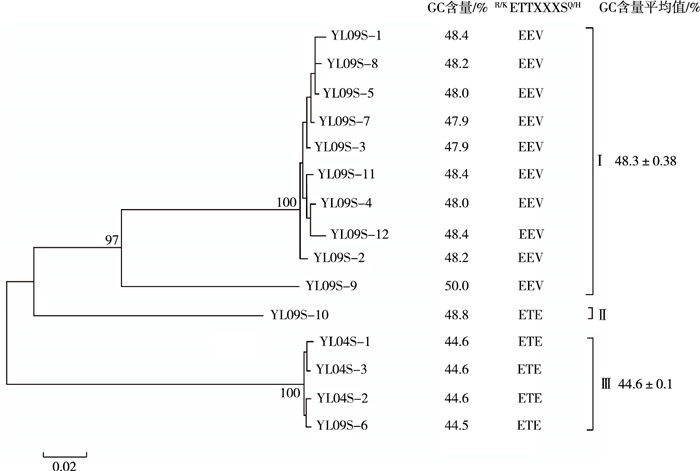
|
图 2 异龙湖噬藻体psbA序列进化关系、GC含量及D1蛋白氨基酸三肽元件模型 Fig. 2 Phylogenetic relationship, GC content and the amino acid triplet sequences from D1 protein motifs of cyanophage psbA gene in Yilong Lake |
将从异龙湖水样中获得的噬藻体psbA序列与具有代表性的蓝藻、绿藻、噬藻体分离株及噬藻体psbA环境序列等参考序列在核苷酸水平构建系统进化树。如图 3所示,所有的序列在进化树结构上可将其划分为10个进化簇(Cluster A~Cluster J)。该研究获得的15条异龙湖噬藻体psbA序列全部分布在Cluster A和C中。其中,Cluster A包括来自中国东北稻田[21]和日本稻田水体psbA序列[22]和该研究得到的11条异龙湖序列;Cluster B主要来自海洋[23]的噬藻体分离株;Cluster C单独成簇,包括该研究得到的4条异龙湖序列;Cluster D主要来自于北冰洋[24]和海洋原绿球藻(Prochlorococcus)及其噬藻体类群;Cluster E和F主要来自于日本稻田水体及奥杜藻属(Odontella rhomboides)的噬藻体psbA序列类群[25];Cluster G主要来自于淡水伊利湖的噬藻体类群[26];Cluster H、I和J包括来自蓝藻[27]、淡水湖[28]、原绿球藻、海洋聚球藻(Synechococcus)、日本稻田水体及其噬藻体分离株的psbA序列。
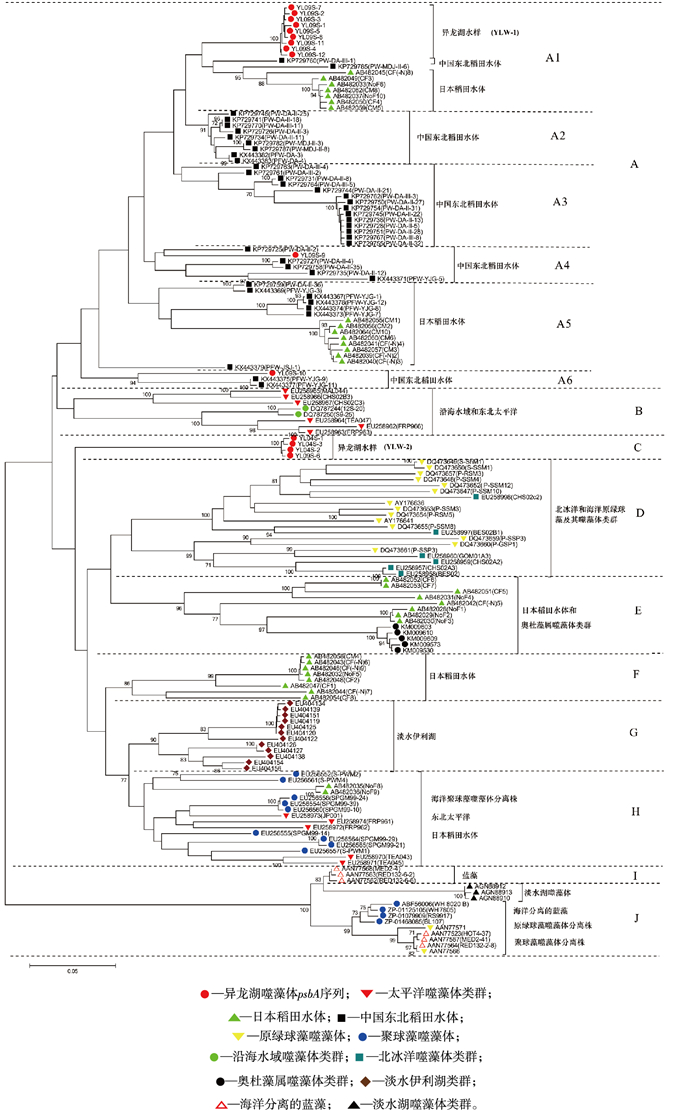
|
图 3 异龙湖与稻田生态系统及其他环境psbA序列构建的系统进化树 Fig. 3 Neighbor-joining phylogenetic tree showing the relationship of psbA amino acid sequence from Yilong Lake, paddy floodwater ecosystems and other environments |
Cluster A的克隆来自稻田生态系统和该研究的11条序列,形成6个进化亚簇,分别为来自中国东北稻田水体中的psbA基因序列形成的4个进化亚簇A2、A3、A4和A6;1个来自日本稻田水体的psbA基因序列形成的进化亚簇A5;其中该研究获得的9条异龙湖psbA基因序列形成一个进化小亚簇(YLW-1),与中国东北稻田水体亚簇的psbA序列进化距离相近,和日本稻田水体共同形成进化亚簇A1;1条异龙湖序列YL09S-9与中国东北稻田水体psbA基因序列同处于A4亚簇;还有1条异龙湖序列YL09S-10与中国东北稻田水体psbA基因序列形成的A6进化亚簇处于同一进化亚簇。Cluster C包括该研究得到的4条异龙湖序列(YL04S-1、YL04S-2、YL04S-3和YL09S-6),形成一个相对独立的进化簇YLW-2,与Cluster D~J在进化距离上相隔较远,推测为新的噬藻体psbA基因类群。
结合图 2可知,Cluster A中的异龙湖psbA基因的氨基酸序列可变的三肽元件除序列YL09S-10之外均为EEV型;且YL09S-9与其他序列进化距离较远,所以其余9条序列聚在一簇。在Cluster C中,4条异龙湖序列氨基酸序列可变的三肽元件均为ETE型,并且单独成簇;与Cluster D~H中所包含的海洋、稻田等psbA序列的进化距离相隔较远。云南高原湖泊异龙湖水样中psbA基因序列和Cluster G中其他淡水湖泊(北美洲伊利湖)中的psbA基因序列在系统进化树上的进化距离表明,虽都是淡水湖,但两者进化历史相互独立,仅存在有限的基因交流。此外,大部分异龙湖psbA克隆都在Cluster A中,且与中国东北稻田的psbA序列相隔较近,这表明异龙湖噬藻体psbA基因的分布狭窄,远离其他淡水湖泊和海洋的分布,与海洋、国外淡水湖和日本稻田水体不同,与中国东北稻田的生态类群进化距离较近,进化相对独立。从异龙湖不同季节春季和秋季采集水样中分别获得的3条和12条psbA基因序列,秋季水样中psbA序列分布于Cluster A和Cluster C这2个进化簇中,春季的3条psbA基因序列分布于Cluster C中,显示秋季噬藻体psbA基因序列的遗传多样性更为丰富。
3 讨论与结论在该研究中,采用光合作用基因psbA作为靶标基因,光合作用基因在噬藻体内广泛存在且十分保守。此前的研究中曾用到g20基因,但是相对于psbA基因来说,g20基因只局限存在于肌尾病毒科噬藻体,对于短尾病毒科的噬藻体,g20无法检测出来,因此,使用g20作为研究噬藻体遗传多样性的分子标记有一定的局限性。CHENARD[24]在研究淡水和海洋中噬藻体的多样性时使用psbA基因作为分子标记的靶标基因;该研究主要参照上述文献的实验技术方法,选用psbA基因作为研究异龙湖噬藻体多样性的分子标记基因,结果证明psbA基因能很好地揭示噬藻体的遗传多样性。
为区分环境样品中获得的psbA基因是噬藻体来源还是宿主来源,目前国内外相关研究中JING等[11]、SHARON等[20]均采用了CHENARD等[24]建议的方法:(1)将采集的水样先经滤纸过滤,再分别经0.45和0.22 μm孔径的滤膜过滤以除去藻类宿主等细胞型生物,滤液通过超滤浓缩,对浓缩液进行psbA基因的PCR扩增前,先用DNase处理以除去浓缩水样中可能含有的游离蓝藻DNA,用蓝藻ITS基因序列扩增结果为阴性后,再提取浓缩水样中的DNA;(2)基于噬藻体和宿主的psbA基因序列的GC含量分析表明,噬藻体的GC含量低于其宿主的GC含量;(3)基于编码D1蛋白的氨基酸序列模型R/KETTXXXSQ/H中三肽元件XXX变化的区分方法表明, 该方法从采集水样的处理、已有噬藻体和宿主的psbA基因序列的GC含量以及基于编码D1蛋白的氨基酸序列模型R/KETTXXXSQ/H中三肽元件XXX变化的统计分析后提出,可以区分出环境样品中获得的psbA基因来源。
在对高原湖泊异龙湖中噬藻体psbA基因多样性进行研究时发现,与淡水、海洋环境比较,异龙湖噬藻体序列与来自中国东北稻田水体中噬藻体psbA基因群集更为相似,与其他淡水湖泊、海洋噬藻体psbA基因进化距离较远;不同环境水域中噬藻体含有其独特的类型,显示不同自然环境中噬藻体psbA基因组成存在一定差异,psbA基因的分布与其存在的环境有很大的关系。ZHONG等[29]对法国的安纳西湖和布尔热湖的噬藻体多样性进行分析,发现这2个湖泊中的噬藻体psbA存在着一定的差异。
CHENARD等[24]对海洋和淡水中不同地点的噬藻体psbA基因进行扩增,并通过构建噬藻体psbA基因的系统进化树发现,海洋中的克隆划分为7个类群,淡水中的克隆则为2个来自不同湖泊的独立类群,表明海洋和淡水中的噬藻体存在着较大的差异。WANG等[22]对日本稻田水中的噬藻体多样性进行了研究,利用PCR扩增得到37条不同的噬藻体psbA序列,发现噬藻体的遗传多样性与其宿主有关,其群落结构的丰富度与蓝藻多样性呈正比。荆瑞勇等[8]在对中国东北稻田水体的研究中获得17条噬藻体psbA序列,并发现其中存在新的psbA基因类群。该研究对富营养化湖泊异龙湖水体的研究得到的结果与上述结果相似,并发现其中也存在新的psbA基因类群;从2015年春季(4月)及秋季(9月)采集的水样中分别获得3条和12条psbA环境系列及其在进化树中的分布,显示异龙湖秋季噬藻体psbA基因遗传多样性更加丰富,表明异龙湖噬藻体psbA基因序列在不同季节的水样中存在差异;这可能与不同季节异龙湖的水温、pH值等环境因素的变化有关。水温是影响浮游藻类生长的重要因子,适宜的水温有利于浮游藻类的生长[30],即在一定的温度范围内,水温升高可促进藻类光合作用。异龙湖春季(4月)水温(20.2 ℃)较低,秋季(9月)水温为23.9 ℃,秋季水温较高对浮游藻类的生长更加有利,同时异龙湖秋季水样pH值(8.6)较春季水样(7.8)高,弱碱性的水体也适宜藻类生长,而噬藻体群落结构的丰富度与蓝藻的多样性呈正比,从而也影响了异龙湖噬藻体种类的不同,显示异龙湖噬藻体随季节、温度等环境因素的变化而导致的主要基因型的差异,表明异龙湖噬藻体基因遗传多样性与环境水质参数有一定的关系。
综上所述,以psbA基因为靶标基因,对高原湖泊异龙湖中噬藻体类群的分析研究表明,异龙湖水体中噬藻体psbA基因类群与海洋、其他淡水湖和日本稻田水体psbA基因类群不同,与中国东北稻田的生态类群进化距离较近,进化相对独立,存在新的噬藻体psbA基因类群,并且在不同季节的水样中噬藻体psbA基因序列存在差异,秋季其遗传多样性更加丰富。
| [1] |
刘腾腾, 刘丽, 魏大巧, 等. 噬藻体和蓝藻间的基因转移及协同进化作用[J]. 生物技术通报, 2011(7): 12-17. [ LIU Teng-teng, LIU Li, WEI Da-qiao, et al. The Role of Horizontal Gene Transfer and Co-Evolution Mechanism Between Cyanophage and Cyanobacteria[J]. Biotechnology Bulletin, 2011(7): 12-17.] (  0) 0) |
| [2] |
张奇亚. 噬藻体生物多样性的研究动态[J]. 微生物学通报, 2014, 41(3): 545-559. [ ZHANG Qi-ya. Advances in Studies on Biodiversity of Cyanophages[J]. Microbiology China, 2014, 41(3): 545-559.] (  0) 0) |
| [3] |
WEI D, FU C, RAYTCHEVA D, et al. Visualizing Virus Assembly Intermediates Inside Marine Cyanobacteria[J]. Nature, 2013, 502(7473): 707-710. DOI:10.1038/nature12604 (  0) 0) |
| [4] |
OU T, GAO X C, LI S H, et al. Genome Analysisand Gene nblA Identification of Microcystis aeruginosa Myovirus (MaMV-DC) Reveal the Evidence for HorizontalGene Transfer Events Between Cyanomyovirus and Host[J]. Journal of General Virology, 2015, 96(12): 3681. DOI:10.1099/jgv.0.000290 (  0) 0) |
| [5] |
荆瑞勇, KIMURA M, 王光华. 不同自然环境下噬藻体 g20 基因多样性研究进展[J]. 微生物学报, 2013, 53(11): 1149-1157. [ JING Rui-yong, KIMURA M, WANG Guang-hua. Genetic Diversity of Capsid Assembly Protein Genes ( g20 ) of Cyanophage in Different Natural Environment:A Review[J]. Acta Microbiologica Sinica, 2013, 53(11): 1149-1157.] (  0) 0) |
| [6] |
BUTINA T V, POTAPOV S A, BELYKH O I, et al. Genetic Diversity of Cyanophages of the Myoviridae Family as a Constituent of the Associated Community of the Baikal Sponge Lubomirskia baicalensis[J]. Russian Journal of Genetics, 2015, 51(3): 313-317. DOI:10.1134/S1022795415030011 (  0) 0) |
| [7] |
XU Y, ZHANG R, WANG N, et al. Novel Phage-host Interactions and Evolution as Revealed by a Cyanomyovirus Isolated From an Estuarine Environment[J]. Environmental Microbiology, 2018, 20(8): 2974-2989. DOI:10.1111/1462-2920.14326 (  0) 0) |
| [8] |
CHEN F, WANG K, HUANG S J, et al. Diverse and Dynamic Populations of Cyanobacterial Podoviruses in the Chesapeake Bay Unveiled Through DNA Polymerase Gene Sequences[J]. Environmental Microbiology, 2009, 11(11): 2884-2892. DOI:10.1111/j.1462-2920.2009.02033.x (  0) 0) |
| [9] |
汪岷, 闫群. 噬藻体遗传多样性的研究进展[J]. 中国海洋大学学报, 2010, 40(8): 73-79. [ WANG Min, YAN Qun. Advances in Research on Genetic Diversity of Cyanophage[J]. Periodical of Ocean University of China, 2010, 40(8): 73-79.] (  0) 0) |
| [10] |
李樾, 刘婷婷, 刘丽, 等. 基于psbA基因的噬藻体遗传多样性研究进展[J]. 生态与农村环境学报, 2016, 32(3): 417-423. [ LI Yue, LIU Ting-ting, LIU Li, et al. Progress of Research on Genetic Diversity of Cyanophage Based on psbA Gene[J]. Journal of Ecology and Rural Environment, 2016, 32(3): 417-423.] (  0) 0) |
| [11] |
荆瑞勇, 曹焜, 刘俊杰, 等. 东北稻田水体噬藻体psbA基因多样性[J]. 微生物学报, 2017, 57(1): 131-139. [ JING Rui-yong, CAO Kun, LIU Jun-jie, et al. Genetic Diversity of psbA of Cyanophage from Paddy Floodwater in Northeast China[J]. Acta Microbiologica Sinica, 2017, 57(1): 131-139.] (  0) 0) |
| [12] |
SULLIVAN M B, LINDELL D, LEE J A, et al. Prevalence and Evolution of Core Photosystem Ⅱ Genes in Marine Cyanobacterial Viruses and Their Hosts[J]. PLoS Biology, 2006, 4(8): e234. DOI:10.1371/journal.pbio.0040234 (  0) 0) |
| [13] |
闫群.青岛近海海域噬藻体g20基因多样性的研究[D].青岛: 中国海洋大学, 2010. [YAN Qun. g20 Gene Diversity of Cyanophage Community in the Yellow Sea Coastal Area Around Qingdao[D]. Qingdao: Ocean University of China, 2010.] http://cdmd.cnki.com.cn/article/cdmd-10423-1011027809.htm
(  0) 0) |
| [14] |
JING R Y, LIU J J, YU Z H, et al. Phylogenetic Distribution of the Capsid Assembly Protein Gene ( g20) of Cyanophage in Paddy Floodwaters in Northeast China[J]. PLoS One, 2014, 9(2): e88634. DOI:10.1371/journal.pone.0088634 (  0) 0) |
| [15] |
WANG M N, GE X Y, WU Y Q, et al. Genetic Diversity and Temporal Dynamics of Phytoplankton Viruses in East Lake, China[J]. Virologica Sinica, 2015, 30(4): 290-300. DOI:10.1007/s12250-015-3603-6 (  0) 0) |
| [16] |
GARCIA M J, ACINAS S G, ANTON A I, et al. Use of the 16S-23S Ribosomal Genes Spacer Region in Studies of Prokaryotic Diversity[J]. Journal of Microbiological Methods, 1999, 36(1/2): 55-64. (  0) 0) |
| [17] |
FERRIS M J, KUHL M, WIELAND A, et al. Cyanobacterial Ecotypes in Different Optical Microenvironments of a 68Degrees C Hot Spring Mat Community Revealed by 16S-23S rRNA Internal Transcribed Spacer Region Variation[J]. Applied and Environmental Microbiology, 2003, 69(5): 2893-2898. DOI:10.1128/AEM.69.5.2893-2898.2003 (  0) 0) |
| [18] |
ROCAP G, DISTEL D L, WATERBURY J B, et al. Resolution of Prochlorococcus and Synechococcus Ecotypes by Using 16S-23S Ribosomal DNA Internal Transcribed Spacer Sequences[J]. Applied and Environmental Microbiology, 2002, 68(3): 1180-1191. DOI:10.1128/AEM.68.3.1180-1191.2002 (  0) 0) |
| [19] |
彭玉辅.滇池水域微藻种群变化、食藻原生动物和溶藻细菌分离纯化及初步研究[D].昆明: 昆明理工大学, 2012. [PENG Yu-fu.Changes of Phyloplankton Population of Lake Dianchi, Isolation and Preliminary Study on Protozoan and Algicidal Bacteria[D]. Kunming: Kunming University of Science and Technology, 2012.] http://cdmd.cnki.com.cn/Article/CDMD-10674-1012430959.htm
(  0) 0) |
| [20] |
SHARON I, TZAHOR S, WILLIAMSON S, et al. Viral Photosynthetic Reaction Center Genes and Transcripts in the Marine Environment[J]. Multidisciplinary Journal of Microbial Ecology, 2007, 1(6): 492-501. (  0) 0) |
| [21] |
WANG X Z, JING R Y, LIU J J, et al. Narrow Distribution of CyanophagepsbA Genes Observed in Two Paddy Waters of Northeast China by an Incubation Experiment[J]. Virologica Sinica, 2016, 31(2): 188-191. DOI:10.1007/s12250-015-3673-5 (  0) 0) |
| [22] |
WANG G H, MURASE J, ASAKAWA S, et al. Novel Cyanophage Photosynthetic Gene psbA in the Floodwater of a Japanese Rice Field[J]. FEMS Microbiology Ecology, 2009, 70(1): 79-86. DOI:10.1111/j.1574-6941.2009.00743.x (  0) 0) |
| [23] |
SANDAA R A, CLOKIE M, MANN N H. Photosynthetic Genes in Viral Populations With a Large Genomic Size Range From Norwegian Coastal Waters[J]. FEMS Microbiology Ecology, 2008, 63(1): 2-11. DOI:10.1111/j.1574-6941.2007.00400.x (  0) 0) |
| [24] |
CHENARD C, SUTTLE CA. Phylogenetic Diversity of Sequences of Cyanophage Photosynthetic Gene psbA in Marine and Freshwaters[J]. Applied and Environmental Microbiology, 2008, 74(17): 5317-5324. DOI:10.1128/AEM.02480-07 (  0) 0) |
| [25] |
THERIOT E C, ASHWORTH M P, NAKOV T, et al. Dissecting Signal and Noise in Diatom Chloroplast Protein Encoding Genes With Phylogenetic Information Profiling[J]. Molecular Phylogenetics and Evolution, 2015, 89: 28-36. DOI:10.1016/j.ympev.2015.03.012 (  0) 0) |
| [26] |
WILHELM S W, MATTESON A R. Freshwater and Marine Virioplankton:a Brief Overview of Commonalities and Differences[J]. Freshwater Biology, 2008, 53(6): 1076-1089. DOI:10.1111/j.1365-2427.2008.01980.x (  0) 0) |
| [27] |
ZEIDENER G, PRESTON C M, DELONG E F, et al. Molecular Diversity Among Marine Picophytoplankton as Revealed by psbA Analyses[J]. Environmental Microbiology, 2010, 5(3): 212-216. (  0) 0) |
| [28] |
ZHONG X, JACQUET S. Prevalence of Viral Photosynthetic and Capsid Protein Genes From Cyanophages in Two Large and Deep Perialpine Lakes[J]. Applied and Environmental Microbiology, 2013, 79(23): 7169-7178. DOI:10.1128/AEM.01914-13 (  0) 0) |
| [29] |
ZHONG Y, CHEN F, WILHELM S W, et al. Phylogenetic Diversity of Marine Cyanophage Isolates and Natural Virus Communities as Revealed by Sequences of Viral Capsid Assembly Protein Gene g20[J]. Applied and Environmental Microbiology, 2002, 68(4): 1576-1584. DOI:10.1128/AEM.68.4.1576-1584.2002 (  0) 0) |
| [30] |
吴阿娜, 朱梦杰, 汤琳, 等. 淀山湖蓝藻水华高发期叶绿素a动态及相关环境因子分析[J]. 湖泊科学, 2011, 23(1): 67-72. [ WU A-na, ZHU Meng-jie, TANG Lin, et al. Dynamics of Chlorophyll-a and Analysis of Environmental Factors in Lake Dianshan During Summer Autumn[J]. Journal of Lake Sciences, 2011, 23(1): 67-72.] (  0) 0) |




