2. 宁夏大学生命科学学院, 宁夏 银川 750021
2. College of Life Science, Ningxia University, Yinchuan 750021, China
宁夏回族自治区地处我国西北内陆农牧交错区,是农业部“全国奶牛优势区域布局规划”的重点区域[1]。截至2016年底,宁夏奶牛存栏量已达57.6万头,每年奶牛养殖业排放的粪污高达350万t[2],大量粪污堆积已经成为威胁健康养殖和公共卫生安全的重大隐患之一。已有研究表明,奶牛粪便中的有机物、重金属元素可溶于废水或雨水,并随之渗入土壤污染地下水[3];同时奶牛粪便会产生大量含甲烷、氨、硫化物等有害物质的气体,污染周边空气,对人和奶牛的呼吸道造成危害[4];更严重的是奶牛粪便携带的多种寄生虫和病原菌会对人和奶牛的健康造成威胁。据世界卫生组织报道,由动物传染给人的疾病有90余种[5],包括细菌感染所导致的疾病[6-7]。SINTON等[8]发现一些食品中病原菌的污染与畜禽排泄物有关,这些病原菌通过粪便还田施入土壤,可在环境中长时间存活,并通过种植的作物最终影响到人类健康[9]。如果对养殖场粪污不采取有效处理措施,粪污将会对水、土、气等造成严重生物性污染,对人类和生态环境造成极大危害[10]。因此,深入研究奶牛养殖场粪便可培养细菌种类及多样性可为奶牛粪便无害化处理和资源化利用提供重要基础数据。
宁夏回族自治区目前对奶牛粪便的利用途径主要有两种:一是作为有机肥料;二是晾干后返回养殖场做为场区垫料。这两种处理方式都没有经过相应的无害化处理,且由于缺少对研究区奶牛粪便中细菌种群分布及耐药性的相关研究,导致无法对这两种粪便利用方式进行有效、准确的风险评估。因此,采用梅里埃全自动微生物鉴定及药敏系统和16S rDNA全长测序方法对银川地区规模化奶牛养殖场粪污及周边土壤中可培养细菌种群分布及多样性进行系统研究,为奶牛养殖业对环境及公共卫生安全影响的风险评估提供有效数据。
1 材料与方法 1.1 样品采集样品采自宁夏回族自治区银川市3家规模化奶牛养殖场,包括108份新鲜粪便(牛体排出还带有体温的新鲜粪便)、81份堆积粪便(堆积3个月以上的粪便及垫料混合物)、68份土壤(养殖场周边未接触粪便的环境土壤)和18份污水(冲洗牛体及圈舍的排放水),共275份。样品采集之后装入自封袋中并编号,保存于冰盒,在4 h内送至实验室后立即将样品混匀、稀释并涂布接种。
1.2 主要试剂与仪器伊红美蓝琼脂培养基、麦康凯琼脂培养基、甘露醇氯化钠琼脂培养基、蜡样芽孢杆菌选择性琼脂培养基和SS琼脂培养基均购于青岛高科园海博生物技术有限公司,营养琼脂培养基、胰蛋白大豆琼脂培养基和TODD-HEWITT BROTH培养基均购于英国OXCID公司,血琼脂平板培养基购于北京陆桥生物科技有限公司,全自动微生物鉴定及药敏系统(VITEK© 2 Compact)、比浊仪(DensiCHEK plus)以及VITEK2革兰氏阴性(21341)、VITEK2革兰氏阳性(21342)和BCL芽孢细菌鉴定卡(21345)均来自法国生物梅里埃公司,高压灭菌锅来自德国AUTOCLAVE有限公司;生物光学显微镜购于北京普瑞赛司仪器有限公司。
1.3 样品悬液的制备将采集的新鲜粪便样品以至少6个为1组进行混样,其他3种样品(堆积粪便、土壤和污水)根据每个养殖场采样数量以至少3个为1组进行混样。混样时每份样品称取5.0 g,取混合后的样品5.0 g加入45 mL无菌生理盐水制成10-1梯度稀释液,置于漩涡振荡仪上振荡(800 r·min-1,1 min)混匀,取1 mL悬液与无菌生理盐水倍比稀释制成10-2和10-3梯度稀释液。
1.4 可培养细菌的分离及纯化取100 μL的10-3梯度稀释液,用无菌涂布棒均匀涂布于培养基上。涂布后将平板倒置于37 ℃条件下恒温培养箱中培养24 h,记录菌落特征及数量,并采用平板划线法,对分离到的菌株进行纯化,每株细菌纯化3~4代,得到纯分离株。
1.5 分离株的鉴定通过对不同分离培养基的初培养菌落形态进行鉴定[11],初步确定细菌种类(表 1)。采用平板划线法,挑取新鲜单个菌落接种于胰蛋白大豆琼脂培养基,37 ℃条件下恒温培养箱中培养24 h后,选取生长良好、性状稳定的单个菌落进行革兰染色并镜检(莱卡显微系统DM2500,100X oil)。对于镜检时形态一致、革兰染色结果明确的分离株使用相对应的检测卡并采用全自动微生物鉴定及药敏系统进行鉴定;对革兰染色镜检结果不明确和全自动微生物鉴定及药敏系统未鉴定出的菌株选取符合测序要求的进行16S rDNA全长测序,通过对测序结果的BLAST序列进行比对,获得其种属信息。16S rDNA全长测序、拼接由广州赛哲生物科技股份有限公司完成。
|
|
表 1 鉴别培养基用途及典型菌落特征 Table 1 Differential media use and typical colony characteristics |
通过对收集样品进行分离培养,共获得429株分离株,对其中383株镜检时形态一致、革兰染色结果明确的分离株使用相对应的检测卡进行鉴定,263株菌株可以确定种属,鉴定率为68.67%;120株无鉴定结果,占鉴定菌株的31.33%。对符合测序要求的102株菌株进行16S rDNA全长测序,鉴定出其中95株。两种方法共鉴定358株细菌分离株(表 2),表明银川地区规模化奶牛养殖场新鲜粪便、堆积粪便、污水和环境土壤样品中可培养细菌种类较丰富。
|
|
表 2 养殖场环境分离株鉴定情况 Table 2 Farms environmental isolates identified |
可培养细菌种群多样性分析结果表明,358株分离株分布于4个门、6个纲、15个目、23个科、39个属和102个种,4种样品在科以上水平上分布差异较小,而在属、种水平上4种样品的细菌种群分布差异较大(表 3)。
|
|
表 3 养殖场环境分离株种群分布多样性 Table 3 Diversity of isolates in dairy farms′ samples |
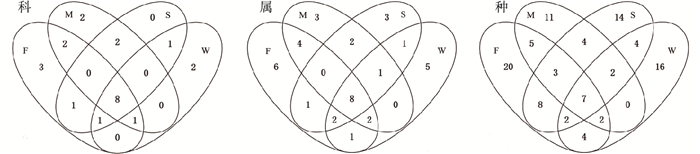
4种样品在不同水平上的Venn图见图 1。
|
F表示新鲜粪便样品;M表示堆积粪便样品;S表示土壤样品;W表示污水样品。 图 1 4种样品在不同分类水平上的Venn图 Fig. 1 The Venn diagram of different level of taxonomy in four samples |
由图 1可知,在科分类水平上,气球菌科(Aerococcaceae)和消化球菌科(Peptococcaceae)为新鲜粪便与堆积粪便样品的共有科,慢生根瘤菌科(Bradyrhizobiaceae)和黄杆菌科(Flavobacteriaceae)为堆积粪便与土壤样品的共有科;假单胞菌科(Pseudomonadaceae)为土壤与污水样品的共有科。在属分类水平上,气球菌属(Aerococcus spp.)、孪生菌属(Gemella spp.)、差异球菌属(Alloiococcus spp.)、变形杆菌属(Proteus spp.)出现在新鲜粪便与堆积粪便样品中;慢生根瘤菌属(Bradyrhizobium spp.)和东海菌属(Donghaeana spp.)为堆积粪便与土壤样品的共有属;假单胞菌属(Pseudomonas spp.)为土壤与污水样品的共有属。进一步在种分类水平上分析,嗜根考克菌(Kocuria rhizophila)、浅绿气球菌(Aerococcus viridans)、嗜艾氏杆菌(Bacillus eiseniae)、耳炎差异球菌(Alloiococcus otitis)和大肠埃希菌O157:H7(Escherichia coli O157:H7)分离自新鲜粪便和堆积粪便样品中;溶血葡萄球菌(Staphylococcus haemolyticus)、木糖葡萄球菌(Staphylococcus xylosus)、放射根瘤菌(Rhizobium radiobacter)、脑膜炎败血伊丽莎白菌(Elizabeth kingia meningosepticum)为堆积粪便与土壤样品的共有种;腐生葡萄球菌(Staphylococcus saprophyticus)、芽孢杆菌(Bacillus oceanisediminis)、施氏假单胞菌(Pseudomonas stutzeri)、荧光假单胞菌(Pseudomonas fluorescens)为土壤与污水样品的共有种;在新鲜粪便与土壤样品中,分离出海氏肠球菌(Enterococcus hirae)、苛养颗粒链菌(Granulicatella elegans)、人葡萄球菌(Staphylococcus hominis ssp. hominis)和表皮葡萄球菌(Staphylococcus epidermidis)等8种共有种。4种样品在各分类阶元均分离出同种属的分离株,表明样品中细菌种群分布具有相似性。
新鲜粪便与堆积粪便样品共有菌种数量在各分类阶元中均高于新鲜粪便与土壤样品或与污水样品,说明堆积粪便保留了大部分来自新鲜粪便的群落,同时还受土壤细菌种群的影响,使两者群落结构具有相似性;污水样品中可培养细菌种群分布与新鲜粪便、堆积粪便和土壤样品都存在较高的相似性,这主要是由于污水的流动性导致大量细菌富集所致。通过对4种样品细菌分离株在不同分类阶元中分布的差异分析,表明各样本具有丰富的细菌种群结构。由于受到人为或自然的影响,细菌会在环境中发生迁移,增加了不同环境样本的种群多样性,同时也增加了病原菌的传播风险。
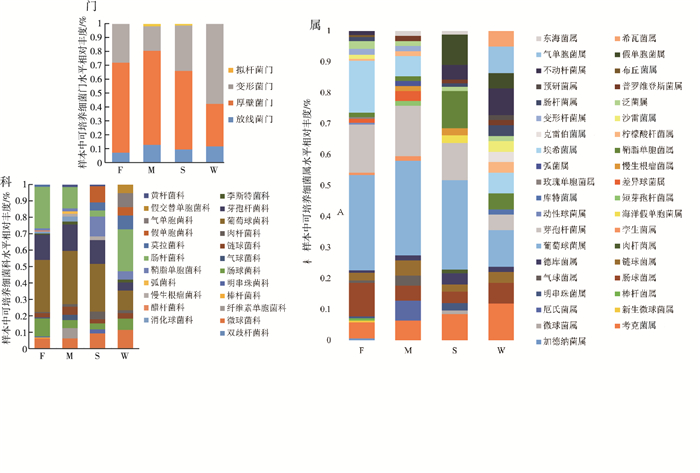
样品中可培养细菌组成和相对丰度见图 2。由图 2可知,厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是优势微生物门,覆盖了90%以上的菌种;微球菌科(Micrococcaceae)、肠球菌科(Enterococcaceae)、链球菌科(Streptococcaceae)、肉杆菌科(Carnobacteriaceae)、葡萄球菌科(Staphylococcaceae)、芽孢杆菌科(Bacillaceae)、鞘脂单胞菌科(Sphingomonadaceae)和肠杆菌科(Enterobacteriaceae)8个科是优势科,占总类群的84.40%;考克菌属(Kocuria spp.)、肠球菌属(Enterococcus spp.)、链球菌属(Streptococcus spp.)、德库菌属(Desemzia spp.)、葡萄球菌属(Staphylococcus spp.)、芽孢杆菌属(Bacillus spp.)、鞘脂单胞菌属(Sphingomonas spp.)和泛菌属(Pantoea spp.)为优势属,占总类群的66.30%。随分类阶元的降低,样本间共有菌属的相对丰度降低,而细菌群落多样性增加。
|
F表示新鲜粪便样品;M表示堆积粪便样品;S表示土壤样品;W表示污水样品。 图 2 奶牛养殖场可培养细菌在门、科和属水平上的组成和相对丰度 Fig. 2 Relative abundance of the bacterial phyla, family and genera in dairy farms communities |
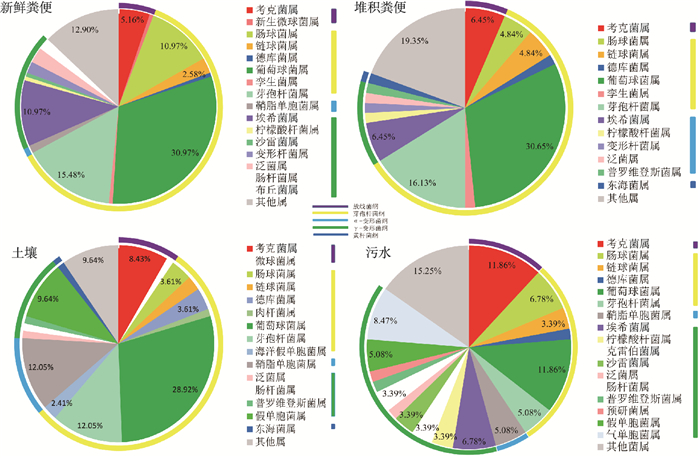
由图 3可知,不同样品优势细菌种群组成较一致,葡萄球菌属作为4种样品的共同优势菌属,分别占新鲜粪便、堆积粪便、土壤和污水样品中可培养细菌种类的30.97%、30.65%、28.92%和11.86%;其次是芽孢杆菌属,分别占15.48%、16.13%、12.05%和5.08%。但是由于样品自身的特性,群落组成也表现出明显差异。例如,在污水样品中,考克菌属所占比例和葡萄球菌属相同,为11.86%,但在其他3种样品中考克菌属所占比例远低于葡萄球菌属。部分细菌种群是各样品所独有的,例如新鲜粪便样品中独有加德纳菌属(Gardnerella spp.)0.65%、新生微球菌属(Newborn micrococcus spp.)0.65%、棒杆菌属(Corynebacterium spp.)0.65%、动性球菌属(Planococcus spp.)0.65%、玫瑰单胞菌属(Roseomonas spp.)0.65%和布丘菌属(Buttiauxella spp.)0.65%;堆积粪便样品中独有短芽孢杆菌属(Brevibacillus spp.)1.61%和弧菌属(Vibrio spp.)1.61%;土壤样品中独有微球菌属(Micrococcus spp.)1.20%、肉杆菌属(Carnobacterium spp.)1.20%和海洋假单胞菌属(Marine pseudomonas spp.)2.41%;污水样品中独有库特菌属(Kurthia spp.)1.69%、克雷伯菌属(Klebsiella spp.)3.39%、预研菌属(Yokenella spp.)1.69%、气单胞菌属(Aeromonas spp.)8.47%和希瓦菌属(Shewanella spp.)5.08%。这种差异不仅展示了各种样品的独特性,也说明粪便堆积过程会使一些细菌种群丰度增加,并且在堆积过程中,细菌会与周围环境发生迁移,增加细菌性疾病传播的风险。
|
图 3 样品中可培养微生物在纲、属水平的分布 Fig. 3 Relative abundance of the distribution of class and genus of culturable bacteria in 4 samples |
健康的人或动物体表及与外界相通的腔道等部位存在多种细菌种群,当机体免疫力处于正常状态时,这些菌群对机体无害甚至是有益的;但是当机体抵抗力降低时,这些细菌可能侵入机体其他部位从而引起感染,这类细菌称为条件致病菌(机会致病菌)[12]。基于《一二三类动物疫病释义》[13]和《动物病原微生物分类名录》(原农业部第53号令)对条件致病菌的定义及归类,对4种样品中可培养细菌分离株进行比对分析,部分结果见表 4。在358株分离株中,条件致病菌约占88.55%(317/358),在种水平上主要为[14-15]金黄色葡萄球菌(Staphylococcus aureus)和大肠埃希菌[16],在属水平上主要为链球菌属、肠球菌属、不动杆菌属(Acinetobacter spp.)、假单胞菌属(Pseudomonas spp.)和鞘脂单胞菌属等。在新鲜粪便样品中金黄色葡萄球菌占4.84%,大肠埃希菌占6.54%;在堆积粪便样品中金黄色葡萄球菌占2.58%,大肠埃希菌占16.77%,炭疽杆菌(Bacillus anthracis)占1.61%;在土壤样品中金黄色葡萄球菌占3.61%;在污水样品中大肠埃希菌占5.08%。这表明奶牛养殖场粪污中含有大量条件致病菌,这些条件致病菌将对人和动物的健康以及对环境造成严重威胁[17],是人类公共卫生安全的潜在风险源。
|
|
表 4 奶牛养殖场环境样品中部分条件致病菌种属信息 Table 4 Conditional pathogenic bacteria in environmental samples of dairy farms |
规模化养殖是目前奶牛养殖产业发展的主要方式之一,而养殖场中可培养细菌种群直接影响着奶牛养殖业能否健康有序发展。因此,养殖场中可培养细菌种群分布及多样性一直是广受关注的热点问题。目前有关奶牛肠道微生物研究较多[18],但是针对奶牛粪便及奶牛养殖场环境土壤及粪污中可培养细菌多样性的研究却鲜有报道。基于细菌种属间典型生理生化反应谱差异和16s rDNA序列多态性,采用全自动微生物鉴定及药敏系统和16S rRNA测序方法[19-22],共鉴定了358株分离株,分布于4个门、6个纲、15个目、23个科、39个属和102个种,表明银川地区规模化奶牛养殖场的粪污及土壤样品中可培养细菌种群丰富且多样,其中条件致病菌占88.55%(317/358),表明奶牛养殖场粪污是潜在的病原菌传播源[23]。
分析结果发现厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是奶牛粪便、污水和环境土壤样品中可培养细菌的优势菌门,占分离株总数的近90%,这与相关研究结果[24-25]一致。其中,堆积粪便样品的细菌种群组成与新鲜粪便相似,但是考克菌属(Kocuria spp.)、链球菌属(Streptococcus spp.)、德库菌属(Desemzia spp.)、芽孢杆菌属(Bacillus spp.)和鞘脂单胞菌属(Sphingomonas spp.)在堆积粪便中的分离株数量比新鲜粪便多,其中包括部分条件致病菌,表明堆积的奶牛粪便有利于细菌,包括条件致病菌的富集或繁殖。污水样品中分离株数量虽然最低,但其分离株中98.31%(58/59)为条件致病菌。因此,污水随意排放造成的安全隐患不容忽视。相对于新鲜粪便和堆积粪便,土壤样品中可培养细菌种群多样性较高,拥有较多样化的种群结构,但部分种属在3者间均有分布,包括克氏库克菌(Kocuria kristinae)、托尔豪特链球菌(Streptococcus thoraltensis)、小牛葡萄球菌(Staphylococcus vitulinus)、金黄色葡萄球菌(Staphylococcus aureus)、地衣芽胞杆菌(Bacillus licheniformis)和少动鞘氨醇单胞菌(Sphingomonas paucimobilis)等,说明养殖场周边环境土壤的可培养细菌种群结构会受到粪污中细菌的影响。这表明细菌可在动物机体(新鲜粪便)、养殖场粪污(堆积粪便和污水)和养殖场环境(土壤)间进行迁移[26-27],甚至可能会迁移到水和空气环境中[28-29],更为严重的是这些细菌所携带的耐药基因也会随之在环境中传播[30-31]。由此可见,如果养殖场粪污不经有效处理就排放到环境中,将会增加细菌性传染病发生的概率从而严重威胁人与动物的健康。
对样品中分离的条件致病菌数量及种类分析发现,实验过程中分离出的病原菌,多是各地区报道过的与奶牛乳房炎相关的病原菌,如大肠杆菌(Escherichia coli)、乳房链球菌(Streptococcus uberis)、金黄色葡萄球菌(Staphylococcus aureus)等[32-34]。有研究表明,牛奶中病原菌的主要来源与挤奶过程中奶牛粪便的污染直接相关[35]。因此如果奶牛养殖场粪污不经科学处理就直接排放或还田,将会对奶牛健康养殖造成影响,进而造成食源性污染,大大增加细菌性传染病发生风险,威胁人类健康[36-37]。系统研究奶牛养殖场粪污细菌群落分布及多样性,将会为奶牛养殖业健康有序发展提供理论依据。
| [1] |
丑天才, 杨波. 关于宁夏规模奶牛养殖模式的探讨[J]. 中国畜牧兽医文摘, 2017, 33(8): 33. (  0) 0) |
| [2] |
宁夏回族自治区统计局. 宁夏统计年鉴[M]. 北京: 中国统计出版社, 2017: 349-351. [ Ningxia Provinsial of Statistic. Ningxia Statistical Yearbook[M]. Beijing: China Statistics Press, 2017: 349-351.]
(  0) 0) |
| [3] |
DAI X R, SAHA C K, NI J Q, et al. Characteristics of Pollutant Gas Releases From Swine, Dairy, Beef, and Layer Manure, and Municipal Wastewater[J]. Water Research, 2015, 76: 110-119. DOI:10.1016/j.watres.2015.02.050 (  0) 0) |
| [4] |
JAHNE M A, ROGERS S W, HOLSEN T M, et al. Emission and Dispersion of Bioaerosols From Dairy Manure Application Sites:Human Health Risk Assessment[J]. Environmental Science & Technology, 2015, 49(16): 9842-9849. (  0) 0) |
| [5] |
BARAKAT A M A, MONA M S, EL FADALY H A A, et al. Zoonotic Hazards of Campylobacteriosis in Some Areas in Egypt[J]. Life Science Journal, 2015, 12(7): 9-14. (  0) 0) |
| [6] |
HEUER H, SCHMITT H, SMALLA K. Antibiotic Resistance Gene Spread Due to Manure Application on Agricultural Fields[J]. Current Opinion in Microbiology, 2011, 14(3): 236-243. DOI:10.1016/j.mib.2011.04.009 (  0) 0) |
| [7] |
SZMOLKA A, NAGY B. Multidrug Resistant Commensal Escherichia coli in Animals and Its Impact for Public Health[J]. Frontiers Microbiology, 2013, 4: 258. DOI:10.3389/fmicb.2013.00258 (  0) 0) |
| [8] |
SINTON L W, BRAITHWAITE R R, HALL C H, et al. Survival of Indicator and Pathogenic Bacteria in Bovine Feces on Pasture[J]. Applied and Environmental Microbiology, 2007, 73(24): 7917-7925. DOI:10.1128/AEM.01620-07 (  0) 0) |
| [9] |
JIANG X P, MORGAN J, Doyle M P. Fate of Escherichia coli O157:H7 in Manure-Amended Soil[J]. Applied and Environmental Microbiology, 2002, 68(5): 2605-2609. DOI:10.1128/AEM.68.5.2605-2609.2002 (  0) 0) |
| [10] |
GAO D, CHEN T B, LIU B, et al. Releases of Pollutants From Poultry Manure in China and Recommended Strategies for the Pollution Prevention[J]. Geographical Research, 2006, 25(2): 311-319. (  0) 0) |
| [11] |
布坎南RE, 吉本斯NE. 伯杰细菌鉴定手册[M]. 8版. 北京: 科学出版社, 1984: 274-1207.
(  0) 0) |
| [12] |
WILLEY J M, SHERWOOD L M, WOOLVERTON C J. Prescott's Principles of Microbiology[M]. 北京: 高等教育出版社, 2009: 713-721.
(  0) 0) |
| [13] |
农业部兽医局. 一二三类动物疫病释义[M]. 北京: 中国农业出版社, 2011: 406-446.
(  0) 0) |
| [14] |
MCAULEY C M, MCMILLAN K, MOORE S C, et al. Prevalence and Characterization of Foodborne Pathogens From Australian Dairy Farm Environments[J]. Journal of Dairy Science, 2014, 97(12): 7402-7412. DOI:10.3168/jds.2014-8735 (  0) 0) |
| [15] |
COLE K J, HOGAN J S. Environmental Mastitis Pathogen Counts in Freestalls Bedded With Composted and Fresh Recycled Manure Solids[J]. Journal of Dairy Science, 2016, 99(2): 1501-1505. DOI:10.3168/jds.2015-10238 (  0) 0) |
| [16] |
ONGENG D, GEERAERD A H, SPRINGAEL D, et al. Fate of Escherichia coli O157:H7 and Salmonella Enterica in the Manure-Amended Soil-Plant Ecosystem of Fresh Vegetable Crops[J]. Critical Reviews in Microbiology, 2015, 41(3): 273-294. DOI:10.3109/1040841X.2013.829415 (  0) 0) |
| [17] |
ZHAO W, ZHANG W, YANG F, et al. Enterocytozoon bieneusi in Dairy Cattle in the Northeast of China:Genetic Diversity of ITS Gene and Evaluation of Zoonotic Transmission Potential[J]. The Journal of Eukaryotic Microbiology, 2015, 62(4): 553-560. DOI:10.1111/jeu.2015.62.issue-4 (  0) 0) |
| [18] |
高凤.奶牛肠道微生物群落结构与多样性研究[D].邯郸: 河北工程大学, 2017. GAO Feng.The Study on Intestinal Microbial Community Structure and Diversity of Dairy Cows[D].Handan: Hebei University of Engineering, 2017. (  0) 0) |
| [19] |
AYENI F A, ANDERSEN C, NØRSKOV-LAURITSEN N. Comparison of Growth on Mannitol Salt Agar, Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Mass Spectrometry, VITEK© 2 With Partial Sequencing of 16S rRNA Gene for Identification of Coagulase-Negative Staphylococci[J]. Microbial Pathogenesis, 2017, 105: 255-259. DOI:10.1016/j.micpath.2017.02.034 (  0) 0) |
| [20] |
BOSSHARD P P, ZBINDEN R, ABELS S, et al. 16S rRNA Gene Sequencing Versus the API 20 NE System and the VITEK 2 ID-GNB Card for Identification of Nonfermenting Gram-Negative Bacteria in the Clinical Laboratory[J]. Journal of Clinical Microbiology, 2006, 44(4): 1359-1366. DOI:10.1128/JCM.44.4.1359-1366.2006 (  0) 0) |
| [21] |
FANG H, OHLSSON A K, ULLBERG M, et al. Evaluation of Species-Specific PCR, Bruker MS, VITEK MS and the VITEK 2 System for the Identification of Clinical Enterococcus Isolates[J]. European Journal of Clinical Microbiology & Infectious Diseases, 2012, 31(11): 3073-3077. (  0) 0) |
| [22] |
FANJAT N, LECLERCQ A, HAN J, et al. Comparison of the Phenotyping Methods ID 32E and VITEK 2 Compact GN With 16S rRNA Gene Sequencing for the Identification of Enterobacter sakazakii[J]. Journal of Clinical Microbiology, 2007, 45(6): 2048-2050. DOI:10.1128/JCM.00961-06 (  0) 0) |
| [23] |
叶小梅, 常志州, 陈欣, 等. 畜禽养殖场排放物病原微生物危险性调查[J]. 生态与农村环境学报, 2007, 23(2): 66-70. [ YE Xiao-mei, CHANG Zhi-zhou, CHEN Xin, et al. Hazard of Pathogenic Microorganisms in Discharge From Livestock and Poultry Breeding Farms[J]. Journal of Ecology and Rural Environment, 2007, 23(2): 66-70. DOI:10.3969/j.issn.1673-4831.2007.02.014] (  0) 0) |
| [24] |
李传海, 尚健, 聂文芳, 等. 家鸽粪便细菌多样性的高通量测序分析及其环境意义[J]. 南京农业大学学报, 2017, 40(1): 116-122. [ LI Chuan-hai, SHANG Jian, NIE Wen-fang, et al. Illumina Sequencing of 16S rDNA Tag Revealed Bacterial Communities in Pigeon Feces and Its Environmental Implication[J]. Journal of Nanjing Agricultural University, 2017, 40(1): 116-122.] (  0) 0) |
| [25] |
NICHOLSON F A, GROVES S J, CHAMBERS B J. Pathogen Survival During Livestock Manure Storage and Following Land Application[J]. Bioresource Technology, 2005, 96(2): 135-143. DOI:10.1016/j.biortech.2004.02.030 (  0) 0) |
| [26] |
DIMITRACOPOULOS G, KALKANIBOUSSIAKOU H, PAPAVASSILIOU J. Animal Fecal Carriership and Biotypes of Staphylococcus aureus[J]. Applied and Environmental Microbiology, 1977, 34(5): 461-464. (  0) 0) |
| [27] |
REUTER T, ALEXANDER T W, MCALLISTER T A. Viability of Bacillus licheniformis and Bacillus thuringiensis Spores as a Model for Predicting the Fate of Bacillus anthracis Spores During Composting of Dead Livestock[J]. Applied and Environmental Microbiology, 2011, 77(5): 1588-1592. DOI:10.1128/AEM.01889-10 (  0) 0) |
| [28] |
ZHOU B R, WANG C, ZHAO Q, et al. Prevalence and Dissemination of Antibiotic Resistance Genes and Coselection of Heavy Metals in Chinese Dairy Farms[J]. Journal of Hazardous Materials, 2016, 320: 10-17. DOI:10.1016/j.jhazmat.2016.08.007 (  0) 0) |
| [29] |
ROQUE K, LIM G D, JO J H, et al. Epizootiological Characteristics of Viable Bacteria and Fungi in Indoor Air From Porcine, Chicken, or Bovine Husbandry Confinement Buildings[J]. Journal of Veterinary Science, 2016, 17(4): 531-538. DOI:10.4142/jvs.2016.17.4.531 (  0) 0) |
| [30] |
王娟, 王新华, 徐海. 多重耐药菌在人类、动物和环境的耐药和传播机制[J]. 微生物学报, 2016, 56(11): 1671-1679. [ WANG Juan, WANG Xin-hua, XU Hai. Antimicrobial Resistance and Dissemination of Multidrug Resistant Organisms[J]. Acta Microbiologica Sinica, 2016, 56(11): 1671-1679.] (  0) 0) |
| [31] |
王娜, 王昝畅, 郭欣妍, 等. 兽药抗生素环境风险控制管理政策探析[J]. 生态与农村环境学报, 2017, 33(7): 586-591. [ WANG Na, WANG Zan-chang, GUO Xin-yan, et al. Management Policies for Control Environmental Risks of Veterinary Antibiotics[J]. Journal of Ecology and Rural Environment, 2017, 33(7): 586-591.] (  0) 0) |
| [32] |
魏凡华, 狄学珍, 牛耀祖, 等. 银川地区奶牛乳房炎流行病学调查与分析[J]. 畜牧与兽医, 2014, 46(12): 127-128. (  0) 0) |
| [33] |
吴亚文, 李志红, 杨佳冰, 等. 宁夏地区奶牛隐性乳房炎主要病原菌分离鉴定及药敏试验[J]. 中国动物检疫, 2015, 32(3): 4-7. [ WU Ya-wen, LI Zhi-hong, YANG Jia-bing, et al. Isolation and Identification of Subclinical Mastitis Pathogens and Drug Sensitivity Tests in Dairy Cows in Ningxia Autonomous Region[J]. China Animal Health Inspection, 2015, 32(3): 4-7. DOI:10.3969/j.issn.1005-944X.2015.03.002] (  0) 0) |
| [34] |
WANG D, ZHANG L, ZHOU X, et al. Antimicrobial Susceptibility, Virulence Genes, and Randomly Amplified Polymorphic DNA Analysis of Staphylococcus Aureus Recovered From Bovine Mastitis in Ningxia, China[J]. Journal of Dairy Science, 2016, 99(12): 9560-9569. DOI:10.3168/jds.2016-11625 (  0) 0) |
| [35] |
OLIVER S P, JAYARAO B M, ALMEIDA R A. Foodborne Pathogens in Milk and the Dairy Farm Environment:Food Safety and Public Health Implications[J]. Foodborne Pathogens and Disease, 2005, 2(2): 115-129. DOI:10.1089/fpd.2005.2.115 (  0) 0) |
| [36] |
魏琼, 沈梅, 刘翔, 等. 宁夏地区2006-2013年食品中食源性致病菌监测结果分析[J]. 医学动物防制, 2016(1): 31-34. [ WEI Qiong, SHEN Mei, LIU Xiang, et al. Pathogenic Bacteria Monitoring Results Analysis in Food From Ningxia Area During 2006-2013[J]. Chinese Journal of Pest Control, 2016(1): 31-34.] (  0) 0) |
| [37] |
MCAULEY C M, MCMILLAN K, MOORE S C, et al. Prevalence and Characterization of Foodborne Pathogens From Australian Dairy Farm Environments[J]. Journal of Dairy Science, 2014, 97(12): 7402-7412. DOI:10.3168/jds.2014-8735 (  0) 0) |




