2. 江苏省农业科学院果树研究所, 江苏 南京 210014
2. Institute of Pomology, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China
根际微生物的种群数量及多样性是评价林地土壤肥力高低的重要生物学指标, 而土壤肥力是土壤质量的重要组成部分, 是林地生产力的基础[1]。大量研究表明, 根际微生物对土壤肥力、植株抗逆性、生长发育以及生产力有着极其重要的作用[2-3]。根瘤菌和弗兰克氏菌等根际固氮菌能够提高作物产量, 增强植物对氮素的吸收[4]。假单胞菌属、芽孢杆菌属和农杆菌属等能通过产生生长激素或信号分子、竞争生长位点、产生铁载体等促生机制来促进植物生长, 增强对病原菌的抗性和忍耐力[5-7]。与此同时, 植物通过根际分泌物来调控和影响根际微生物的数量、种类及生态分布[8-9]。近年来, 利用生物工程技术在分子水平上研究植物根际微生物多样性的研究已有大量报道[10-11], 但大部分研究主要针对农作物, 林木根际土壤微生物多样性研究鲜有报道。因此, 开展林木根际土壤微生物多样性研究, 将有助于通过人工调节根际微环境的生物活性来改善林地的生态环境和质量, 从而为解决林木连栽引起的地力衰退和林木根部土传病害等问题以及增强林地的生产力提供技术支持。
薄壳山核桃(Carya illinoensis), 又名美国山核桃和长山核桃, 为胡桃科(Juglandaceae)山核桃属(Carya nutt)植物, 是我国主要的干果经济作物, 也是最早发现的化感植物之一[12]。目前, 对薄壳山核桃根际微生物多样性的研究鲜有报道。鉴于此, 笔者以江苏主产区的薄壳山核桃为研究对象, 利用高通量测序技术研究4个主要薄壳山核桃品种的根际土壤细菌区系组成和群落结构差异, 为薄壳山核桃根际微生态研究提供基础数据, 同时为进一步研究不同土壤环境以及营林方式对薄壳山核桃根际土壤微生物菌群结构的多样性影响提供理论基础。
1 材料与方法 1.1 研究区概况试验地位于泗洪县峰山乡大红山, 地处苏北平原西部, 属北亚热带和北暖温带季风气候区, 四季分明, 年均气温15 ℃, 年均日照时数2 206.2 h, 年均降水量960.4 mm, 年均无霜期203 d。试验地地形属岗坡地, 土壤为砂礓土, 基本理化指标如下:pH值为6.74, 有机碳含量为12.81 g·kg-1, 有机质含量为22.83 g·kg-1, 阳离子交换量为25.1 cmol·kg-1, 黏粒( < 0.002 mm)、粉粒(0.002~0.05 mm)、砂粒( > 0.05 mm)体积分数分别为7.4%、32.5%和52.2%。
供试材料为试验园结实稳定的4个品种:波尼(BN)、马汉(MH)、威斯顿(WSD)和肖肖尼(XXN), 树龄为8年生, 株行距为6 m×8 m, 南北行向, 树势较强。土壤采样时间为2015年5月, 是薄壳山核桃的果实灌浆期。
1.2 研究方法 1.2.1 土壤取样在上述4个品种薄壳山核桃种植区各选择1个20 m×30 m的标准地, 在每个标准地内选取3株树龄相同的薄壳山核桃树, 在距树干基部0.5 m处分别设置5个取样点, 用内径10 cm的土钻分层挖取0~20、> 20~40和 > 40~60 cm深度土壤, 采集各层直径0.1~0.5 cm的细根, 用抖落法获取其上粘附的土壤作为根际土壤, 将根际土壤分别混合装入已消毒的密封塑料袋中, 放入冰盒内带回实验室。在实验室内将同一品种的根际土壤充分混合后, 共形成4个根际土样, 一部分于-70 ℃条件下冷冻备用, 用于分析根际土壤细菌群落结构; 另一部分风干、过筛, 用于测定土壤理化性质。
1.2.2 土壤微生物DNA的提取采用MOBIO Power Soil DNA Isolation kit试剂盒(MOBIO公司, 美国)称取0.5 g新鲜土壤, 按照说明书的步骤进行提取。将提取得到的土壤微生物总DNA溶解于100 μL去离子水中, 取5 μL DNA用φ=1.0%的琼脂糖凝胶电泳检测, 分析DNA的完整性和相对浓度。
1.2.3 16S rRNA基因扩增及Miseq测序采用引物515F/806R (F5′-AYTGGGYDTAAAGNG-3′, R5′-AYTGGGYDTAAAGNG-3′)扩增16S rRNA的V4可变区片段[13]。PCR扩增程序如下:95 ℃预变性5 min; 95 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸90 s, 共25个循环; 最后72 ℃延伸10 min结束。获得的PCR产物进行琼脂糖凝胶电泳后, 使用AXYGEN公司的AxyPrep DNA凝胶回收试剂盒, 切胶回收PCR产物。将每个样品等比例混合后, 依托Illumina公司Miseq PE300平台进行测序(由上海生工生物技术有限公司提供)。
1.2.4 数据分析将Miseq双端测序数据, 首先根据单端测序序列(PE reads)之间的重叠关系, 用FLASH软件(Version 1.2.3) 进行拼接, 然后依托Prinseq软件(Version 0.20.4) 对低复杂度的序列进行质量控制过滤。采用RDP classifier软件对处理后序列进行物种分类, 依次为域、门、纲、目、科和属[14]。
1.2.5 微生物多样性及聚类分析利用Monthur软件(Version 1.35.1) 计算样品的α多样性指数, 反映不同品种薄壳山核桃根际土壤细菌群落的丰度和多样性, 包括Chao指数、ACE指数、Coverage指数、Simpson指数和Shannon指数[15]。
β多样性分析:基于物种分类分析, 采用RDP classifier将序列进行物种分类, 对不同样本和每个物种单元分类进行序列丰度计算, 构建代表性序列为节点的进化树与物种丰度矩阵, 分析比较不同薄壳山核桃品种根际细菌群落的同源性。
2 结果与分析 2.1 根际细菌的多样性指数分析将4种不同薄壳山核桃根际土壤样品的reads标准化后分析得到的OTU数量进行比较, 发现并无显著性差异。通过微生物多样性指数比较不同品种薄壳山核桃根际土壤的α多样性(表 1), 由ANOVA分析可知, 就不同薄壳山核桃品种的根际土壤微生物多样性比较而言, 除Chao指数有一定差异以外, 其他指数并无显著性差异(P > 0.05)。BN、MH、XXN和WSD的根际土壤微生物ACE指数分别为27 855、24 059、23 686和22 175。从Shannon指数分析来看, BN(8.067) 和MH (7.877) 的根际土壤微生物物种丰富度较高, 其次为XXN(7.631), WSD的根际土壤微生物物种丰富度最低(7.468)。就Chao指数而言, BN(19 848)、MH (18 008) 和XXN(16 861) 显著高于WSD (14 886)。可见, 以上3种多样性指数的计算结果相似, 均表明BN的根际土壤微生物多样性最高, WSD的根际土壤微生物多样性最低, MH和XXN的根际土壤微生物多样性在两者之间。
|
|
表 1 4种薄壳山核桃根际土壤细菌多样性指数 Table 1 Diversity index of the soil bacteria in the rhizosphere under Carya illinoensis relative to variety |
BN、MH、XXN和WSD的根际土壤分别含有19、20、20和21个门类菌群。4个薄壳山核桃品种根际土壤菌群中丰度最高的10大主要门类相同, 分别为变形菌门、类细菌门、芽单胞菌门、浮霉菌门、绿弯菌门、厚壁菌门、疣微菌门、硝化螺旋菌门、装甲菌门和酸杆菌门。由门水平菌群聚类堆积图(图 1)可以看出, BN和XXN根际细菌主要门水平相对丰度的同源性较高, 而WSD则与其他品种薄壳山核桃土壤根际细菌的相对丰度分布差异最大。XXN和WSD土壤根际菌群结构更为相似, 聚类于1簇。
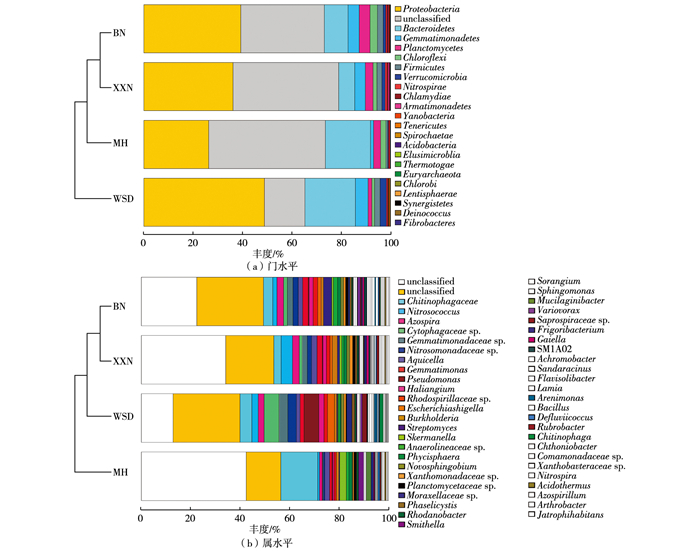
|
图 1 不同分类水平的根际细菌组成聚类堆积图 Figure 1 Cluster stacking graph of composition of the soil bacteria in rhizosphere relative to taxonomic level |
从其他分类层级上看, 4个薄壳山核桃品种的根际土壤微生物分布也较为相似, 从属水平上的菌群聚类堆积图(图 1)可以看出, BN和XXN土壤根际细菌在属水平的相对丰度同源性较高, 而MH则与其他品种土壤根际细菌的相对丰度分布差异最大。
主成分分析(PCA)结果(图 2)与聚类图结果一致, XXN和BN聚类于1簇。未鉴定到属的OTU以及丰度较低的微生物种群占50%左右, 其他优势菌属主要集中在50个种属间, 分别为亚硝化球菌属(Nitrosococcus)、Azospira、Aquicella、芽单胞菌属(Gemmatimonas)、假单胞菌属(Pseudomonas)、Hallangium和链霉菌属(Steptomyces)等, 丰度均在1%以上。其中Chitinophagaceae科在4种薄壳山核桃根际土壤样品中丰度均在5%以上, Chitinophagaceae科由Balneola、Filimonas、Flavisolibacter、Gracilimonas、Lacibacter、Niastella、Terrimonas和Chitinophaga共8个属组成[16]。
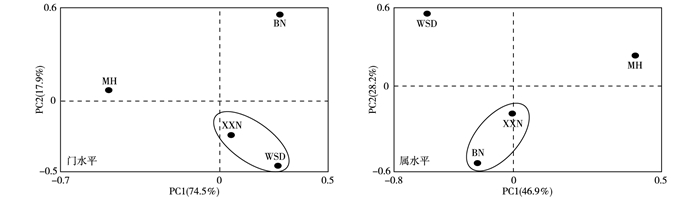
|
图 2 不同分类水平的根际细菌组成聚类主成分分析 Figure 2 Composition of the rhizosphere bacterial community at different taxonomic levels |
将4种薄壳山核桃品种根际土壤样品的OTU分布进行Venn图分析, 结果见图 3。4种薄壳山核桃的根际土壤样品中均有分布的OTU数量为2 145, 共有OTU数量最多的是BN和XXN(5 338), 最少是BN和WSD(3 450);3种美国薄壳山核桃品种的根际土壤样品共有最多的组合是BN、XXN和MH(3 429), 说明这3个品种根际土壤微生物群落组成的同源性较高, 该结果与图 1一致; 4种薄壳山核桃的根际土壤样品中独有OTU数从大到小依次为BN(2 634)、MH(2 378)、WSD(1 775) 和XXN(1 009)。
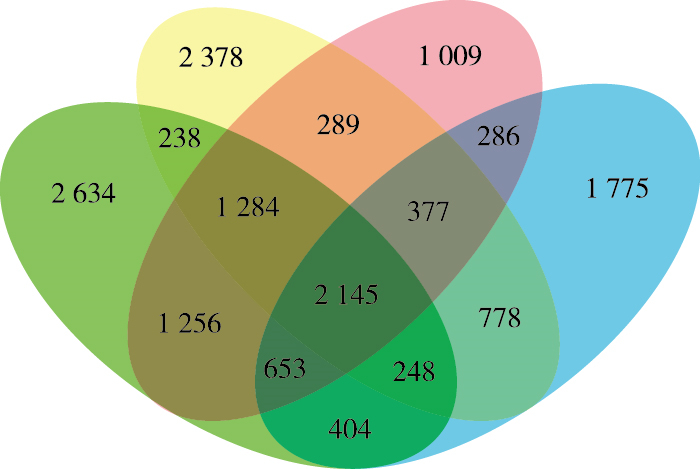
|
图 3 不同薄壳山核桃品种根际土壤样品OTU分布的Venn分析图 Figure 3 Venn diagrams of OTU analysis of soil samples from the rhizosphere of Carya illinoensis relative to variety 绿色、黄色、红色、蓝色分别代表波尼(BN)、马汉(MH)、肖肖尼(XXN)和威斯顿(WSD)这4个不同薄壳山核桃品种。图中数据代表不同品种山核桃根际土壤之间共有OTU数量。 |
在门分类水平上对4种薄壳山核桃根际土壤样品所含菌属进行聚类, 对聚类后的各样品中不同OTU所含序列丰度进行Heatmap分析(图 4), 该图能够反映门水平上各根际土壤样品细菌群落结构的差异性。变形菌门在4种薄壳山核桃的根际土壤微生物菌群中丰度最高。MH根际土壤细菌群落中黏胶球形菌门丰度高于其他3种薄壳山核桃品种; XXN根际土壤细菌群落的衣原体门和装甲菌门丰度高于其他3种薄壳山核桃品种; BN根际土壤细菌群落的蓝藻门丰度高于其他3种薄壳山核桃品种。
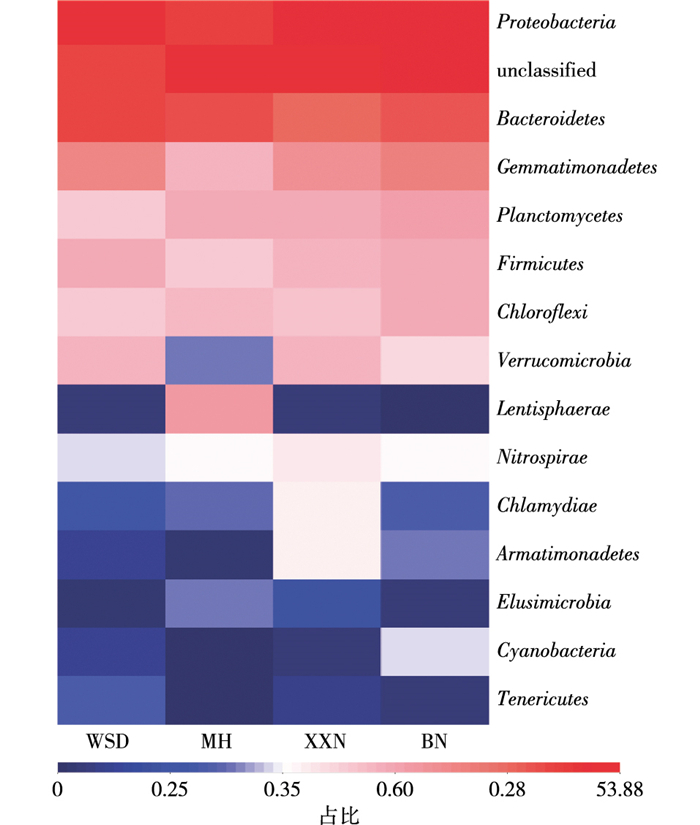
|
图 4 基于门分类水平的不同土壤样品OTU分布的Heatmap分析 Figure 4 Heatmap tree based on OTU analysis of soil bacteria at the phylum level in soil samples from the rhizosphere of Carya illinoensis relative to variety |
根际指受植物根部释放物质而影响微生物活性的土壤区域, 由于受植物种类、生长阶段和土壤性质等影响, 根际土壤微生物的数量和种类要多于根外的土壤微生物, 它们在根上的繁殖和分布受根系生长发育的影响而表现出较为明显的根际效应。由于根际土壤微生物在植物的物质循环和能量转换中扮演着重要角色, 一定程度上决定着植物生产力的高低[17]。因此, 根际土壤微生物多样性一直以来是根际效应研究的主体[18-19]。该研究基于高通量测序技术对4种薄壳山核桃的根际土壤细菌多样性进行了初步研究, α多样性指数显示, 4种薄壳山核桃的根际土壤细菌多样性无显著性差异; β多样性分析结果表明, 4种薄壳山核桃的根际土壤细菌群落结构在门、属水平相似, 但在低丰度的根际细菌群落结构方面存在一定特异性。变形菌门、类细菌门和芽单胞菌门在4种薄壳山核桃的根际土壤细菌中都属于优势菌群。从属水平和OUT多样性分析结果来看, BN、XXN和MH的根际土壤微生物群落组成的同源性较高, 但不同品种薄壳山核桃根际土壤的优势菌门存在差异性, 这也说明薄壳山核桃根际土壤菌群结构存在一定的种间差异。有研究发现, 不同种的海棠根系代谢活力存在一定差异, 导致其根系分泌物的物质数量和组成不同, 进而直接或间接影响根际土壤微生物的数量和种群结构[20]。
化感植物通过根系分泌各种化感物质对根际微生物的种类、数量和分布产生影响, 进一步对根际定殖的微生物分类群和功能群进行选择[10]。水稻根际存在30多种特异微生物, 其中7种被鉴定为黏细菌(myxobacteria), 进一步研究发现, 黏细菌对水稻根系分泌物中的酚酸类物质具有明显的趋化促进作用[11]。薄壳山核桃根系分泌的胡桃醌在土壤种性质极其稳定, 且不随季节发生改变, 是一种典型的化感植物[8]。有研究表明, 假单胞菌属能通过自身水解酶降解胡桃醌等化感物质而在核桃根际有效定植[9], 笔者的研究结果与其一致。Chitinophagaceae菌群在4种薄壳山核桃根际土壤中丰度较高, 而大量研究结果也发现该菌群广泛存在于水稻[4]、小麦[5]、人参[6]和紫茎泽兰[10]等产化感物质的植物根际土壤中。因此, 推测薄壳山核桃根系的化感物质胡桃醌可能诱导假单胞菌属和Chitinophagaceae菌群这类具有趋化性的细菌在其根际中大量定植。
土壤理化因子、林木种类及不同营林方式是影响林木根际微生物群落结构的主要因素[21]。化感植物通过根系分泌各种化感物质对根际微生物的种类、数量和分布产生影响, 进一步对根际定殖的微生物分类群和功能群进行选择[22-23]。土壤温度、pH值及有机质含量通过影响土壤微生物的数量与种类间接影响根际微生物的多样性[24-25]。哪些环境因子影响着薄壳山核桃的根际细菌多样性?不同营林方式对薄壳山核桃根际细菌多样性的影响如何?这些问题还需通过进一步试验开展深入研究。
综上所述, 笔者通过Miseq高通量测序技术对4种薄壳山核桃根际土壤细菌多样性及菌群结构进行了分析, 发现薄壳山核桃根际土壤的细菌以变形菌门和类细菌门为主要优势门类, 同时发现了薄壳山核桃根际土壤中含有的特有菌属主要是假单胞菌属和Chitinophagaceae菌群, 关于这2类菌群对胡桃醌的趋化性研究仍需一步开展。
| [1] |
白晓旭, 史荣久, 尤业明, 等. 河南宝天曼不同林龄与林型森林土壤的细菌群落结构与多样性[J]. 应用生态学报, 2015, 26(8): 2273-2281. BAI Xiao-xu, SHI Rong-jiu, YOU Ye-ming, et al. Bacterial Community Structure and Diversity in Soils of Different Forest Ages and Types in Baotianman Forest, Henan Province, China[J]. Chinese Journal of Applied Ecology, 2015, 26(8): 2273-2281. (  0) 0) |
| [2] |
EISENHAUER N, SCHEU S, JOUSSET A. Bacterial Diversity Stabilizes Community Productivity[J]. PLoS One, 2012, 7(3): e34517. DOI:10.1371/journal.pone.0034517 (  0) 0) |
| [3] |
吴林坤, 林向民, 林文雄. 根系分泌物介导下植物-土塘-微生物互作关系研究进展与展望[J]. 植物生态学报, 2014, 38(3): 298-310. WU Lin-kun, LIN Xiang-min, LIN Wen-xiong. Advances and Perspective in Research on Plant-Soil-Microbe Interactions Mediated by Root Exudates[J]. Chinese Journal of Plant Ecology, 2014, 38(3): 298-310. (  0) 0) |
| [4] |
ADESEMOYE A O, KLOEPPER J W. Plant-Microbes Interactions in Enhanced Fertilizer-Use Efficiency[J]. Applied Microbiology and Biotechnology, 2009, 85(1): 1-12. DOI:10.1007/s00253-009-2196-0 (  0) 0) |
| [5] |
康贻军, 程洁, 梅丽娟, 等. 植物根际促生菌作用机制研究进展[J]. 应用生态学报, 2010, 21(1): 232-238. KANG Yi-jun, CHENG Jie, MEI Li-juan, et al. Action Mechanisms of Plant Growth-Promoting Rhizobacteria (PGPR):A Review[J]. Chinese Journal of Applied Ecology, 2010, 21(1): 232-238. (  0) 0) |
| [6] |
JETIYANON K, PLIANBANGCHANG P. Potential of Bacillus Cereus Strain RS87 for Partial Replacement of Chemical Fertilisers in the Production of Thai Rice Cultivars[J]. Journal of the Science of Food and Agriculture, 2012, 92(5): 1080-1085. DOI:10.1002/jsfa.v92.5 (  0) 0) |
| [7] |
RICHARDSON A E, BAREA J M, MCNEILL A M, et al. Acquisition of Phosphorus and Nitrogen in the Rhizosphere and Plant Growth Promotion by Microorganisms[J]. Plant and Soil, 2009, 321(1): 305-339. (  0) 0) |
| [8] |
孙婧钰, 孙跃志. 林木根系与根际微生物的相互作用[J]. 世界林业研究, 2015, 28(2): 14-18. SUN Jing-jue, SUN Yun-zhi. Interaction Between Tree Roots and Soil Microorganisms in Rhizosphere[J]. World Forestry Research, 2015, 28(2): 14-18. (  0) 0) |
| [9] |
ZHOU X, WU F. P-Coumaric Acid Influenced Cucumber Rhizosphere Soil Microbial Communities and the Growth of Fusarium oxysporum f.sp.Cucumerinum Owen[J]. PLoS One, 2012, 7(10): e48288. DOI:10.1371/journal.pone.0048288 (  0) 0) |
| [10] |
朱珣之, 李强, 李扬苹, 等. 紫茎泽兰入侵对土壤细菌的群落组成和多样性的影响[J]. 生物多样性, 2015, 23(5): 665-672. ZHU Xun-zhi, LI Qiang, LI Yang-ping, et al. Eupatorium adenophorum Invasion Alters Soil Bacterial Community and Diversity[J]. Biodiversity Science, 2015, 23(5): 665-672. DOI:10.17520/biods.2014158 (  0) 0) |
| [11] |
JOSE S, GILLESPIE A R. Allelopathy in Black Walnut (Juglans nigra L.) Alley Cropping:Ⅰ. Spatio-Temporal Variation in Soil Juglone in a Black Walnut-Corn (Zea mays L.) Alley Cropping System in the Midwestern USA[J]. Plant and Soil, 1998, 203(2): 191-197. DOI:10.1023/A:1004301309997 (  0) 0) |
| [12] |
崔翠, 蔡靖, 张硕新. 核桃根系分泌物化感物质的分离与鉴定[J]. 林业科学, 2013, 49(2): 54-60. CUI Cui, CAI Jing, ZHANG Shuo-xin. Isolation and Identification of the Allelochemicals in Walnut (Juglans regia) Root Exudates[J]. Scientia Silvae Sinicae, 2013, 49(2): 54-60. DOI:10.11707/j.1001-7488.20130209 (  0) 0) |
| [13] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Global Patterns of 16S rRNA Diversity at a Depth of Millions of Sequences Per Sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl. 1): 4516-4522. (  0) 0) |
| [14] |
李靖宇, 张琇, 孙敏, 等. 腾格里沙漠沙坡头地区土壤微生物多样性分析[J]. 生态与农村环境学报, 2016, 32(5): 780-787. LI Jing-yu, ZHANG Xiu, SUN Min, et al. Analysis of Microbial Diversity in Shapotou Area of Tengger Desert[J]. Journal of Ecology and Rural Environment, 2016, 32(5): 780-787. DOI:10.11934/j.issn.1673-4831.2016.05.014 (  0) 0) |
| [15] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME Allows Analysis of High-Throughput Community Sequencing Data[J]. Nature Methods, 2010, 7: 335-336. DOI:10.1038/nmeth.f.303 (  0) 0) |
| [16] |
ROSENBERG E. The Prokaryotes[M]. Berlin, Germany: Springer, 2014, 493-495.
(  0) 0) |
| [17] |
GOBAT J M, ARAGNO M, MATTHEY W. The Living Soil:Fundamentals of Soil Science and Soil Biology[M]. New York, USA: Science Publishers, 2004, 622.
(  0) 0) |
| [18] |
BANNING N C, GLEESON D B, GRIGG A H, et al. Soil Microbial Community Successional Patterns During Forest Ecosystem Restoration[J]. Applied and Environmental Microbiology, 2011, 77(17): 6158-6164. DOI:10.1128/AEM.00764-11 (  0) 0) |
| [19] |
HARRIS J. Soil Microbial Communities and Restoration Ecology:Facilitators or Followers?[J]. Science, 2009, 325(5940): 573-574. DOI:10.1126/science.1172975 (  0) 0) |
| [20] |
陈汝, 王海宁, 姜远茂, 等. 不同苹果砧木的根际土壤微生物数量及酶活性[J]. 中国农业科学, 2012, 45(10): 2099-2106. CHEN Ru, WANG Hai-ning, JIANG Yuan-mao, et al. Rhizosphere Soil Microbial Quantity and Enzyme Activity of Different Apple Rootstocks[J]. Scientia Agricultura Sinica, 2012, 45(10): 2099-2106. DOI:10.3864/j.issn.0578-1752.2012.10.024 (  0) 0) |
| [21] |
GRIFFITHS R I, THOMSON B C, JAMES P, et al. The Bacterial Biogeography of British Soils[J]. Environmental Microbiology, 2011, 13(6): 1642-1654. DOI:10.1111/j.1462-2920.2011.02480.x (  0) 0) |
| [22] |
CHUNG E J, PARK T S, JEON C O, et al. Chitinophaga oryziterrae sp Nov., Isolated From the Rhizosphere Soil of Rice (Oryza sativa L.)[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(12): 3030-3035. (  0) 0) |
| [23] |
YIN C, HULBERT S, SCHROEDER K, et al.Natural Suppression of Rhizoctonia Root Rot by Soil Microbial Communities in Wheat From a Rhizoctonia Decline Site[C]//:Annual Meeting of the American Phytopathological Society (APS), 2012.
(  0) 0) |
| [24] |
BARENDSEN R L, PIETERSE C M, BAKKER P A. The Rhizosphere Microbiome and Plant Health[J]. Trends in Plant Science, 2012, 17(8): 478-486. DOI:10.1016/j.tplants.2012.04.001 (  0) 0) |
| [25] |
EAST R. Microbiome:Soil Science Comes to Life[J]. Nature, 2013, 501(7468): 18-19. DOI:10.1038/501S18a (  0) 0) |




