理解蛋白质的氨基酸序列与其结构之间的潜在关系是结构生物信息学中的一个基本问题[1]。蛋白质二级结构是氨基酸残基在蛋白质多肽链中的局部空间构象,其具有8种类型[2],分别是α-螺旋 (H)、β-桥 (B)、折叠 (E)、螺旋-3(G)、螺旋-5(I)、转角 (T)、卷曲 (S) 和环 (L)。每个二级结构类型都是由其所在蛋白质链中氨基酸残基间的局部和长程相互作用共同决定的。蛋白质二级结构预测任务就是把一个由20种氨基酸A、C、D、E、F、G、H、I、K、L、M、N、P、Q、R、S、T、V、W和Y组成的氨基酸序列映射为对应的二级结构序列。与蛋白质二级结构预测相关的问题有蛋白质折叠预测[3]和蛋白质三级结构预测。特别地,蛋白质的二级结构预测信息不仅有助于确定蛋白质的三维结构,而且能够用于蛋白质的功能预测与互作预测[4]。
早期的蛋白质二级结构预测研究主要集中在粗粒度的三类二级结构预测,即把八类二级结构粗略地归类为螺旋、折叠和卷曲三类, 代表的算法有PHD[5]、PSIPRED[6]和JPred[7]等。相对于三类二级结构预测,细粒度的八类二级结构预测能够提供更为丰富的信息,并且更具有挑战性。针对八类蛋白质二级结构预测,文献[8]基于双向朴素递归神经网络提出了预测算法SSpro8。考虑到SSpro8不能建模相邻残基二级结构类型之间的依赖关系,文献[9]提出采用条件神经场构建二级结构预测模型。此外,基于结构模板,文献[10]提出了八类二级结构预测算法C8-SCORPION。通过采用带卷积的生成随机网络 (Generative Stochastic Network, GSN),文献[11]获得了66.4%的Q8预测精度。
深度学习是一种通过多个非线性处理层来建模数据中抽象信息的机器学习技术。由于深度学习具有较强的建模能力并且能够基于数据自适应地进行参数学习,因此其已成功地应用于图像分类[12]、物体检测[13]、人脸识别[14]、人类行为识别[15]、图像识别[16]和图像描述生成[17]等多个领域。为此,本文提出了一种基于深度模型的八类蛋白质二级结构预测算法,并与其他八类蛋白质二级结构算法进行了比较。实验结果表明,提出的算法能够获得较好的预测精度。
1 氨基酸的编码为了基于氨基酸序列预测蛋白质二级结构,需要为序列中的每个氨基酸构造数值编码。当前常用的编码是正交编码和位置特异性计分矩阵 (Position-Specific Scoring Matrices, PSSM) 谱编码。图 1给出了氨基酸的21位正交编码。图中的前20个字母A、C、E、D、G、F、I、H、K、M、L、N、Q、P、S、R、T、W、V和Y是构成蛋白质链的基本氨基酸,冒号后面0和1组成的序列是对应的正交编码。显然,任意两个氨基酸编码向量的内积都为0。除了上述20个字母以外,在蛋白质序列数据库中额外地引入了字母X表示氨基酸的具体类型未知,这是由于实验方法有时不能确定一个氨基酸的具体类型。图 1也给出了X对应的21位正交编码。

|
图 1 氨基酸的正交编码 Figure 1 Orthogonal encoding of amino acids |
PSSM谱编码是通过把目标蛋白质链与蛋白质序列数据库中的蛋白质链进行多序列比对获得的。为了生成目标蛋白质链的PSSM谱编码,需要把NCBI nr (ftp://ftp.uniprot.org/pub/databases/uniprot/uniref/) 数据库下载到本机。在NCBI nr数据库中,有基于序列一致性阈值100%、90%和50%分别构造的三种非冗余蛋白质数据库,本文采用序列一致性为50%的数据库uniref50.fasta。对于uniref50.fasta,需要首先调用pfilt程序对其蛋白质序列数据进行过滤,以便删除卷曲-卷曲片段、跨膜区域和低复杂性区域; 然后调用makeblastdb程序对其进行格式化生成便于多序列比对的数据和索引文件。在处理完uniref50.fasta数据库后,可调用3次迭代的psiblast程序把目标蛋白质链的氨基酸序列与数据库中的蛋白质链进行多序列比对。在迭代完成后,psiblast程序会生成目标蛋白质链的PSSM谱编码文件。图 2给出了蛋白质4Y3F的部分氨基酸的PSSM谱编码。

|
图 2 蛋白质4Y3F的前26个氨基酸的PSSM谱编码 Figure 2 PSSM profile encoding of the first 26 amono acid residues of the protein 4Y3F |
图 2中,最左边的数字是氨基酸在蛋白质链中的编号,字母表示氨基酸,字母右边的20个数是对其的数值编码。比如,对于第12个氨基酸S,图中虚线长方形中的20个数值组成的向量就是它的PSSM谱编码。此外,在使用PSSM谱编码之前,本文进一步采用Sigmoid函数把编码中的整数值映射到0~1。
2 蛋白质二级结构的深度预测模型蛋白质二级结构预测是氨基酸序列到二级结构序列的映射问题。为了按照序列方式预测蛋白质二级结构,本文通过组合递归神经网络和前馈神经网络构造深度神经网络预测模型。图 3给出了具体的深度预测模型。特别地,预测模型的具体数据处理流程如下:首先氨基酸序列中的每个氨基酸通过氨基酸编码形式化为数值向量送入到递归神经网络——双向长短时记忆模型 (Long Short-Term Memory, LSTM) 中,然后组合双向LSTM的前向和后向隐层输出送入到前馈神经网络的输入层中,最后根据前馈神经网络输出层的结果确定预测的蛋白质二级结构序列并输出。
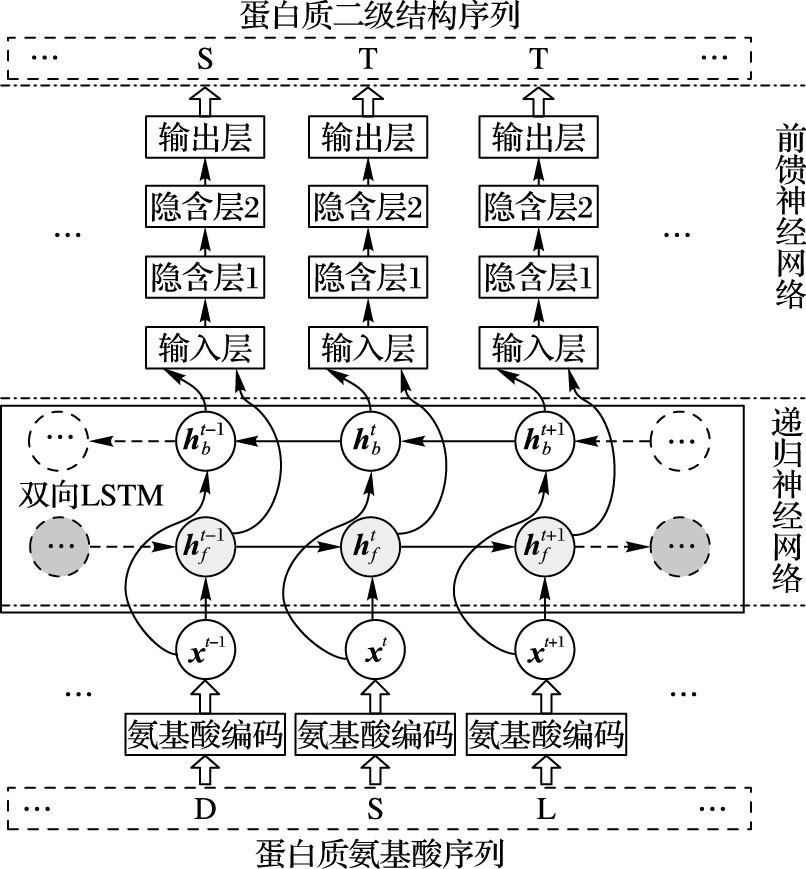
|
图 3 蛋白质二级结构的深度预测模型 Figure 3 Deep prediction model of protein secondary structure |
对于氨基酸序列,本文采用组合的PSSM谱编码和21位正交编码表示氨基酸,因此每个氨基酸可由长度为41的特征向量表示。当用xt表示蛋白质链中第t个位置氨基酸的特征向量时,则长度为τ的蛋白质可形式化为序列数据{x1, …, xt, …, xτ}。此外,对应的目标二级结构序列可表示为{y1, …, yt, …, yτ},其中yt是处于第t个位置的二级结构类型的8位正交编码。
由于单个氨基酸的二级结构类型是由蛋白质链中的整个序列信息决定的,因此我们采用双向LSTM处理数值化的氨基酸序列信息。对于双向LSTM,前向LSTM在t时刻可以获取目标xt以前的序列信息x1, x2, …, xt,后向LSTM对应的获取目标xt后面的序列信息xt, xt+1, …, xτ。通过组合双向LSTM在t时刻的两个隐层输出向量hft和hbt,可以获取整个氨基酸序列的信息。特别地,LSTM的基本单元由下面的方程定义:
| $\begin{array}{l} {\mathit{\boldsymbol{i}}^t} = \sigma \left( {{\mathit{\boldsymbol{U}}_i}{\mathit{\boldsymbol{x}}^t} + {\mathit{\boldsymbol{V}}_i}{\mathit{\boldsymbol{h}}^{t - 1}} + {\mathit{\boldsymbol{b}}_i}} \right)\\ {\mathit{\boldsymbol{o}}^t} = \sigma \left( {{\mathit{\boldsymbol{U}}_o}{\mathit{\boldsymbol{x}}^t} + {V_o}{h^{t - 1}} + {\mathit{\boldsymbol{b}}_o}} \right)\\ {\mathit{\boldsymbol{f}}^t} = \sigma \left( {{\mathit{\boldsymbol{U}}_f}{\mathit{\boldsymbol{x}}^t} + {\mathit{\boldsymbol{V}}_f}{\mathit{\boldsymbol{h}}^{t - 1}} + {\mathit{\boldsymbol{b}}_f}} \right)\\ {\mathit{\boldsymbol{g}}^t} = {\mathop{\rm Tanh}\nolimits} \left( {{\mathit{\boldsymbol{U}}_g}{\mathit{\boldsymbol{x}}^t} + {V_g}{\mathit{\boldsymbol{h}}^{t - 1}} + {\mathit{\boldsymbol{b}}_g}} \right)\\ {\mathit{\boldsymbol{s}}^t} = {\mathit{\boldsymbol{s}}^{t - 1}} \odot {\mathit{\boldsymbol{f}}^t} + {\mathit{\boldsymbol{i}}^t} \odot {\mathit{\boldsymbol{g}}^t}\\ {\mathit{\boldsymbol{h}}^t} = {\mathop{\rm Tanh}\nolimits} ({\mathit{\boldsymbol{s}}^t}) \odot {\mathit{\boldsymbol{o}}^t} \end{array}$ |
其中:符号⊙表示两个向量按元素依次相乘,it、ot、ft、gt和st分别是t时刻的输入网关向量、输出网关向量、遗忘网关向量、候选节点向量和内部记忆向量,Ui、Uo、Uf、Ug、Vi、Vo、Vf和Vg是需要学习的权值矩阵,bi、bo、bf和bg是偏置向量。通过引入网关和内部记忆机制,LSTM不仅可以克服消失的梯度问题,而且能够显式地建模序列数据之间的长距离依赖关系。特别地,通过学习模型参数,LSTM可以针对不同的任务自适应地学习采用何种记忆机制以便建模序列数据的长距离依赖关系。
对于前馈神经网络,本文采用三层的多层感知机 (MultiLayer Perceptron)。多层感知机的输入层是由双向LSTM的前向和后向隐层输出向量hft和hbt组成的特征向量,两个隐含层都采用校正激活单元 (Rectified Linear Unit, ReLU) 作为激活函数,输出层通过采用Softmax函数可以计算预测的8类二级结构概率分布。给定输入序列数据x={x1, …, xt, …, xτ},则深度预测模型将输出对应的序列数据
| $\mathit{\boldsymbol{\xi }}(\hat y,y) = - \sum\limits_{t = 1}^\tau {\sum\limits_{k = 1}^8 {y_k^t\ln \hat y_k^t} } $ |
当训练数据包括n个序列对时,深度预测模型的目标损失函数就是n个序列对对应损失函数的均值。通过采用基于时间的反向传播算法 (Back Propagation Through Time, BPTT) 最小化深度预测模型的目标函数,可以自适应地学习双向LSTM和多层感知机的参数。
3 实验 3.1 数据集本文采用文献[11]使用的非冗余蛋白质二级结构数据集PISCES CullPDB训练深度预测模型,该数据集包含6 128个蛋白质链,由PISCES CullPDB服务器按照蛋白质分辨率不大于2.5和蛋白质序列一致性小于30%的约束条件生成。同时,常用的CB513数据集被当作测试集,以便评估提出算法的分类性能。为了消除训练数据与测试数据之间的序列相似性,本文进一步从PISCES CullPDB数据集中删除与CB513中的蛋白质链存在序列一致性大于25%的蛋白质链。通过删除操作,PISCES CullPDB数据集最后剩下5 534条蛋白质链。对于剩下的蛋白质链,本文选取5 278个蛋白质链作为深度预测模型的训练数据集,余下的256条蛋白质链作为验证集。引入验证集的目的是为了采用早期停止方法防止过训练,也就是说,当训练模型在验证集上的验证误差不再降低时停止参数训练。
3.2 实验结果与分析为了进行实验比较,需要对提出的深度预测模型进行超参数设置。对于双向LSTM,隐层单元个数设置为96,初始隐层状态设为零向量。对于多层感知机,两个隐层节点的个数都设置为192,激活函数采用ReLU,输出层采用输出类别为8类的Softmax函数。此外,本文采用带冲量的随机梯度下降算法训练网络参数,其中冲量系数设为0.9,学习率为0.01,minibatch的大小设置为128,梯度剪切的阈值设置为5。当网络在验证集上的预测精度不再增加时停止训练。
表 1给出了本文算法在CB513数据集上的混淆矩阵,其中粗体字标出了8种蛋白质二级结构类型α-螺旋 (H)、β-桥 (B)、折叠 (E)、螺旋-3(G)、螺旋-5(I)、转角 (T)、卷曲 (S) 和环 (L) 的预测正确率。从表中可以看出,α-螺旋和折叠正确率最高,分别达到了91.98%和81.92%;β-桥和螺旋-5的正确率最低,分别为1.19%和0%,这主要是由于它们在蛋白质结构数据库所占的比率极低的缘故。特别地,螺旋-5出现的比率只有0.000 2。因此,八类蛋白质二级结构预测是不平衡分类问题。
| 表 1 本文算法在CB513测试集上的混淆矩阵 % Table 1 Confusion matrix of the proposed algorithm on the CB513 dataset % |
表 2给出了本文算法与4种算法SC-GSN (Supervised Convolutional-Generative Stochastic Network)[11]、RaptorX-SS8[9]、SSpro8[8]和LSTM large[18]在CB513数据集上的Q8预测精度。对于所比较的4个算法,SC-GSN采用的是带卷积的生成随机网络,RaptorX-SS8使用的预测模型是条件神经场,SSpro8采用的是双向朴素递归神经网络,LSTM large使用双向LSTM。从表中可以看出,本文算法获得了最高的预测精度,并且明显地优于SSpro8和SC-GSN。
| 表 2 CB513数据集上的分类性能比较 Table 2 Performance comparision on the CB513 dataset |
针对八类蛋白质二级结构预测,本文通过组合递归神经网络和前馈神经网络提出了一种新的基于深度学习的预测算法。该方法首先采用双向LSTM模型处理氨基酸序列数据,以便建模氨基酸之间的长距离依赖关系。然后,双向LSTM的两个隐层输出被组合成单个特征向量进一步送入到三层的多层感知机。最后,采用交叉熵作为目标函数以便训练深度网络参数。特别地,通过采用递归神经网络,提出的算法能够直接进行氨基酸序列到蛋白质二级结构序列的预测。CB513数据集上的实验结果表明,本文算法能够获得较好的预测精度,并且明显地优于SSpro8和SC-GSN。
| [1] | CHENG J, TEGGE A N, BALDI P. Machine learning methods for protein structure prediction[J]. IEEE Reviews in Biomedical Engineering, 2008, 1: 41-49. doi: 10.1109/RBME.2008.2008239 |
| [2] | TOUW W G, BAAKMAN C, BLACK J, et al. A series of PDB-related databanks for everyday needs[J]. Nucleic Acids Research, 2015, 43(D1): 364-368. doi: 10.1093/nar/gku1028 |
| [3] | LIN C, ZOU Y, QIN J, et al. Hierarchical classification of protein folds using a novel ensemble classifier[J]. PLOS ONE, 2013, 8(2): e56499. doi: 10.1371/journal.pone.0056499 |
| [4] | ROST B. Review:protein secondary structure prediction continues to rise[J]. Journal of Structural Biology, 2001, 134(2): 204-218. |
| [5] | ROST B, SANDER C. Prediction of protein secondary structure at better than 70% accuracy[J]. Journal of Molecular Biology, 1993, 232(2): 584-599. doi: 10.1006/jmbi.1993.1413 |
| [6] | BUCHAN D W, WARD S, LOBLEY A E, et al. Protein annotation and modelling servers at university college london[J]. Nucleic Acids Research, 2010, 38(Web Server issue): Web Server issue-W568. |
| [7] | DROZDETSKIY A, COLE C, PROCTER J, et al. JPred4:a protein secondary structure prediction server[J]. Nucleic Acids Research, 2015, 43(1): 389-394. |
| [8] | POLLASTRI G, PRZYBYLSKI D, ROST B, et al. Improving the prediction of protein secondary structure in three and eight classes using recurrent neural networks and profiles[J]. Proteins:Structure, Function, and Bioinformatics, 2002, 47(2): 228-235. doi: 10.1002/(ISSN)1097-0134 |
| [9] | WANG Z, ZHAO F, PENG J, et al. Protein 8-class secondary structure prediction using conditional neural fields[J]. Proteomics, 2011, 11(19): 3786-3792. doi: 10.1002/pmic.v11.19 |
| [10] | YASEEN A, LI Y. Template-based C8-SCORPION:a protein 8-state secondary structure prediction method using structural information and context-based features[J]. BMC Bioinformatics, 2014, 15(8): 1-8. |
| [11] | ZHOU J, TROYANSKAYA O G. Deep supervised and convolutional generative stochastic network for protein secondary structure prediction[EB/OL].[2016-06-20]. https://core.ac.uk/download/pdf/25008331.pdf. |
| [12] | KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[C]//Proceedings of the 25th International Conference on Neural Information Processing Systems. Red Hook:Curran Associates Inc, 2012:1097-1105. |
| [13] | GUPTA S, GIRSHICK R, ARBELÁEZ P, et al. Learning rich features from RGB-D images for object detection and segmentation[C]//Proceedings of the 13th European Conference on Computer Vision. Berlin:Springer, 2014:345-360. |
| [14] | 张雯, 王文伟. 基于局部二值模式和深度学习的人脸识别[J]. 计算机应用, 2015, 35(5): 1474-1478. ( ZHANG W, WANG W W. Face recognition based on local binary pattern and deep learning[J]. Journal of Computer Applications, 2015, 35(5): 1474-1478. doi: 10.11772/j.issn.1001-9081.2015.05.1474 ) |
| [15] | JI S, XU W, YANG M, et al. 3D convolutional neural networks for human action recognition[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2013, 35(1): 221-231. doi: 10.1109/TPAMI.2012.59 |
| [16] | 康晓东, 王昊, 郭军, 等. 无监督深度学习彩色图像识别方法[J]. 计算机应用, 2015, 35(9): 2636-2639. ( KANG X D, WANG H, GUO J, et al. Unsupervised deep learning method for color image recognition[J]. Journal of Computer Applications, 2015, 35(9): 2636-2639. doi: 10.11772/j.issn.1001-9081.2015.09.2636 ) |
| [17] | KARPATHY A, LI F F. Deep visual-semantic alignments for generating image descriptions[C]//Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway, NJ:IEEE, 2015:3128-3137. |
| [18] | SØNDERBY S K, WINTHER O. Protein secondary structure prediction with long short term memory networks[EB/OL].[2016-06-20]. http://arxiv-web1.library.cornell.edu/pdf/1412.7828.pdf. |




