2. 中国科学院 自动化研究所, 北京 100190
2. Institute of Automation, Chinese Academy of Sciences, Beijing 100190, China
LI Yan, born in 1987, Ph. D. candidate. His research interests include information retrieval, deep learning.
ZHANG Bowen, born in 1992, Ph. D. candidate. His research interests include information retrieval.
HAO Hongwei, born in 1967, Ph. D., professor. His research interests include pattern recognition, machine learning.
随着信息时代的到来,源源不断的信息从互联网中涌现,对信息的检索已是用户日常生活中不可缺少的一部分。由于信息种类之多,内容涉及范围之广,这使得从海量信息中准确定位到满足用户需求的信息成为一个亟待解决的问题。然而用户的需求变得越来越模糊,用户甚至可能不知道与需求相关的关键词进而给定一些简短的查询语句或几个查询词。在这种情况下,仅使用用户给出的少量查询信息进行关键词匹配可能找不到相关的文档或信息[1]。因此,根据用户给定的查询语句,对其进行内容扩展方法的研究是势在必行的。
用户提出的查询语句是进行信息检索的主要依据,但查询语句中所提供的少量信息会导致检索结果不够准确,导致信息量不足的原因主要体现在两个方面:1)在复杂专业领域检索时,用户由于知识受限,使得提出的查询内容不能满足检索的需求;2)由于表达和叙述方式的不同,基于关键词匹配的检索方法不能检索出相关的结果。查询扩展可从一定程度上弥补检索过程中用户提供信息量不足的问题[2],它在原有查询语句基础上,通过多种方式和策略对查询语句中的词进行扩展,用更为丰富的查询词进行信息检索。
扩展查询词的选择是查询扩展最关键的步骤之一,当前国内外研究学者对此技术进行了一些卓有成效的研究,提出了一些具有启发性的方法与技术。总的来说,扩展查询词的选择通常有三种方式:第一种是根据语言学知识构建大规模的手工词典。如Dipasree等[3]提出一种基于WordNet定义的方法,使用词汇的定义扩展查询;Parapar等[4]使用WordNet中不同类型的语言信息扩展查询,但实验结果表明,并不是每次都有明显的效果,而且WordNet的使用在引入扩展词的过程中增加了大量与查询内容无关的查询词。第二种是基于大规模通用语料库信息统计的方法来选择扩展查询词。如王水利等[5]提出了利用基于互信息的共现模型分析初检文档,结合语义词典两方面选取扩展词对原查询进行扩展形成新的查询。这类查询扩展方法带来的噪声信息小,但是由于互信息或者共现和同义词的概念是不同的,因此达不到同义词扩展的目的,效果上一般没有明显的提高。第三种是基于Web的查询扩展。如Xu等[6]提出了一种基于维基百科的伪相关反馈方法,分别对查询对象为实体、查询对象具有歧义和其他类型的查询对象设计了不同的选择扩展词的方案。这类方法结合了前两种方法的优点,在开放领域的信息检索中有很好的效果;但在特定领域中,由于对维基百科语料库的依赖,上述方法不能全面地包含某些特定领域的词汇,导致查询扩展的效果不佳。更重要的是,以上三种方法均忽略了语义在查询扩展中的重要性,即尽可能保留原始查询的局部语义和全局语义,既要强调扩展词与查询词的同义性,又要强调扩展后的查询与原始查询的同义性。
文献[7-8]提出结合上下文的内容来学习词的语义向量表示,得到的向量在语义空间中具有一定的语义关联,即在语义空间中,相似或相关语义的词距离更近。针对专业领域来说,如生物医学领域,其检索过程涉及了大量专业词汇以及特殊的含义,而对查询词的扩展往往需要一个已有的词典或者扩展词来源,但这些仍然不能满足专业领域的知识需要,如“IL6,IL-6”是白细胞介素6的意思,但词典中可能不会含有这个词的英文简写,进而不能进一步寻找其相关词。又如“系统性红斑狼疮”一词,在同一文献中的形式是一致的,但在其他文献中可能还会出现“狼疮血管炎”“全身性红斑狼疮”等同义词,这些词可能不会全部出现在同义词库中,而且用户在查询时不会将同义词也逐一列出,同时使用共现等方式得到的扩展词仅是与该词有共现关系的词,并不一定是相关的词。因此,引入语义向量表示模型对专业词汇进行语义表示从而得到相似词作为扩展词是很有必要的。
1 基于上下文的语义向量表示近年来随着深度学习的发展,语义表示学习可以采用低维度连续向量来刻画词的语义,使得具有相似或相关语义的词向量在语义空间中距离比其他语义的词近。由于用户在进行查询时使用的词,在其相关的结果中可能以相近词的形式出现,故而可以这种方式来选择扩展查询词。因此,可以通过引入语义向量表示模型,针对专业领域相关文档(如生物医学文档)的词进行语义向量表示,通过相似度计算帮助选择专业领域查询词的扩展词,从而改善查询扩展的结果。本章将对基于上下文的语义向量表示方法进行介绍。
对于一个词来说,它所包含的语义应该由该词所在的语义环境,即该词周围的词的语义来决定,那么就是将一个词语义向量的训练过程转化成一个判断该词语义在其上下文语义中是否成立的过程。语义向量表示方法的目标是根据该语义单元的上下文语义,学习得到其语义表示,使得该语义单元在其上下文所形成的语言环境中具有最高的不可替代性。
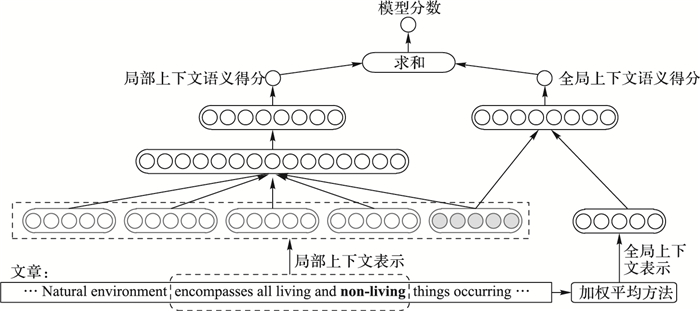
模型中的上下文包括局部上下文(s)和全局上下文(d)。其中局部上下文指词在文档中其周围的词,全局上下文指词所在文档。模型从文档中逐一取出每个词(中心词)之前的n个词作为其局部上下文,连同该中心词,联结其对应的向量成为模型局部上下文的正样本输入。词所在的段落作为全局上下文,使用平均加权的方式将段落中的所有词向量求平均,所得到的向量连同中心词向量一同作为模型全局样本输入。将中心词随机替换为其他词,其局部上下文保持不变,所组成的向量作为模型局部上下文的负样本输入,而替换的词与原有的全局上下文表示所组成的向量作为模型全局上下文的负样本输入。
如图 1所示,模型由两个神经网络构成,其输入为上下文正负样本的向量表示,输出均为一个数值。两个神经网络分别用于将每个中心词的局部上下文正负样本和全局上下文正负样本转化为分数。用于局部上下文计算的神经网络为四层,用于全局上下文计算的神经网络为三层。局部上下文计算过程为:
|
图 1 语义向量表示模型 |
| ${h_1} = \tanh \left( {{W_1} + {s_0} + {b_1}} \right)$ | (1) |
| ${h_2} = \tanh \left( {{W_2} + {h_1} + {b_2}} \right)$ | (2) |
| ${S_L}\left( s \right) = {W_3} \times {h_2} + {b_3}$ | (3) |
其中:s0为中心词及与其局部上下文的向量表示,h1、h2表示计算得到的隐藏层向量,W1、W2、W3、b1、b2、b3表示网络中的权值和偏置。全局上下文计算过程为:
| ${h_g} = \tanh \left( {{W_{g1}} \times {s_g} + {b_{g1}}} \right)$ | (4) |
| ${S_C}\left( {s,d} \right) = {W_{g2}} \times {h_g} + {b_{g2}}$ | (5) |
其中:sg为中心词与其全局上下文的向量表示,hg表示计算得到的隐藏层向量,Wg1、Wg2、bg1、bg2表示网络中的权值和偏置。将SL(s)和SG(s, d)相加,作为模型的最终输出,即中心词对应其上下文正样本的得分为Sp,负样本得分为Sn,表示该词在其局部上下文和全局上下文中符合语言环境的程度。那么,对于一个中心词来说,Sp值越高越好,而Sn值越低越好,本文将二者的差值定义为ΔS,表示该中心词在其上下文中不可替代的程度,即:
| $\Delta S = {S_{\text{p}}} - {S_n}$ | (6) |
本文设定一个阈值T,如果ΔS超过阈值,则表示该语义单元的向量表示已具有一定程度的不可替代性,那么模型的损失为0;反之,如果ΔS小于阈值,则表示该语义单元的向量表示的不可替代性不足,则为模型添加损失。即:
| $cos{t_{s,d}} = \left\{ \begin{gathered} 0,\;\;\;\;\;\;\Delta S > T \hfill \\ T - \Delta s,\Delta S \leqslant T \hfill \\ \end{gathered} \right.$ | (7) |
其中:s表示中心词的局部上下文,d表示中心词的全局上下文。因此该模型的损失函数表示为:
| $cost = \sum\limits_{s \in S} {\sum\limits_{d \in D} {\max \left( {\cos {t_{s,t}}} \right)} } $ | (8) |
其中:S表示所有正样本的集合,D表示所有文本自然段或文档的集合。cost表示所有样本中其语义单元向量表示不可替代性不足的程度之和,因此需要寻找最优的模型参数和语义单元向量表示使得cost值达到最小,即语义单元根据其上下文语境得到的向量能够清楚地表示其语义,然而具有明显的不可替代性。本文采用有限内存BFGS(Limited-memory BFGS, L-BFGS)方法优化函数作为参数的学习方法寻找模型的最优参数,使得损失函数值最小,从而得到生物医学词的最佳语义向量表示集合F。
2 扩展的新查询语句形成对用户提出的一条查询来说,查询中并不是所有的词语都与其目标文档相关,又由于每个用户对查询的表达方式各不相同,所以也并不是查询中所有的词语都可以直接匹配到相关文档的关键词语。因此,需要将一些内容相关的替换词,即查询扩展词,引入到原始的查询中。作为查询扩展的前提条件,本文首先基于上下文的语义单元向量表示模型,对专业领域语料库中所涉及到的所有词进行训练,得到所有词的语义向量表示F,然后文本通过计算两个词向量Fi={fi1, fi2, …, fim}和Fj={fj1, fj2, …, fjm}的相似度来衡量两个词的相似度,计算公式为:
| $si{m_{ij}} = \cos \left\langle {{F_i},{F_j}} \right\rangle = \frac{{\sum\limits_{k = 1}^m {{f_{ik}}{f_{jk}}} }}{{\sqrt {\sum\limits_{k = 1}^m {f_{ik}^2} } \sqrt {\sum\limits_{k = 1}^m {f_{jk}^2} } }}$ | (9) |
其中: fip表示向量Fi中第k个数值,m表示向量维数。通过计算一个查询词与其余所有词的相似度,本文取出其中相似度最高的n个词作为扩展查询词。
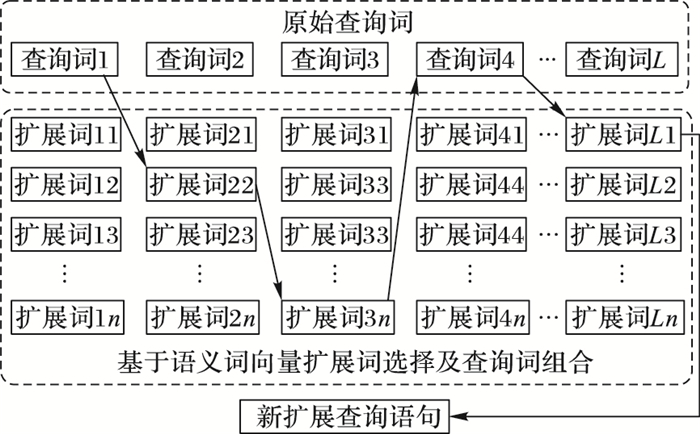
查询语句扩展示意图如图 2所示,在给定一个查询语句时,经过停用词处理后,得到一个包含关键原始查询词的序列,即:
|
图 2 查询语句扩展示意图 |
| $Q = \left( {{q_1},{q_2}, \cdots ,{q_i}, \cdots ,{q_L}} \right)$ | (10) |
其中:qi表示序列中依次出现的关键原始查询词,并且有一个语义向量与之对应。通过式(9)计算每一个原始查询词与词典中其余所有词的相似度,取出相似度最高的n个语义向量对应的词。本文将这n个词作为qi的查询扩展词集合:
| ${P^{\left( i \right)}} = \left\{ {p_1^{\left( i \right)},p_3^{\left( i \right)},p_3^{\left( i \right)}, \cdots ,p_n^{\left( i \right)}} \right\}$ | (11) |
其中:pn(i)表示根据原始查询词qi的语义所扩展得到的与其语义相近的第n个查询扩展词,集合P(i)中的每一个查询扩展词都可以作为原始查询词qi的替换词。那么原始查询语句中的词则可以由式(12)中的任意一项替换,即为原始查询语句中第i个词可被替换的所有可能,替换后的语句如式(13)所示为扩展后的新语:
| ${Q^{\left( i \right)}} = \left\{ {{q_i}} \right\} \cup {P^{\left( i \right)}}$ | (12) |
| ${Q_{{\text{new}}}} = \left( {{Q^{\left( 1 \right)}},{Q^{\left( 2 \right)}},{Q^{\left( 3 \right)}}, \cdots ,{Q^{\left( L \right)}}} \right)$ | (13) |
综上所述,从原始查询语句Q依次通过式(11)~(13)得到Qnew的过程即为基于语义向量表示的查询语句扩展过程。由此可知,新形成的查询语句会有多种不同表达方式,从而扩展了原始查询语句的多样性。
理论上来说,相比其他查询扩展方法,本文提出的查询扩展方法具有两个明显的优势:一方面,基于语义向量的查询扩展方法利用了基于上下文的词向量表示,在训练的过程中,体现了扩展词与查询词在语料库中的可替代性,这种可替代性在查询中可以同样得到体现;另一方面,在获得语义向量后,查询语句形成过程中,本文提出的方法保留了查询的原始语序,在最大限度上保留了原始查询的语义,降低查询扩展过程带来的无关信息的影响。
3 生物医学文档检索系统及实验分析以生物医学文档检索为例,将基于生物医学词汇的语义向量表示引入到查询扩展当中,设计一个生物医学文档检索系统; 然后,将结果分别与传统查询扩展方法和BioASQ生物医学竞赛中其他信息检索的方法对比,通过对比验证本文提出的查询扩展方法既优于传统的查询扩展方法,又具有足够的实际应用价值。
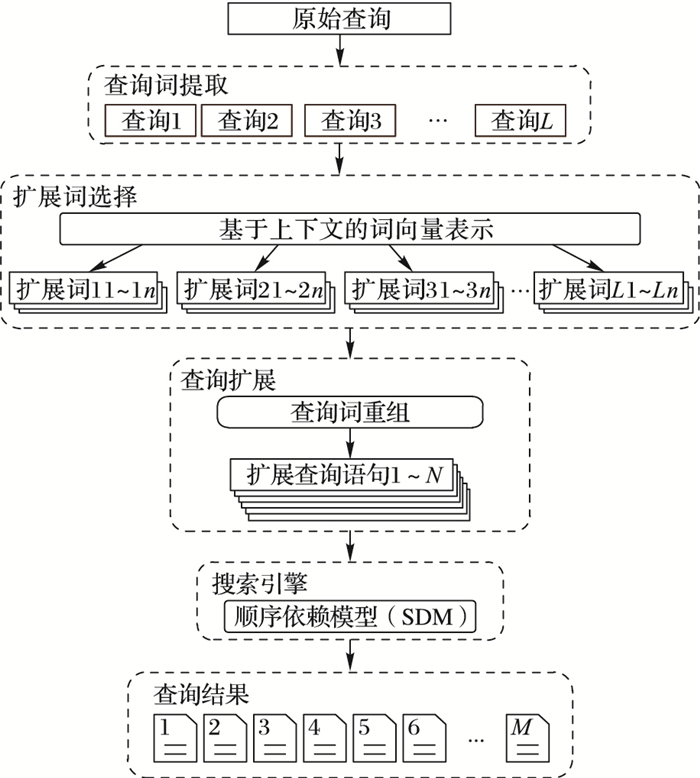
3.1 生物医学文档检索系统如图 3所示,为本文设计的一个生物医学文档检索系统,该系统主要有以下四部分组成:
|
图 3 生物医学文档检索系统 |
1) 原始查询词提取。给定一个查询语句,使用一个通用的停用词表对原始查询中的词进行过滤,去掉常用词以及不起作用的词,仅保留有实体意义的查询词。
2) 扩展查询词选择。使用语义单元向量模型对生物医学领域的词汇进行语义向量表示。针对每个原始查询词与其他词计算相似度,从中找出与之相似度最接近的n个词,作为扩展查询词。
3) 查询语句扩展。使用原始查询中对应的扩展查询词在原始查询语句中进行一定几率的替换,将新生成的查询词序列作为扩展查询语句。
4) 使用检索工具查询。使用Galago作为检索工具进行生物医学文档检索。在检索时,使用顺序依赖模型(Sequence Dependence Model, SDM)来强化检索的方式,以便提高查询结果的准确率。最后,Galago返回一个文档列表,该列表中的文档按照与查询语句的相关程度排序。
3.2 BioASQ数据集及实验设置本实验使用来自2014年和2015年BioASQ生物医学竞赛阶段A生物医学语义问答任务的官方真实数据以及从MEDLINE数据库中获取的生物医学文档集作为检索库。阶段A的数据由专家提出的相关专业问题组成,数据集共发布了1个训练集和5个竞赛的测试集,其中训练集包含300个问题,每个测试集包含100个问题。文档集包含了从建库之日起到2013年间的2 200万篇生物医学相关文章的标题和摘要。该阶段任务是根据给定的问题,从检索库中找出与问题答案最相关的100篇文章,测评的指标是平均准确率(Mean Average Precision, MAP)。
本文从MEDLINE数据库中选择120万篇文章的标题和摘要作为生物医学领域词向量的训练数据,文章涵盖了从1948年以来出现的生物医学相关的词,这些词的语义最初由随机初始的向量来表示。本文采用语义向量表示模型进行语义表示训练,通过出现频率筛选,得到了48 361个主要词的向量表示。
3.3 扩展查询词数量选择实验本文将每一个原始查询词对应的扩展查询词数量缩小到从1到10的范围。为了进一步确定扩展查询词选取的数量,依次使用不同数量的扩展查询词在训练集和测试集进行实验,并对最终的MAP评价结果进行了比较,如表 1所示。从结果中可以看出,当扩展查询词的数量取3时,该方法在训练集和测试集上的表现最好。
| 表 1 使用不同数量查询扩展词的结果对比(MAP) |
在本实验中,将本文查询扩展方法(OurM)与传统方法进行对比,对比实验方法及设置如下:
Wikipedia(Wiki):Wikipedia(维基百科)是一个免费的百科网站,如果原始查询语句中的词与网站中的实体页关联,则将实体页中的关键词作为查询扩展词。
WordNet(WN):根据每个原始查询词在WordNet中进行查询,将该词对应的相关词作为查询扩展词。
实验中,本文将Galago的参数设置为2 000,使用TRAC_EVAL作为评价工具来计算MAP值,结果如表 2所示。
| 表 2 使用不同扩展方法结果对比(MAP) |
从表 2的结果可以看出,基于维基百科和WordNet的两种查询扩展方法的检索结果相当,而使用生物医学语义词向量得到的检索结果优于传统的查询扩展方法,MAP值分别最多提高了5.6%和6.0%。原因在于一些专业词汇或者其特殊含义不能在维基百科知识库和WordNet同义词表中找到,因此使得扩展词的语义有所偏差。
3.5 BioASQ实际评测结果使用文本所提出的生物医学文档检索系统(OurS)与BioASQ测评竞赛的参赛队伍成绩进行比较。表 3为2014年BioASQ参赛队伍的成绩,表中包含参加了至少两组测试的队伍。其中,SNUMedinfo团队使用了UMLS生物医学专有名词集对查询进行了扩展[9];UMass团队使用了文档中的图片,对图片内容进行识别,并把识别出的文本作为关键信息扩展到查询中[10];BTM是由美国美国国家生物技术信息中心通过其拥有的大量的生物医学资源进行对于查询和文档同时扩展,通过检索得到的结果[11];main system是由BioASQ官方通过PubMed搜索引擎在文档集上检索的结果,作为评测的基准;Wishart则是采取了支持向量机(Support Vector Machine, SVM)等分类模型,判断某一个文档是否与查询有关;而All-Figdoc和HPI团队,在技术报告中缺乏对方法的描述,因此只有官方的比赛成绩[12]。这些方法中,和扩展相关的方法大都依赖于额外的生物医学数据集或名词集,而且扩展的过程主要是基于词频或基于名词在集合中的共现概率,而忽略了该名词本身的语义。通过几组实验结果可以看出这些方法并不适合。
| 表 3 本文方法与BioASQ2014参赛队伍成绩(MAP) |
为了进一步验证系统的有效性,本文针对表中每一组参赛队伍的结果进行了显著性差异的计算[13]。如表 4所示,经逐一计算可知,所有概率p均小于0.05,同时结合效应值(Effect Size, ES)和95%置信区间(Confidence Interval, CI)的结果对比,说明文本系统的检索结果与其他队伍相比有显著的提高。
| 表 4 本文方法与BioASQ2014参赛队伍成绩的显著性差异比较 |
将本文生物医学文档检索系统应用于2015年的BioASQ测评比赛,表 5所示为官方给出的仅前2名的比赛成绩[14],其中本文队伍(USTB)在第3和第4测试组别中排名第一,在第1和第5测试组别中排名第二,该成绩也验证了本文提出的基于语义向量表示的查询扩展方法在生物医学文档检索系统中的有效性。
| 表 5 BioASQ2015官方成绩排名 |
相比其他方法,本文的方法重点关注了查询词本身的语义。通过语义向量表示的方法,确保查询扩展过程中添加到查询中的扩展词与原查询词语义相同或者相近,从而保证了查询扩展的质量。
4 结语本文针对查询扩展中扩展词与原始查询之间缺乏语义关联的问题,提出了一种基于语义向量表示的查询扩展方法,并设计了一个生物医学文档检索系统。以生物医学领域为例,训练了生物医学词汇的语义向量,通过计算相似度的方法选取语义相近的词,并用于扩展查询语句。最后,在与基于维基百科或WordNet的传统查询扩展方法和BioASQ 2014参加竞赛的系统的实验对比中,基于语义向量表示的查询扩展方法在检索结果上优于传统的查询扩展方法。生物医学文档检索系统在BioASQ 2014年测试中,检索结果均优于参赛各队伍成绩,并且均有显著性提高(p < 0.5);在BioASQ 2015年的比赛中,生物医学文档检索系统取得了竞赛中第一名两次和第二名两次。实验结果表明,基于语义向量表示的查询扩展方法是有效的。
本文方法仍有可改进和优化的空间:1)可以将基于语义向量表示的查询扩展方法应用更多的特殊领域信息检索;2)可以将语义向量表示模型应用与信息检索的其他研究方向。
| [1] |
XU J, CROFT W B. Query expansion using local and global document analysis [C]// SIGIR '96: Proceedings of the 19th Annual International ACM SIGIR Conference on Research and Development in Information Retrieval. New York: ACM, 1996: 4-11.
( 0) 0)
|
| [2] |
VOORHEES E M. Query expansion using lexical-semantic relations [C]// SIGIR '94: Proceedings of the 17th Annual International ACM SIGIR Conference on Research and Development in Information Retrieval. Berlin: Springer, 1994: 61-69.
( 0) 0)
|
| [3] |
DIPASREE P, MAR M, KALYANKUMAR D. Improving query expansion using WordNet[J].
Journal of the Association for Information Science and Technology, 2013, 65 (12) : 2469-2478.
( 0) 0)
|
| [4] |
PARAPAR D, BARREIRO A, LOSADA D E. Query expansion using WordNet with a logical model of information retrieval [EB/OL]. [2015-11-23]. http://www-gsi.dec.usc.es/~dlosada/iadis05.pdf.
( 0) 0)
|
| [5] |
王水利, 黄广君, 霍亚格. 基于语义分析的查询扩展方法[J].
计算机工程, 2011, 37 (16) : 77-79.
( WANG S L, HUANG G J, HUO Y G. Query expansion method based on semantic analysis[J].
Computer Engineering, 2011, 37 (16) : 77-79.
) ( 0) 0)
|
| [6] |
XU Y, JONES G J F, WANG B. Query dependent pseudo-relevance feedback based on wikipedia [C]// SIGIR '09: Proceedings of the 32nd International ACM SIGIR Conference on Research and Development in Information Retrieval. New York: ACM, 2009: 59-66.
( 0) 0)
|
| [7] |
HUANG E H, SOCHER R, MANNING C D, et al. Improving word representations via global context and multiple word prototypes [C]// ACL '12: Proceedings of the 50th Annual Meeting of the Association for Computational Linguistics. Stroudsburg, PA: Association for Computational Linguistics, 2012, 1: 873-882.
( 0) 0)
|
| [8] |
LI Y, ZHANG Y, HUANG X, et al. Chinese word segmentation with local and global context representation learning[J].
High Technology Letters, 2015, 21 (1) : 71-77.
( 0) 0)
|
| [9] |
CHOI S, CHOI J. Classification and retrieval of biomedical literatures: SNUMedinfo at CLEF QA track BioASQ 2014 [EB/OL]. [2016-01-02]. http://ceur-ws.org/Vol-1180/CLEF2014wn-QA-ChoiEt2014.pdf.
( 0) 0)
|
| [10] |
JESSE L, LAURA D. UMass at BioASQ 2014: figure-inspired text retrieval [C]// CLEF: Proceedings of the 2014 Conference and Labs of the Evaluation Forum (Working Notes). Berlin: Springer, 2014: 1296-1310.
( 0) 0)
|
| [11] |
MAO Y, WEI C H, LU Z. NCBI at the 2014 BioASQ challenge task: large-scale biomedical semantic indexing and question answering [C]// CLEF: Proceedings of the 2014 Conference and Labs of the Evaluation Forum (Working Notes). Berlin: Springer, 2014: 1319-1327.
( 0) 0)
|
| [12] |
GEORGIOS B, IOANNIS P, AXEL-CYRILLE N N, et al. Results of the BioASQ track of the question answering lab at CLEF 2014 [C]// CLEF: Proceedings of the 2014 Conference and Labs of the Evaluation Forum (Working Notes). Berlin: Springer, 2014: 1181-1193.
( 0) 0)
|
| [13] |
SAKAI T. Statistical reform in information retrieval?[J].
ACM SIGIR Forum, 2014, 48 (1) : 3-12.
doi: 10.1145/2641383 ( 0) 0)
|
| [14] |
TSATSARONIS G, BALIKAS G, MALAKASIOTIS P, et al. An overview of the BIOASQ large-scale biomedical semantic indexing and question answering competition[J].
BMC Bioinformatics, 2015, 16 (1) : 1.
doi: 10.1186/s12859-014-0430-y ( 0) 0)
|




