用计算机分析识别肿瘤细胞图像对降低医生的劳动强度和提高诊断精度都具有十分重要的意义,也是人工智能领域一个活跃的分支,肿瘤细胞图像识别研究近年来一直受到众多学者的关注。在肿瘤细胞图像分类识别方面,流行的识别方法有稀疏表示、支持向量机(Support Vector Machine,SVM)、神经网络以及深度学习。
稀疏编码作为一种有效理解人类神经系统信息加工机制的理论工具,已广泛地应用于图像特征提取[1]、图像去噪[2],以及人脸识别[3]等方面。孙俊等[4]等利用稀疏编码来提取人脸图像的整体特征,并引证了利用稀疏编码提取的特征具有更好的分类特性。有的学者将其应用于彩色肝细胞图像识别[5]和脑脊液细胞图像快速分类[6],取得了较好的识别效果。对于一幅胃上皮肿瘤显微细胞图像(以下简称肿瘤细胞图像),其细胞图像具有稀疏响应的特性,所以也可以利用稀疏编码进行特征提取和模式识别。
许多学者对于肿瘤细胞图像识别进行了深入的研究[7-8],但是由于肿瘤细胞图像存在组织器官形状不规则、不同类别的细胞差异性较大且大量细胞核聚集粘连等问题,所以提取肿瘤细胞图像的局部特征是非常困难的。针对这个问题,可以采用稀疏编码来提取肿瘤细胞图像的全局特征来解决。然而,利用稀疏表示方法对肿瘤细胞图像进行模式分类时注意到以下两方面的问题:
一是以往的稀疏表示模型忽略了图像的类别信息,如果将Fisher准则引入稀疏表示模型,使得字典和稀疏系数都具有判别性,可以提高模式分类性能。
二是肿瘤细胞图像在采集成像过程中可能会受到噪声的影响,这种情况下,图像的编码误差不满足高斯分布。在传统的稀疏编码框架下,利用 L2 范数来度量编码误差,这也就意味着编码误差必须服从高斯分布或拉普拉斯分布,因此传统的稀疏表示模型不能够有效描述肿瘤细胞图像实际的编码残差。同时,编码系数的稀疏约束使得传统稀疏编码的计算量非常大,而正则化鲁棒稀疏表示(Regularized Robust Coding,RRC)模型[9] 可以很好地解决这些问题。
基于上述分析,本文提出了一种基于Fisher判别字典学习(Fisher Discrimination Dictionary Learning,FDDL)[10]的RRC肿瘤细胞图像的识别方法——FDDL-RRC。首先利用FDDL得到肿瘤细胞图像的全局特征基即字典,然后将字典用于RRC模型实现对肿瘤细胞图像测试样本的分类。实验结果表明本文方法对于肿瘤细胞图像具有良好的分类效果及较快的分类速度。
1 稀疏表示分类的原理稀疏编码模型成功地模拟了哺乳动物主视皮层V1区简单细胞对外界视觉刺激图像的处理过程,自然图像可由基函数的线性叠加来描述,如式(1)所示:
| $Y=SX$ | (1) |
其中:N维随机向量Y=[Y1,Y2,…,YN]T表示输入图像;N×M维矩阵S=[S1,S2,…,SM]表示图像集合的特征基函数空间,S满足超完备特性,即N>M;M维向量X=[X1,X2,…,XM]T则表示输入图像Y在特征基函数空间S上的编码系数,X满足稀疏性。
基于稀疏表示的分类(Sparse Representation based Classification,SRC)方法[11]将测试样本表示为来自同一类别的训练样本的线性组合。假设样本有K类,训练样本矩阵为A=[A1,A2,…,Ai,…,AK],Ai是第i类的训练样本子矩阵。令y表示测试样本,则稀疏表示分类的过程如下:
1) 通过L1范数最小化求解y在A上的稀疏表示:
| $\dot{\alpha }=\arg {{\min }_{\alpha }}\left\{ \left\| y-A\alpha \right\|_{2}^{2}+\gamma {{\left\| \alpha \right\|}_{1}} \right\}$ | (2) |
其中:γ为参数。
2) 计算残差:
| ${{e}_{i}}\left( y \right)={{\left\| y-{{A}_{i}}{{{\dot{\alpha }}}_{i}} \right\|}_{2}}$ | (3) |
其中:$\dot{\alpha }=\left[ {{{\dot{\alpha }}}_{1}};{{{\dot{\alpha }}}_{\text{2}}};\cdots ;{{{\dot{\alpha }}}_{i}};\ldots ;{{{\dot{\alpha }}}_{K}} \right]$是与类别i相关联的编码系数向量。
3) 输出y的类别:
| $identity\left( y \right)=\arg {{\min }_{i}}\left\{ {{e}_{i}}\left( y \right) \right\}$ | (4) |
具有最小重构误差的那一类就是测试样本y所属的类别。
2 字典学习与分类模型对于自然图像,基于SRC[11]有两个阶段:编码和分类。首先,图像被具有稀疏约束的字典原子协同编码;然后根据编码系数和字典实现分类。肿瘤细胞图像作为一种特殊的自然图像,具有稀疏性,因此也可以采用稀疏表示分类的方法进行分类。
2.1 FDDL为了得到一个性能更好的字典,FDDL方法[10]学习得到的不是共享字典,而是结构化的字典。本文将经过预处理后的肿瘤细胞图像的特征矩阵作为初始字典进行学习。假设A=[A1,A2,…,Ai,…,AK]表示K类训练样本的特征矩阵,令字典学习后得到的新字典为D,即A≈DX。其中:D=[D1,D2,…,Di,…,DK],Di是第i类的子字典;X为A在字典D上的编码系数矩阵,记作X=[X1,X2,…,Xi,…,XK],Xi是Ai在字典D上编码系数的子矩阵。字典D除了对A具有较强的重构能力外,还希望对A的图像具有较强的区分能力,所以,定义Fisher判别字典模型如下:
| ${{J}_{\left( D,X \right)}}=\underset{\left( D,X \right)}{\mathop{\arg \min }}\,\left\{ r\left( A,D,X \right)+{{\lambda }_{1}}{{\left\| X \right\|}_{1}}+{{\lambda }_{2}}f\left( X \right) \right\}$ | (5) |
假设Ai在字典D上的表示为Xi=[X1i;X2i;…;Xji;…;XKi],Xji是Ai在子字典Dj上的编码系数。由于某一类别的特征若被其他类的子字典较好地表示的话,会降低字典的表示能力。为了增强字典对同类特征的表示能力,将判别保真项定义为:
| $r\left( {{A}_{i}},D,{{X}_{i}} \right)=\left\| {{A}_{i}}-D{{X}_{i}} \right\|_{F}^{2}+\left\| {{A}_{i}}-{{D}_{i}}X_{i}^{i} \right\|_{F}^{2}+{{\sum\limits_{\begin{smallmatrix} j=1 \\ j\ne i \end{smallmatrix}}^{K}{\left\| {{D}_{j}}X_{i}^{j} \right\|}}^{2}}$ | (6) |
首先,字典D应该能够很好地表示Ai,因此有Ai≈DXi=D1X1i+D2X2i+…+DiXii+…+DKXKi;其次,为了强化子字典Di对与其相关联的Ai的表示能力,弱化其他子字典Dj(j≠i)对Ai的表示能力,应赋予Xii较大的系数使得‖Ai-DiXii‖2F的值较小,而Xji的系数应近似为0才能使‖DjXji‖2F的值很小。
为了使字典D对样本A具有判别性,可以使A在字典D上的编码系数具有判别性。基于Fisher判别准则,这种判别性可以通过最小化X的类内散度SW(X)和最大化X的类间散度SB(X)来实现。SW(X)和SB(X)的定义如下:
| $\eqalign{ & \sum\limits_{i = 1}^K {\sum\limits_{{x_c} \in {X_i}} {\left( {{x_c} - {m_i}} \right)} } {\left( {{x_c} - {m_i}} \right)^T} \cr & {S_B}\left( X \right) = \sum\limits_{i = 1}^K {{n_i}} \left( {{m_i} - m} \right){\left( {{m_i} - m} \right)^T} \cr} $ |
其中:mi和m分别是Xi和X的均值向量,ni是类别Ai的样本个数。为了保证判别约束项f(X)是凸函数,在f(X)中添加松弛项‖X‖2F,故f(X)定义如下:
| $f\left( X \right)=tr\left( {{S}_{W}}\left( X \right) \right)-tr\left( {{S}_{B}}\left( X \right) \right)+\eta \left\| X \right\|_{F}^{2}$ | (7) |
将式(6)和式(7)代入式(5)中可得最终FDDL的目标函数:
| ${{J}_{\left( D,X \right)}}=\underset{\left( D,X \right)}{\mathop{\arg \min }}\,\left\{ \begin{align} & \sum\limits_{i=1}^{K}{r\left( {{A}_{i}},D,{{X}_{i}} \right)+{{\lambda }_{1}}{{\left\| X \right\|}_{1}}+} \\ & {{\lambda }_{2}}\left( tr\left( {{S}_{W}}\left( X \right)-{{S}_{B}}\left( X \right) \right)+\eta \left\| X \right\|_{F}^{2} \right) \\ \end{align} \right\}$ | (8) |
其中:r(A,D,X)是判别保真项;‖X‖1是稀疏约束项;tr(SW(X)-SB(X))+η‖X‖2F是施加在稀疏系数X上的判别约束项;λ1和λ2为平衡因子。该目标函数通过分别固定D和X迭代优化来求解判别字典D和判别系数X。
2.2 RRC模型当图像出现遮挡和噪声污染时,传统的稀疏编码模型学习到的字典是有噪声的。RRC模型[9]提高了传统稀疏表示模型对于图像受到光照、遮挡或其他因素等干扰和污染的鲁棒性和有效性。
首先将训练样本的字典改写为D=[d1;d2;…;di;…;dn],di是D的第i行。在字典D上对测试样本y进行编码,编码向量α的最大后验概率估计为:
由贝叶斯公式可得:
| $\dot{\alpha }=\arg {{\max }_{\alpha }}\left\{ \ln P\left( y\left| \alpha \right. \right)+\ln P\left( \alpha \right) \right\}$ | (9) |
假设编码残差e= y-Dα=[e1;e2;…;ei;…;en]中的元素ei是独立同分布的;同时,编码向量α=[α1;α2;…;αj;…;αm]的元素αj也是独立同分布的,α的概率密度函数为fο(αj),则有P(α)=∏mj=1fο(αj),式(9)中α的最大后验估计变为:
| $\dot{\alpha }=\arg {{\max }_{\alpha }}\left\{ \underset{i=1}{\overset{n}{\mathop{\prod }}}\,{{f}_{\theta }}\left( {{y}_{i}}-{{d}_{i}}\alpha \right)+\underset{j=1}{\overset{m}{\mathop{\prod }}}\,{{f}_{o}}\left( {{\alpha }_{j}} \right) \right\}$ | (10) |
令ρθ(e)=-ln fθ(e),ρο(α)=-ln fο(α),式(10)转化为:
| $\dot{\alpha }=\arg {{\max }_{\alpha }}\left\{ \sum\limits_{i=1}^{n}{{{\rho }_{\theta }}\left( {{y}_{i}}-{{d}_{i}}\alpha \right)+\sum\limits_{j=1}^{m}{{{\rho }_{o}}\left( {{\alpha }_{j}} \right)}} \right\}$ | (11) |
由于保真项$\sum\limits_{i=1}^{n}{{{\rho }_{\theta }}\left( {{y}_{i}}-{{d}_{i}}\alpha \right)}$对异常像素点具有鲁棒性,而$\sum\limits_{j=1}^{m}{{{\rho }_{o}}}\left( {{\alpha }_{j}} \right)$则是取决于先验概率P(α)的正则化项,因此,式(11)被称作RRC模型。
式(11)又可进一步转化为:
| $\dot{\alpha }=\arg {{\min }_{\alpha }}\left\{ \frac{1}{2}\left\| {{W}^{\frac{1}{2}}}\left( y-D\alpha \right) \right\|_{2}^{2}+\sum\limits_{j=1}^{m}{{{\rho }_{o}}\left( {{\alpha }_{j}} \right)} \right\}$ | (12) |
式(12)是式(11) 的局部逼近,它通过迭代正则化的加权L2范数求解模型最小值,把传统的稀疏表示模型转化成加权的正则化稀疏表示模型。W是对角权值矩阵,元素Wi,i即ωθ(ei)是分配给测试图像y的像素点i的权值。权值系数的确定是RRC模型的关键,为了减少异常像素点对编码的影响,对于被遮挡或噪声干扰的像素点赋予较小的权重,而其他像素点的权重相对较大。不失一般性,令ωθ(ei)∈[0,1],选择logistic函数来计算权值:
| ${{\omega }_{\theta }}\left( {{e}_{i}} \right)=\frac{\exp \left( -\mu e_{i}^{2}+\mu \delta \right)}{\left( 1+\exp \left( -\mu e_{i}^{2}+\mu \delta \right) \right)}$ | (13) |
其中: μ和δ是正参数,μ控制权值从1到0的下降速度,δ控制分界点的位置。通过不断迭代优化得到最终的权值矩阵Wfinal后,可求得最优的稀疏系数,然后根据最小逼近残差准则输出测试图像的所属类别。
3 本文FDDL-RRC方法 3.1 主要思想为了提高肿瘤细胞图像稀疏表示的判别性,同时考虑到肿瘤细胞图像在采集过程中不可避免地会受到污染,所以本文方法的主要思想为:首先,肿瘤细胞图像的训练样本通过FDDL得到结构化的字典和投影到字典上的系数,字典就是图像的全局特征,系数就是特征描述子。由于对编码系数施加了Fisher判别约束,使得编码系数具有最小的类内散度和最大的类间散度,所以得到的特征字典具有很强的区分能力。然后,将特征字典用于RRC模型对测试样本进行分类。RRC模型将稀疏表示的保真度表示为余项的最大后验估计函数,从而将识别问题转化为正则化加权范数的优化逼近问题。最后,通过最小残差逼近准则得到测试样本的类别。
根据上述分析,本文将FDDL和RRC两种方法有效结合起来,提出了FDDL-RRC肿瘤细胞图像的识别方法。肿瘤细胞图像经过预处理后,采用FDDL生成图像的特征字典,将RRC模型作为分类策略就可以实现肿瘤细胞图像的分类。本文方法可分为训练和测试两个阶段。
在训练阶段,首先从训练图像样本库集随机地选取图像,然后对图像进行预处理(4.1节),假设训练样本矩阵为Ft=[Ft1,Ft2,…,Fti,…,FtN],N为训练样本的总数,Fti为训练样本矩阵Ft中第i(i∈[1,N])个样本的数据;Ft为初始特征集,利用上述的识别算法生成训练图像的特征字典Dt=[Dt1,Dt2,…,Dti,…,DtK],K为样本类别数。
在测试阶段,对测试图像进行预处理后,可以得到测试图像的样本矩阵Fc=[Fc1,Fc2,…,Fcj,…,FcM],M为测试样本的总个数。将Fc和学习得到的训练样本的特征字典Dt用于RRC模型中,通过求解
| $\dot{\alpha }=\arg {{\min }_{\alpha }}\left\{ \frac{1}{2}\left\| {{W}^{\frac{1}{2}}}\left( {{F}_{c}}-{{D}_{t}}\alpha \right) \right\|_{2}^{2}+\sum\limits_{j=1}^{M}{{{\rho }_{o}}\left( {{\alpha }_{j}} \right)} \right\}$ |
得到最优的稀疏系数矩阵,最后根据最小残差逼近准则来实现对测试图像的分类:
| $identity\left( {{F}_{c}} \right)=\arg {{\min }_{j}}\left\{ {{\left\| {{F}_{cj}}-{{D}_{tj}}{{\alpha }_{j}} \right\|}_{2}} \right\},j=1,2,\ldots ,K$ |
本文选取了N张肿瘤细胞图像作为训练样本,记为Ft(Ft=[Ft1,Ft2,…,Fti,…,FtN]);M张肿瘤细胞图像作为测试样本,记为Fc(Fc=[Fc1,Fc2,…,Fcj,…,FcM]);训练样本和测试样本均为K类。在2.3节中所阐述的肿瘤细胞图像识别方法的理论基础上,将经过预处理后的训练样本矩阵Ft作为初始字典进行学习。利用FDDL方法学习得到的训练样本的特征字典记为Dt,故Ft≈DtX,Dt=[Dt1,Dt2,…,Dti,…,DtK},Dti是类别i的子字典;X为Ft在Dt上的编码系数,X=[X1,X2,…,Xj,…,XK],Xj为类别j的系数矩阵的子矩阵。本文方法的流程如图 1所示。
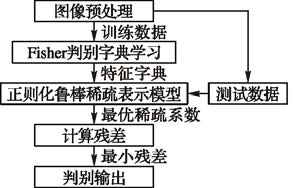
|
图 1 肿瘤细胞图像识别算法流程 |
本文方法的具体实现步骤如下:
第1步 首先对训练样本和测试样本进行灰度化处理并对训练样本矩阵和测试样本矩阵分别按列进行L2范数归一化操作,得到训练数据和测试数据;然后初始化字典Dt,Dti中的每一个原子是经过L2范数规范化的随机向量。
第2步 更新稀疏编码系数X:固定Dt,根据式(8)求解Xj( j=1,2,…,K)。
第3步 更新字典Dt:固定X,根据式(8)求解Dti(i=1,2,…,K)。
第4步 返回第2步,直到在相邻的迭代中J(D,X)的值足够接近或者迭代次数达到最大值时停止,输出Dt和X。
第5步 初始化稀疏系数${{\alpha }^{\left( 1 \right)}}=\left[ \frac{1}{M};\frac{1}{M};\ldots ;\frac{1}{M} \right]$。
第6步 计算残差e(Time)=Fc-Dtα(Time),Time初值为1。此处的Dt由第4步得到。
第7步 在每次迭代过程中可以得到μ和δ的值,计算权值:
| ${{\omega }_{\theta }}\left( e_{i}^{\left( Time \right)} \right)=\frac{1}{1+\exp \left( \mu {{\left( e_{i}^{\left( Time \right)} \right)}^{2}}-\mu \delta \right)}$ |
第8步 对测试样本Fc进行加权正则化鲁棒稀疏编码:
| ${{\alpha }^{*}}=\arg {{\min }_{\alpha }}\left\{ \begin{align} & \frac{1}{2}\left\| {{\left( {{W}^{\left( Time \right)}} \right)}^{\frac{1}{2}}}\left( {{F}_{c}}-{{D}_{t}}\alpha \right) \right\|_{2}^{2}+ \\ & \sum\limits_{j=1}^{M}{{{\rho }_{o}}\left( {{\alpha }_{j}} \right)} \\ \end{align} \right\}$ |
其中:W(Time)是对角权值矩阵,即W(Time)i,i=ωθ(e(Time)i); ρο(αj)=λαj2+b,λ和b均为常量。
第9步 更新稀疏编码系数:若Time=1,则α(Time)=α*;若Time>1,则α(Time)=α(Time-1)+v(Time)(α*-α(Time-1))。其中v(Time)是区间[0,1]内合适的步长,使得
| $\begin{align} & \sum\limits_{i=1}^{N}{{{\rho }_{\theta }}\left( {{F}_{c}}-{{D}_{ti}}{{\alpha }^{\left( Time \right)}} \right)}+\sum\limits_{j=1}^{M}{{{\rho }_{o}}}\left( \alpha _{j}^{\left( Time \right)} \right) \\ & <\sum\limits_{i=1}^{N}{{{\rho }_{\theta }}}*\left( {{F}_{c}}-{{D}_{ti}}{{\alpha }^{\left( Time-1 \right)}} \right)+\sum\limits_{j=1}^{M}{{{\rho }_{o}}}\left( \alpha _{j}^{\left( Time-1 \right)} \right) \\ \end{align}$ |
第10步 计算测试样本的重构误差:F(Time)crec=Dtα(Time),令Time=Time+1。
第11步 返回第6步,直到满足以下条件或者迭代次数达到最大值时停止优化:
| $\frac{{{\left\| {{W}^{\left( Time-1 \right)}}-{{W}^{\left( Time \right)}} \right\|}_{2}}}{{{\left\| {{W}^{\left( Time \right)}} \right\|}_{2}}}<{{\sigma }_{W}}$ |
其中:σW是一个极小的正参数。
第12步 利用式$identity\left( {{F}_{c}} \right)=\arg {{\min }_{i}}\left\{ {{\ell }_{i}} \right\}$对测试样本进行分类,其中:${{\ell }_{i}}={{\left\| W_{final}^{\frac{1}{2}}\left( {{F}_{c}}-{{D}_{ti}}{{{\dot{\alpha }}}_{i}} \right) \right\|}_{2}}$,Dti和${{{\dot{\alpha }}}_{i}}$分别是与类别i(i=1,2,…,K)相关联的子字典和子编码向量,Wfinal是最终的权值矩阵。
4 实验结果与分析胃黏膜肿瘤细胞图像总共可分为正常、增生和癌变三大类,实验中将这三类的标签依次设为1、2、3。实验分别选取各50幅正常、增生和癌变且大小为320×240的肿瘤细胞图像作为训练样本,另选取三类各20幅相同维度的图像作为测试样本。
4.1 数据预处理原始的肿瘤细胞图像是经过染色的彩色图像,图像的维度非常高。在数字图像处理中,一般先将图像转化为灰度图像以减少后续的图像的计算量。灰度图像的描述仍然反映了整幅图像的整体和局部的色度以及色度、亮度等级的分布和特征。图像的灰度化处理有两种实现方法:1)求出每个像素点的R、G、B三个分量的平均值,然后将这个平均值赋予这个像素点的三个分量;2)根据YUV的颜色空间中, Y 的分量的物理意义是点的亮度,由该值反映亮度等级,根据RGB和YUV颜色空间的变化关系可建立亮度 Y与R、G、B三个颜色分量的对应:Y=0.3R+0.59G+0.11B, 用这个亮度值来表达图像的灰度值。方法1)是对RGB三个分量取简单的平均,从而得到对应的灰度值;方法2)是依据人眼对不同颜色的敏感度不同,对RGB分量以不同系数的加权平均,得到较为合理的灰度化结果。经过实验比对,本文选取第二种灰度化方法对肿瘤细胞图像进行预处理效果比较好。
预白化处理能够增强图像边缘和线段等有效高频信息,同时降低噪声有效高频信息的干扰。在前面的工作基础上,对灰度图像进行预白化处理(白化滤波器滤波)之后发现,对预白化后的图像的识别率比不作预白化处理低,分析可能是过度的预处理同时会去掉更多的特征信息,因此,对灰度化图像不作预白化处理。所以,本文的数据预处理即是在图像分类之前,对所有的染色肿瘤细胞图像进行灰度化,以降低图像的维数,去除不必要的冗余信息。图 2是灰度化后的肿瘤细胞图像。
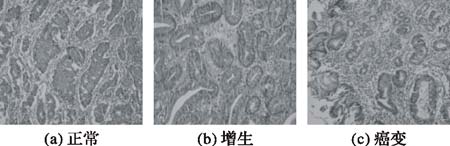
|
图 2 典型的正常、增生和癌变肿瘤细胞灰度图像 |
在RRC模型中,为了使模型对异常像素点具有鲁棒性,用参数τ来评价δ,按照式δ=ψ1(e)l来计算δ的值,其中向量e∈Rn,ψ1(e)k是集合{e2j,j=1,2,…,n}中第k个最大的元素,l=τn,τ∈(0,1)。本节讨论了RRC中的参数δ对最终识别率的影响。根据式(13)可知,参数δ是区分正常像素点和异常像素点的关键参数(若一个像素点的残差的方差比δ大,那么该像素点的权值小于0.5;否则,权值大于0.5)。因此,有必要讨论一下τ值的选择。图 3是在特征维度为792×33×24)时,不同的τ值下肿瘤细胞图像的识别率。从图 3可看出,τ值的区间可以设为[0.5,0.8]。
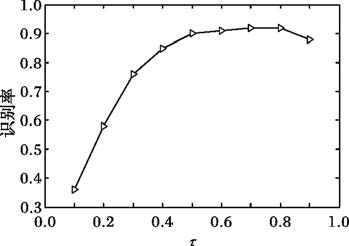
|
图 3 不同τ值下的识别率 |
常用的稀疏表示分类模型有两种:RRC模型和鲁棒稀疏表示(Robust Sparse Coding,RSC)模型[12]。由于图像的特征维度对识别率和识别时间有重要的影响,在经过大量实验之后,选取了具有几种具有代表性的特征维度(12×8,21×15,33×24,42×30,62×45)来验证本文方法的性能。将由FDDL方法得到的特征字典应用于RSC模型进行分类的方法(FDDL-RSC)与本文方法(FDDL-RSC)进行比较。表 1为两种分类模型在不同特征维度下的识别准确率和识别时间对比结果。
从表 1可看出:
| 表 1 FDDL-RRC和FDDL-RSC的识别准确率和识别时间对比 |
1) 相同维度下,FDDL-RRC方法的识别率要略低于FDDL-RSC。两种分类方法的识别率在维度为42×30时均达到了最高值,但比维度为33×24时提高不多,FDDL-RRC方法仅提高0.4个百分点,FDDL-RSC方法仅提高0.6个百分点。当维度大于42×30时,两种方法的识别率都开始下降。
2) 识别时间随着维度的提高不断加大,在33×24维度之后呈倍数增长。FDDL-RRC方法的识别时间要远少于FDDL-RSC方法。 综合考虑:当维度为33×24时,两种方法的识别性能均最好,并且识别率相差不大;但FDDL-RSC耗时约为FDDL-RRC的5倍。所以,经过对比可得出FDDL-RRC的分类性能比FDDL-RSC要好。
4.4 不同稀疏表示分类方法的比较为了验证本文肿瘤细胞图像识别方法的鲁棒性和有效性,与FDDL稀疏表示分类方法[10]和传统的稀疏表示分类方法[11]做了对比实验,样本集、训练集、测试集大小均相同,样本维度为各种方法最优时的维度,其中SRC方法的最优维度为12×10,FDDL为60×43,RSC和RRC均为42×30,本文方法FDDL-RRC和FDDL-RSC均为33×24,对比结果如表 2所示。
| 表 2 不同稀疏表示分类方法的识别性能比较 |
从表 2可以看出,由于对稀疏编码系数添加了Fisher判别约束,使得稀疏编码系数的类内散度尽可能小,而类间散度尽可能大,从而使得到的特征字典的原子与样本的类别信息相对应,整个字典具有良好的表达能力,所以FDDL的分类准确率要比SRC分类高。由于RRC比稀疏表示模型和FDDL稀疏表示模型具有更好的鲁棒性,可以更好地解决肿瘤细胞图像中存在的噪声和其他干扰等问题,所以RRC的识别率要高于FDDL和SRC,但是低于RSC。作为FDDL与RRC的有效结合的本文方法要比单独采用FDDL和RRC进行分类的效果要好。
4.5 不同识别方法的比较将本文方法与常用的肿瘤细胞图像特征提取与分类算法从识别率和运行时间这两方面进行比较。
对比方法如下:压缩感知(Compressed Sensing,CS)与自组织神经网络(Self-Organizing Feature Maps,SOFM)相结合的分类方法[7]、非负稀疏编码(Non-negative Sparse Coding,NNSC)与SOFM相结合的分类方法[8]、局部线性嵌入(Locally Linear Embedding,LLE)与最小二乘支持向量机(Least Squares SVM,LS-SVM)相结合的分类方法、双向2DPCA(Bidirectional Two-Dimensional Principal Component Analysis,Bi2DPCA)与SVM相结合的分类方法、基于卷积神经网络(Convolutional Neural Network,CNN)的分类方法。
所有的实验数据集来自同一肿瘤细胞图像集,CNN采用传统的Lenet5结构对肿瘤细胞灰度图像进行分类。不同方法的识别率和识别时间对比如表 3所示。
| 表 3 不同肿瘤细胞图像识别方法的分类性能比较 |
从表 3可看出:本文方法的识别性能最佳。本文方法比其他方法的识别率提高了至少2.4个百分点,并且大幅减少了识别时间。
5 结语考虑到以往稀疏表示模型在图像识别过程中忽略了图像的类别信息以及不能够有效地描述图像实际的编码残差,本文提出了FDDL-RRC肿瘤细胞图像的识别方法。本文方法对训练样本矩阵进行字典学习,在字典学习过程中引入了Fisher准则,使字典更具判别性,最后采用RRC模型进行分类识别。在肿瘤细胞图像数据集上的实验结果表明,相对于目前比较流行的稀疏表示模型以及其他肿瘤细胞图像识别方法,本文方法具有较高的分类准确率和良好的鲁棒性。
| [1] |
赵晓健, 曾晓勤. 基于稠密光流轨迹和稀疏编码算法的行为识别方法[J].
计算机应用, 2016, 36 (1) : 182-187.
( ZHAO X J, ZENG X Q. Behavior recognition method based on dense optical flow path and sparse coding algorithm[J].
Journal of Computer Applications, 2016, 36 (1) : 182-187.
) ( 0) 0)
|
| [2] |
马路, 邓承志, 汪胜前, 等. 特征保留的稀疏表示图像去噪[J].
计算机应用, 2013, 33 (5) : 1416-1419.
( MA L, DENG C Z, WANG S Q, et al. Feature preserving sparse representation for image denoising[J].
Journal of Computer Applications, 2013, 33 (5) : 1416-1419.
doi: 10.3724/SP.J.1087.2013.01416 ) ( 0) 0)
|
| [3] |
何玲丽, 李文波. 基于对称Gabor特征和稀疏表示的人脸识别[J].
计算机应用, 2014, 34 (2) : 550-552.
( HE L L, LI W B. Face recognition based on symmetric Gabor feature and sparse representation[J].
Journal of Computer Applications, 2014, 34 (2) : 550-552.
) ( 0) 0)
|
| [4] |
孙俊, 王文渊, 卓晴. 基于稀疏编码的提取人脸整体特征算法[J].
清华大学学报(自然科学版), 2002, 42 (3) : 411-413.
( SUN J, WANG W Y, ZHUO Q. Facial global feature extraction algorithm based on sparse coding[J].
Journal of Tsinghua University (Science and Technology), 2002, 42 (3) : 411-413.
) ( 0) 0)
|
| [5] |
SHI J, LI Y, ZHU J, et al. Joint sparse coding based spatial pyramid matching for classification of color medical image[J].
Computerized Medical Imaging and Graphics, 2015, 41 (1) : 61-66.
( 0) 0)
|
| [6] |
黄文明, 蔡文正, 邓珍荣. 基于稀疏编码的脑脊液图像快速识别[J].
计算机应用, 2014, 34 (7) : 2040-2043.
( HUANG W M, CAI W Z, DENG Z R. Fast identification of cerebrospinal fluid images based on sparse coding[J].
Journal of Computer Applications, 2014, 34 (7) : 2040-2043.
) ( 0) 0)
|
| [7] |
谢丽娟. 基于压缩感知和神经网络的肿瘤细胞图像识别研究[D]. 南昌:华东交通大学, 2014: 43-44.
( XIE L J. Research on recognition for tumor cell images based on compressive sensing and neural network [D]. Nanchang: East China Jiaotong University, 2014: 43-44.
) ( 0) 0)
|
| [8] |
刘梦清. 基于非负稀疏编码和神经网络的肿瘤细胞图像识别[D]. 南昌:华东交通大学, 2013: 45-46.
( LIU M Q. Tumor cell image recognition based on non-negative sparse coding and neural network [D]. Nanchang: East China Jiaotong University, 2013: 45-46.
) http://www.doc88.com/p-7062012724315.html ( 0) 0)
|
| [9] |
YANG M, ZHANG L, YANG J, et al. Regularized robust coding for face recognition[J].
IEEE Transactions on Image Processing, 2013, 22 (5) : 1753-1766.
doi: 10.1109/TIP.2012.2235849 ( 0) 0)
|
| [10] |
YANG M, ZHANG L, FENG X, et al. Fisher discrimination dictionary learning for sparse representation[C]//Proceedings of the 2011 International Conference on Computer Vision. Piscataway, NJ: IEEE, 2011: 543-550.
( 0) 0)
|
| [11] |
WRIGHT J, YANG A Y, GANESH A, et al. Robust face recognition via sparse representation[J].
IEEE Transactions on Pattern Analysis and Machine Intelligence, 2009, 31 (2) : 210-227.
doi: 10.1109/TPAMI.2008.79 ( 0) 0)
|
| [12] |
YANG M, ZHANG L, YANG J, et al. Robust sparse coding for face recognition[C]//Proceedings of the 2011 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway, NJ: IEEE, 2011: 625-632.
( 0) 0)
|




