2. 石家庄市农林科学研究院, 石家庄 050041
2. Shijiazhuang Academy of Agriculture and Forestry, Shijiazhuang 050041, China
小檗碱(BBR)俗称黄连素,因其低廉的价格和良好的生物安全性在医药及农业领域受到广泛关注。近年来,小檗碱多用于治疗胃肠疾病及防治植物病害[1],而在工业生物技术领域的应用鲜有报道。开发受小檗碱诱导的遗传元件可有望使其替代现有的昂贵诱导剂,从而建立小檗碱-DNA探针介导的生物传感器,灵敏、安全地完成靶标检测[2-4]。而筛选能与小檗碱高特异性、高亲和力结合的核酸适配体(aptamer)是实现该目标的前提。
Tuerk和Gold于1990年建立了一种指数富集的配体系统进化技术(SELEX)[5],可用于从随机单链寡核苷酸文库中筛选出能与靶物质特异性结合且高度亲和的核酸适体。目前,该技术已成功用于抗生素、蛋白质、有机物、金属离子、细胞等靶标的适配体筛选[6-8]。本文拟利用SELEX技术从随机单链DNA文库中筛选出能与小檗碱高特异性结合的适配体,并对其亲和力进行表征,为构建受小檗碱调控的遗传元件奠定基础。
1 实验部分 1.1 材料 1.1.1 单链DNA随机文库用于SELEX的随机单链寡核苷酸文库由上海生工生物工程技术有限公司合成,两端各含15 nt固定序列,中间为40 nt的随机序列,理论库容为1014~1015条不同的碱基序列。具体序列为5′-CCGGCGTAGAGGATC-40N-GGCCTCTAAACGGGT-3′。
1.1.2 引物捕获引物F0用于将单链寡核苷酸库固定到链霉亲和素磁珠上,其3′端标记生物素。扩增引物F1/R1用于聚合酶链式反应(PCR)过程对每轮筛选得到的单链次级库进行富集,其中R1的5′端标记生物素。具体引物序列见表 1。
| 下载CSV 表 1 引物序列 Table 1 Primers used in this study |
链霉亲和素磁珠,New England Biolabs (Beijing)有限公司;DNA分子量Marker、DNA聚合酶,博迈德生物技术有限公司;小檗碱,陕西慧科植物开发有限公司;克隆载体pClone007,大肠杆菌感受态细胞DH5α,北京擎科新业生物技术有限公司;其余试剂为国产分析纯,购自宝如亿(北京)生物技术有限公司。
1.1.4 缓冲液及培养基结合缓冲液:0.5 mol/L NaCl, 20 mmol/L Tris-HCl, 1 mmol/L乙二胺四乙酸(EDTA),调节pH至7.5。
Luria-Bertani(LB)培养基(g/L):酵母提取物5,NaCl 10,胰蛋白胨10。固体培养基需加琼脂至终质量浓度15 g/L;抗性培养基需加入氨苄青霉素至终质量浓度60 μg/mL。
1.2 实验方法 1.2.1 PCR反应条件优化每轮筛选得到的单链次级库需经PCR反应扩增得到双链DNA,再经过不对称PCR富集目的单链[9]。首先,分别设置PCR过程的循环数为5、8、12、16、20、24、30,扩增后经琼脂糖凝胶电泳鉴定,确定最佳循环数;之后,为确定不对称PCR过程的最佳引物浓度比例,分别设置上游扩增引物F1与下游扩增引物R1的浓度比为30:1、50:1、80:1、100:1,模板为之前对称PCR纯化产物,凝胶电泳鉴定。
PCR反应参数:95 ℃,4 min;94 ℃,1 min,55 ℃,40 s,72 ℃,30 s,重复x个循环;72 ℃,5 min;16 ℃保存。
1.2.2 SELEX筛选小檗碱的核酸适配体具体筛选步骤如下。
(1) 取2 μL单链随机库(1 μmol/L),加入200 μL结合缓冲液,混匀后加入4 μL捕获引物F0(100 μmol/L),95 ℃解链10 min,自然冷却至室温,此时捕获引物与单链随机库5′端互补配对,因此退火形成双链,同时捕获引物3′端标记生物素,用于将单链随机库固定至链霉亲和素磁珠上。
(2) 取150 μL链霉亲和素磁珠,加入800 μL结合缓冲液,磁分离,弃上清,洗涤两次。
(3) 将结合有捕获引物的单链随机库与链霉亲和素磁珠混匀,30 ℃轻振30 min,此时单链随机库通过生物素-链霉亲和素的作用固定到磁珠表面。经磁分离、去上清后,用结合缓冲液洗涤3次,备用。
(4) 在已连接单链随机库的磁珠中加入200 μL质量浓度为50 μg/mL的小檗碱溶液,充分吸打混匀,37 ℃振荡孵育1 h,间隙摇匀,与BBR结合而引起构象变化的单链寡核苷酸片段会与捕获引物脱离。
(5) 孵育1 h后,磁分离,收集清液,测定单链DNA浓度,以单链DNA为模板、F1/R1为引物进行对称PCR扩增得到双链DNA。
(6) 以对称PCR产物为模板,一定比例的F1/R1为引物进行不对称PCR,获得大量不含生物素标记的目的单链DNA,而双链DNA的一条链上携带生物素标记,用于后续分离。
(7) 取150 μL链霉亲和素磁珠用缓冲液洗涤3次后,用200 μL缓冲液重悬,加入300 μL不对称PCR产物,30 ℃下100 r/min振荡30 min。之后磁分离收集上清,测定单链次级库浓度,用于第二轮筛选。
重复步骤(1)~(7),反复筛选以得到特异性较高的核酸适配体。
1.2.3 适配体序列的测定多轮筛选结束后,将单链次级库进行对称PCR扩增,纯化后与载体pClone007进行TA克隆,并转化到大肠杆菌感受态细胞DH5α中,37 ℃培养12 h后,挑选单菌落,PCR鉴定后测序。
1.2.4 适配体结构预测分析适配体经测序后,使用Clustal X对其一级结构的同源性进行分析,并使用RNA Structure软件进行二级结构预测。
1.2.5 适配体与小檗碱亲和力的测定采用平衡渗透法评估适配体与小檗碱的亲和力[10]。选取同源性较高的几条适配体进行体外合成,用结合缓冲液配制成一系列浓度梯度的适配体溶液(0、1、2、3、4、5、6 μmol/L),分别加入终质量浓度为20 μg/mL的小檗碱溶液,30 ℃结合反应1 h。之后将混合液加入到截留率为3 kD的超滤离心管中,12 000 r/min离心30 min,收集清液并用高效液相色谱(HPLC)测定小檗碱含量。根据式(1)对每一个适配体的Kd值进行计算。
| $ y = \frac{{{B_{\max }}x}}{{{K_{\rm{d}}} + x}} $ | (1) |
式中y表示饱和度,即适配体结合的小檗碱占总小檗碱的质量分数;x表示加入适配体的浓度,μmol/L;Bmax为适配体与小檗碱的最大结合数目;Kd表示二者的解离常数,μmol/L。
2 结果与讨论 2.1 最佳PCR反应条件的确定SELEX技术可通过多轮循环将与小分子高特异性亲和的片段筛选出来,利用PCR扩增富集筛得的片段是SELEX过程中的重要步骤。
首先,以随机库为模板,进行对称PCR扩增得到双链DNA。由图 1可知,70 nt单链随机库条带大致位于50 bp DNA Marker附近,经过PCR后的70 bp双链DNA位于75 bp DNA Marker附近。同时PCR循环数为20、24、30时,均能得到较清晰的条带,考虑到扩增产物浓度,最终确定PCR循环数为30。
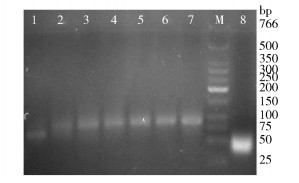
|
M—DNA分子量Marker;1~7—对称PCR产物(对应循环数分别为5, 8, 12, 16, 20, 24, 30);8—单链随机库。 图 1 对称PCR扩增双链DNA Fig.1 PCR amplification of dsDNA |
为获得大量目的单链DNA作为后续筛选的次级库,采用不对称PCR方法进行扩增,其中引物比例是影响不对称PCR的重要因素。由图 2可知,当上下游扩增引物F1与R1浓度比为30:1、50:1时,均得到明显的单链条带;而F1与R1浓度比为80:1、100:1时,可能由于上游引物浓度过大导致非特异性结合明显,扩增结果出现了多条杂带。为保证单链次级库的浓度与质量,选择F1与R1浓度比为50:1作为不对称PCR的引物条件。
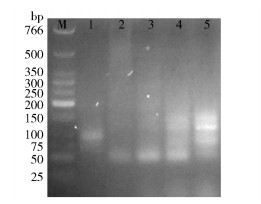
|
M—DNA分子量Marker;1—双链DNA;2~5—不对称PCR产物(F1与R1浓度比分别为30:1, 50:1, 80:1, 100:1)。 图 2 不对称PCR扩增单链DNA Fig.2 Asymmetric PCR amplification of ssDNA |
经过8轮筛选,将每轮得到的单链次级库进行琼脂糖凝胶电泳鉴定,结果如图 3所示。在50 bp Marker附近均出现目标条带,且随着筛选轮数的增加,单链条带越发致密单一,说明单链库中的成分已得到较充分的富集。
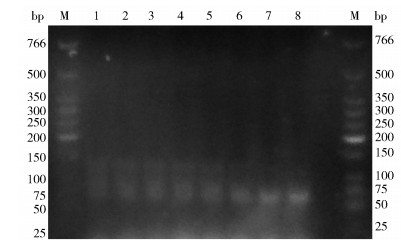
|
M—DNA分子量Marker;1~8—每轮筛选的不对称PCR产物。 图 3 1~8轮筛选不对称PCR产物 Fig.3 Asymmetric PCR amplification of one to eight rounds of screening products |
第8轮的筛选结果经过PCR扩增,纯化回收后与载体进行TA克隆,转化宿主菌E.coli DH5α,经菌落PCR鉴定后,选取了70株阳性克隆进行序列测定。共有67个重组子测序成功,结果分为4组,其中S2和S4两组序列重现性较低,S1、S3两组在整体结果中分别出现22及38次重复,证明了经8轮筛选后,适配体已得到充分富集,序列特异性有了明显提高。同时各组结果之间也存在一些保守序列,主要包括富含GC的序列TCC和GCC(表 2中以斜体及下划线标明)。具体序列及保守性分析如表 2所示。
| 下载CSV 表 2 适配体测序结果 Table 2 Sequencing results of aptamers |
分别对S1、S3 2组适配体进行二级结构模拟预测。如图 4所示,适配体中都存在明显的茎环结构,且S1结构较为饱满(图 4(a)),而S3则由多个小茎环组成(图 4(b))。核酸适配体形成发夹、茎、环、假结等构象,可以在空间结构上与靶分子互补契合,这是适配体高特异性、高亲和力结合靶标的机制之一。同时,适配体一级结构包含多个富含鸟嘌呤(G)的区域,可能会形成常见的G-四链体结构,这也是核酸适配体的重要特征之一[11-12]。
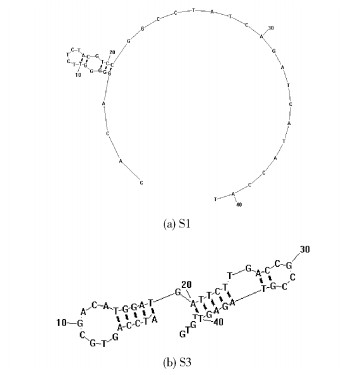
|
图 4 小檗碱适配体二级结构模拟 Fig.4 Secondary structure simulation of aptamers |
利用平衡渗透法对适配体与小檗碱的亲和力进行评估。固定小檗碱质量浓度为20 μg/mL,分别加入不同浓度的适配体混合反应。混合液经过超滤离心管离心后,与适配体结合的小檗碱被截留在膜内,滤液中仅包括未与适配体结合的游离小檗碱,其含量用HPLC法进行测定。
与米氏常数的表征方法类似,将式(1)进行双倒数变换,得到式(2)
| $ \frac{1}{y} = \frac{{{K_{\rm{d}}}}}{{{B_{\max }}}}\frac{1}{x} + \frac{1}{{{B_{\max }}}} $ | (2) |
根据适配体的浓度梯度及测得的小檗碱含量对式(2)进行线性拟合,即可计算得到每条适配体与小檗碱作用的解离常数Kd,具体结果如表 3所示。
| 下载CSV 表 3 适配体与小檗碱作用的解离常数 Table 3 Dissociation constants of aptamers |
从表 3可以看出,2条适配体的Kd值均为nmol级别,说明经过8轮SELEX筛选,成功获得了与小檗碱具有高亲和力作用的适配体序列。其中S1的Kd值仅为523 nmol/L,与小檗碱的亲和力最大,能够满足后续小檗碱介导遗传元件的构建或生物传感器的开发。
3 结论(1) 采用不对称PCR方法成功扩增得到大量可用于SELEX筛选的单链DNA,经反应条件优化,确定PCR最佳循环数为30,最佳上下游引物浓度比为50:1。
(2) 经8轮筛选,对67个重组子进行测序,成功获得4条与小檗碱高度特异性结合的适配体,对其中2条重现性较好的序列进行一级结构分析及二级结构模拟的结果表明,适配体序列富含GC,且易形成茎环及G-四链体结构。
(3) 采用平衡渗透法对2条适配体与小檗碱作用的亲和力测定结果表明,2条适配体均表现出对小檗碱的高亲和力,其中S1的Kd值最小,仅为523 nmol/L,与小檗碱亲和力最大。
| [1] |
HOU D Y, YAN C Q, LIU H X, et al. Berberine as a natural compound inhibits the development of brown rot fungus Monilinia fructicola[J]. Crop Protection, 2010, 29(9): 979-984. DOI:10.1016/j.cropro.2010.05.015 |
| [2] |
PARK J H, BYUN J Y, JANG H, et al. A highly sensitive and widely adaptable plasmonic aptasensor using berberine for small-molecule detection[J]. Biosensors & Bioelectronics, 2017, 97: 292-298. |
| [3] |
OVÁDEKOVÁ R, JANTOVÁ S, LETAŠIOVÁ S, et al. Nanostructured electrochemical DNA biosensors for detection of the effect of berberine on DNA from cancer cells[J]. Analytical & Bioanalytical Chemistry, 2006, 386(7/8): 2055-2062. |
| [4] |
LETAŠIOVÁ S, JANTOVÁ S, MIKO M, et al. Effect of berberine on proliferation, biosynthesis of macromolecules, cell cycle and induction of intercalation with DNA, dsDNA damage and apoptosis in Ehrlich ascites carcinoma cells[J]. Journal of Pharmacy & Pharmacology, 2006, 58(2): 263-270. |
| [5] |
TUERK C, GOLD L. Systematic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase[J]. Science, 1990, 249(4968): 505-510. DOI:10.1126/science.2200121 |
| [6] |
YANG X J, BING T, MEI H C, et al. Characterization and application of a DNA aptamer binding to L-tryptophan[J]. Analyst, 2011, 136(3): 577-585. DOI:10.1039/C0AN00550A |
| [7] |
CERCHIA L, DUCONGÉ F, PESTOURIE C, et al. Neutralizing aptamers from whole-cell SELEX inhibit the RET receptor tyrosine kinase[J]. PLoS Biology, 2005, 3(4): e123. DOI:10.1371/journal.pbio.0030123 |
| [8] |
WANG L H, LIU X F, HU X F, et al. Unmodified gold nanoparticles as a colorimetric probe for potassium DNA aptamers[J]. Chemical Communications, 2006, 36: 3780-3782. |
| [9] |
SVOBODOVÁ M, PINTO A, NADAL P, et al. Comparison of different methods for generation of single-stranded DNA for SELEX processes[J]. Analytical & Bioanalytical Chemistry, 2012, 404(3): 835-842. |
| [10] |
ZIMMERMANN G R, JENISON R D, WICK C L, et al. Interlocking structural motifs mediate molecular discrimination by a theophylline-binding RNA[J]. Nature Structural Biology, 1997, 4(8): 644-649. DOI:10.1038/nsb0897-644 |
| [11] |
ZHANG L H, LIU H, SHAO Y, et al. Selective lighting up of epiberberine alkaloid fluorescence by fluorophore-switching aptamer and stoichiometric targeting of human telomeric DNA G-quadruplex multimer[J]. Analytical Chemistry, 2015, 87(1): 730-737. DOI:10.1021/ac503730j |
| [12] |
WANG J, ZHANG Y J, WANG H Y, et al. Selection and analysis of DNA aptamers to berberine to develop a label-free light-up fluorescent probe[J]. New Journal of Chemistry, 2016, 40: 9768-9773. DOI:10.1039/C6NJ02290A |








